| |
| Med Sci (Paris). 38(8-9): 740–742. doi: 10.1051/medsci/2022114.Rôle du facteur de transcription HES6 dans le mélanome uvéal Léa Nucera,1a Elise Perrée,1b Pauline Pujas,1c Chloé Rouchon,1d Maryem Wachich,1e Sangavi Selveswaran,1f and Nicole Arrighi1g 1
Polytech Nice Sophia, Spécialité Génie biologique 5
e
année, Pharmacologie et Biotechnologies, Université Côte d’Azur
,
France MeSH keywords: Facteurs de transcription à motif basique hélice-boucle-hélice, Régulation de l'expression des gènes, Humains, Mélanome, Protéines de répression, Facteurs de transcription, Tumeurs de l'uvée, génétique |
Environ 50 % des patients atteints d’un mélanome uvéal (MU) décèdent dans l’année qui suit le diagnostic de métastases, présentes dans 90 % des cas au niveau du foie [
1
]. Or, les métastases de MU surviennent dans environ 50 % des cas à 15 ans du diagnostic initial, et ce, malgré un traitement efficace de la tumeur primaire [
2
]. Récemment, le tebentafusp (IMC-gp100)
1
a montré des résultats encourageants, permettant d’augmenter pour la première fois la survie globale des patients atteints de MU métastatique (MUm). Comparé à un traitement ciblant un checkpoint immunitaire (PD1 – pembrolizumab), le bénéfice du tebentafusp a montré un meilleur taux de survie à un an (73,2 % contre 57,5 %). Ce traitement est d’autant plus prometteur qu’il a obtenu une autorisation temporaire d’utilisation (ATU) effective au 30 mars 2022 pour les patients porteurs de l’allèle HLA-A*02:01. Sachant que 45 % de la population caucasienne est porteuse de cet allèle et que 20 % des patients traités par tebentafusp répondent au traitement, le tebentafusp bénéficierait en théorie à 9 % des patients atteints de MUm [
3
]. Il est donc nécessaire de continuer à comprendre les mécanismes moléculaires impliqués dans le MU afin d’identifier de nouvelles cibles thérapeutiques permettant d’améliorer la survie d’une majorité de patients.
Parmi les signatures moléculaires analysées à partir de MU primaires à l’échelle de la cellule unique et mises en évidence par des analyses bio-informatiques (méthode SCENIC), celle montrant une forte expression du facteur HES6 (
hairy and enhancer of split (HES) family BHLH Transcription factor 6
) est associée à un mauvais pronostic [
4
].
Dans cette même étude, l’hypothèse de l’implication d’HES6 dans la progression du MU a été confirmée par des analyses de Kaplan-Meier montrant que l’expression d’HES6 est inversement corrélée à la survie des patients atteints de MU primaires (données du TCGA,
The Cancer Genome Atlas
).
Plusieurs autres observations renforcent l’idée qu’HES6 est un facteur de mauvais pronostic dans les MU. En effet, l’expression d’HES6 est associée à une diminution de celle du gène suppresseur de tumeur BAP1 (
BRCA1-associated protein 1
) qui code une enzyme de déubiquitination intervenant dans les mécanismes de réparation de l’ADN ou de la différenciation cellulaire. Le gène
BAP1
est souvent muté chez les personnes atteintes de MU, ce qui peut être un facteur de mauvais pronostic. Enfin, l’expression d’HES6 est plus importante dans les MU contenant plus de 70 % de cellules épithélioïdes (de moins bon pronostic comparés à ceux en contenant moins de 30 %). HES6 est également impliqué dans d’autres cancers comme le glioblastome, le cancer colorectal ou de la prostate.
Dans cette nouvelle, nous proposons tout d’abord de décrire le facteur de transcription HES6 puis d’aborder son rôle dans le MU. |
HES6, un membre peu étudié de la famille des facteurs de transcription HES
Les facteurs de transcription HES, au nombre de 7 (HES1-HES7), sont des protéines à domaine basique hélice-boucle-hélice (bHLH). La région basique est responsable de la liaison à l’ADN tandis que la région HLH, comprenant principalement des résidus hydrophobes, permet aux protéines de se dimériser [
5
]. Les facteurs HES possèdent aussi un domaine Orange (motif d’environ 35 acides aminés également appelé hélice II/IV) impliqué dans la spécificité des dimères formés, et un domaine WRPW conservé (motif tétrapeptidique Trp-Arg-Pro-Trp de la partie C-terminale de HES) qui régule leur dégradation par le protéasome.
HES6 est une protéine de 22 acides aminés, codée par le gène du même nom localisé sur le chromosome 2 [
5
]. Dans certains cancers, comme le glioblastome, les cancers de l’estomac, du côlon, du sein, du poumon et du rein, HES6 est décrit comme un biomarqueur de mauvais pronostic, dont l’expression élevée est associée à une survie diminuée des patients [
8
,
9
]. De plus, des études fonctionnelles montrent qu’il contrôle différents processus impliqués dans la progression tumorale, comme la prolifération et la motilité cellulaire, suggérant qu’il représente aussi une cible thérapeutique potentielle [
4
].
|
Rôle du facteur de transcription HES6 dans le mélanome uvéal
Alors que l’expression ectopique de HES6 dans les cellules de lignées primaires ou métastatiques de MU entraine une augmentation de la migration cellulaire
in vitro
, la motilité des cellules de MU induite par l’activation de NOTCH est diminuée lorsque l’expression d’HES6 est inhibée, suggérant qu’HES6 est impliqué dans les effets résultant de l’activation de la voie NOTCH [
4
]. Chez les mammifères, la voie NOTCH est activée par des ligands de la famille Delta-like (DLL1, 3-4) ou Jagged (JAG1/2), conduisant à des clivages protéolytiques successifs et à la libération du domaine intracellulaire de NOTCH, NICD (
Notch intracellular domain
) dans le cytoplasme
(
Figure 1
)
. NICD va ensuite rentrer dans le noyau, pour former un complexe ternaire avec le facteur de transcription CSL (assemblage des sous-unités : CBF1 – Su(H) et LAG-1) et un coactivateur transcriptionnel de la famille Mastermind (MAM), puis participer à la régulation de la transcription [
6
]. Parmi les cibles de cette voie de signalisation, se retrouvent les facteurs de transcription HES, qui vont se dimériser et se lier à l’ADN, jouant ainsi le rôle de répresseur transcriptionnel. NOTCH joue un rôle important dans le développement et le renouvellement des tissus et régule des processus biologiques tels que la prolifération, la migration, la différenciation cellulaire et l’apoptose. Il n’est donc pas surprenant qu’une dérégulation de l’activité de NOTCH et de l’expression des différents acteurs de cette voie, en particulier HES6, soit liée à des troubles du développement ou à des maladies comme le cancer [
7
].
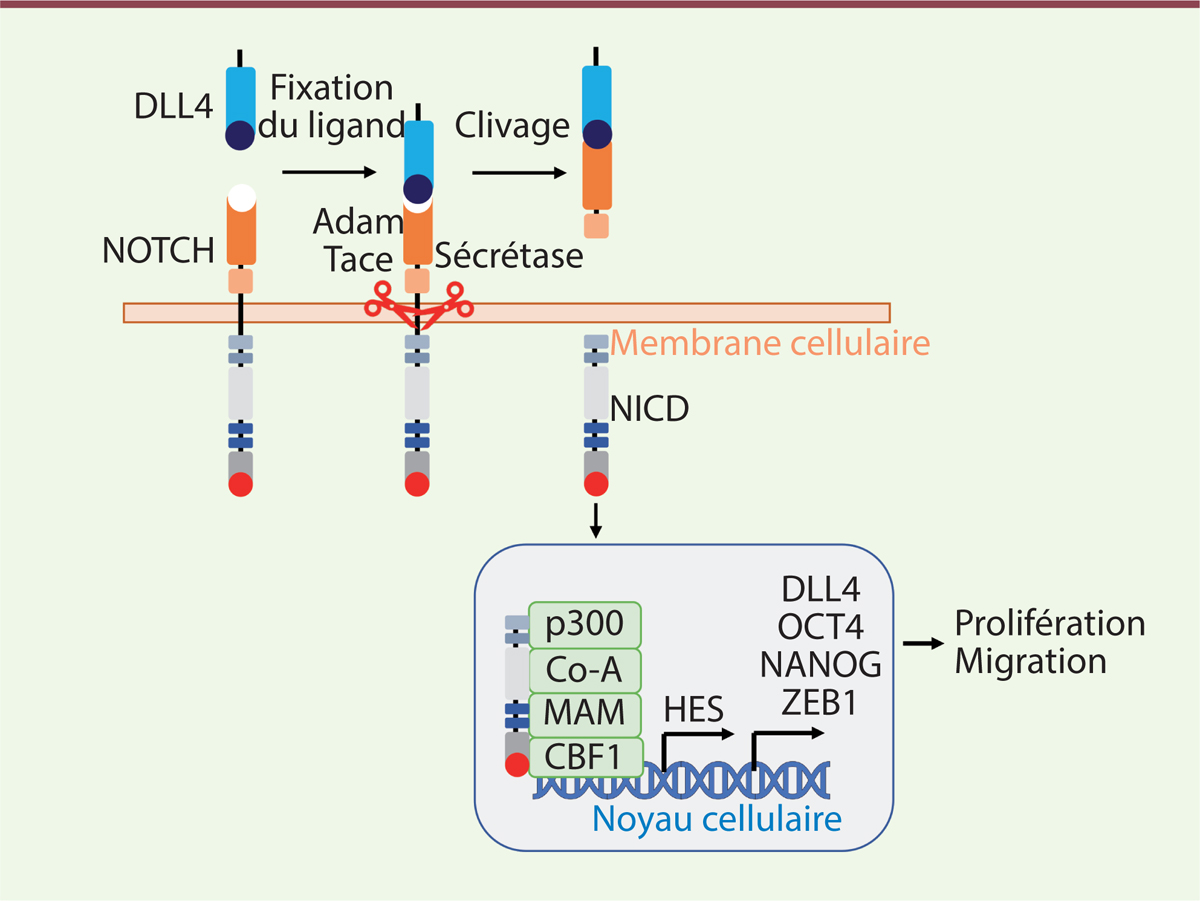
 | Figure 1.
Régulation des facteurs de transcription HES par la voie de signalisation de NOTCH.
La fixation du ligand DLL4 sur le récepteur membranaire NOTCH 1 active une cascade de clivages enzymatiques (métalloprotéase ADAM/TACE et γ sécrétase) des régions transmembranaire et sous-membranaire de ce récepteur, isolant le complexe NICD. Un complexe protéique comprenant le coactivateur Co-A, la protéine MAM, et NICD se forme alors et se fixe sur la protéine CBF1 elle-même fixée à l’ADN. Cette fixation permet alors de recruter un nouveau coactivateur, p300, induisant la transcription des gènes effecteurs de NOTCH, dont font partie les gènes de la famille HES. HES6 joue un rôle en aval de NOTCH, mais il reste à démontrer que son expression est régulée par la voie NOTCH. DLL4 :
delta-like ligand
4 ;
ADAM
:
A-disintegrin and metalloproteinase
; TACE :
tumor necrosis factor(TNF)-alpha-converting enzyme
;
NICD : Notch intracellular domain
; Co-A : coactivateur de la transcription ; MAM : coactivateur transcriptionnel de la famille Mastermind ; CBF1 : facteur de transcription CBF1 pour
C promoter-binding factor 1
; OCT4 : Facteur de transcription OCT4 pour
octamer-binding transcription factor 4
; NANOG : facteur de transcription NANOG, ZEB1 :
zinc finger E-box-binding homeobox 1
.
|
Le rôle d’HES6 a aussi été étudié
in vivo
dans un modèle de greffe de tumeurs humaines dans la membrane chorioallantoïde (CAM) d’embryon de poulet [
4
]. Des cellules cancéreuses de MU, dans lesquelles l’expression de HES6 peut-être inhibée par traitement par la doxycycline, ont été greffées au niveau de la CAM supérieure. Neuf jours plus tard, les tumeurs ont été collectées et mesurées. Une diminution de la croissance et des capacités métastatiques a été observée pour les tumeurs se développant à partir de cellules n’exprimant pas HES6 en comparaison avec les cellules témoins. Ainsi, une diminution d’HES6 entraine une inhibition de la prolifération et de la motilité des cellules du MU
in vitro
et
in vivo
[
4
].
En conclusion, HES6 représente un nouveau marqueur pronostique et une cible thérapeutique pour limiter la prolifération et la dissémination du MU [
10
]. Actuellement, il n’existe pas d’inhibiteurs spécifiques d’HES6. Il est donc nécessaire de comprendre le fonctionnement et la régulation de ce facteur pour identifier des régulateurs, en amont, et/ou des effecteurs, en aval, susceptibles d’être ciblés. Remarquablement, le régulon HES6 contient différents effecteurs qui représentent d’autres cibles potentielles à tester [
4
]. Une autre piste intéressante pourrait être l’approche PROTAC (
proteolysis-targeting chimeras
), qui permet de cibler spécifiquement la dégradation de protéines, en particulier grâce à de petites molécules bifonctionnelles, qui induisent une dégradation par le système ubiquitine du protéasome [
11
]. Cette approche permettrait d’étudier l’impact de la dégradation d’HES6 sur l’expression de gènes pro- ou anti-tumoraux et sur la croissance tumorale dans les mélanomes uvéaux.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
|
BAP1
|
BRCA1-associated protein 1
|
|
Domaine WRPW
|
Motif tétrapeptidique Trp-Arg-Pro-Trp de la partie C-terminale de HES |
|
Domaine Orange
|
Motif d’environ 35 acides aminés, appelé aussi hélice II/IV, proche de la partie C-terminale du domaine bHLH responsable d’interaction protéine-protéine, noté bHLH-O. |
|
HES6
|
Hairy
/
Enhancer of Split
(
Hes
) 6
|
|
PROTAC
|
Proteolysis Targeting Chimeras |
|
TCGA
|
The Cancer Genome Atlas
|
|
Footnotes |
1.
Rantala
ES
,
Hernberg
MM
,
Piperno-Neumann
S
,
et al.
Metastatic uveal melanoma: The final frontier.
.
Prog Retin Eye Res.
2022
;
:101041.
.
2.
Kujala
E
,
Mäkitie
T
,
Kivelä
T
Very long-term prognosis of patients with malignant uveal melanoma.
.
Invest. Ophthalmo Vis Sci.
2003;
;
44
:
:4651.
–
4659
.
3.
Nathan
P
,
Hassel
JC
,
Rutkowski
P
,
et al.
Overall survival benefit with tebentafusp in metastatic uveal melanoma.
.
N Engl J Med.
2021;
;
385
:
:1196.
–
206
.
4.
Pandiani
C
,
Strub
T
,
Nottet
N
,
et al.
Single-cell RNA sequencing reveals intratumoral heterogeneity in primary uveal melanomas and identifies HES6 as a driver of the metastatic disease.
.
Cell Death Differ.
2021;
;
28
:
:1990.
–
2000
.
5.
Neklesa
T
,
Winkler
J
. and
Crews
C
.
Targeted protein degradation by PROTACs.
.
Pharmacol Ther.
2017;
;
174
:
:138.
–
44
.
6.
Baron
M.
An overview of the Notch signalling pathway.
.
Semin Cell Dev Biol.
2003;
;
14
:
:113.
–
119
.
7.
Kopan
R
,
Ilagan
MX
.
The canonical Notch signaling pathway: Unfolding the activation mechanism.
.
Cell.
2009;
;
137
:
:216.
–
233
.
8.
Xu
Y
,
Liu
X
,
Zhang
H
.
et al.
Overexpression of HES6 has prognostic value and promotes metastasis via the Wnt/β-catenin signaling pathway in colorectal cancer.
.
Oncol Rep.
2018;
;
40
:
:1261.
–
1274
.
9.
Haapa-Paananen
S
,
Kiviluoto
S
,
Waltari
M
et al.
HES6 gene is selectively overexpressed in glioma and represents an important transcriptional regulator of glioma proliferation.
.
Oncogene.
2012;
;
31
:
:1299.
–
1310
.
10.
Bertolotto
C
.
Cutaneous and uveal melanoma: two different cancers in therapeutic needs.
.
CR Biol.
2021;
;
344
:
:219.
–
31
.
11.
Churcher
I.
Protac-induced protein degradation in drug discovery: breaking the Rules or just making new ones ?
.
J Med Chem.
2018;
;
61
:
:444.
–
452
.
|