| |
| Med Sci (Paris). 38(12): 990–998. doi: 10.1051/medsci/2022164.Les virus et l’émergence des cellules eucaryotes modernes Patrick Forterre1,2* and Morgan Gaïa3** 1Département de microbiologie, Institut Pasteur
,
25 rue du Docteur Roux
,
75015Paris
,
France 2Institut de biologie intégrative de la cellule (I2BC), Département de microbiologie, CEA, CNRS, Université Paris-Saclay
,
91198Gif-sur-Yvette
,
France 3Génomique métabolique, Genoscope, Institut François Jacob, CEA, CNRS, Univ. Evry, Université Paris-Saclay
,
91057Évry
,
France |
Vignette (© Philippe Roingeard).
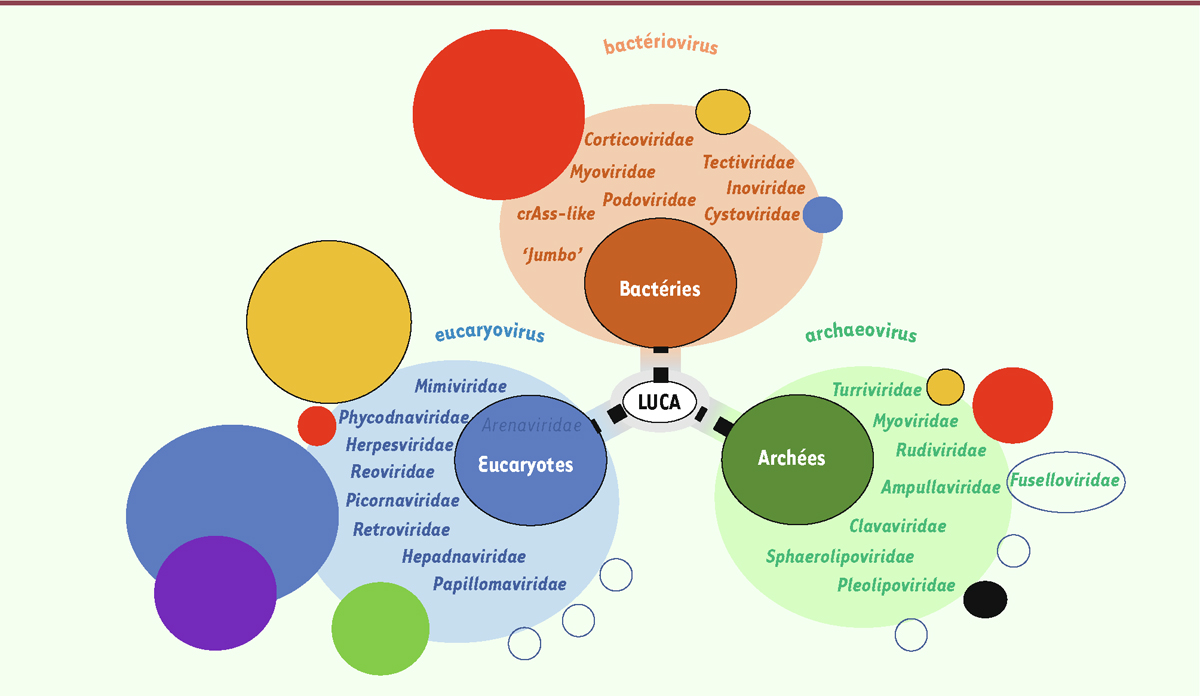
Nos connaissances sur les virus, leur diversité, leurs rôles mais aussi leurs possibles origines ont explosé ces dernières années, bousculant au passage leur définition et notre perception de leur place dans la biosphère [
1
–
4
]. Trois ensembles de virus ont été identifiés, chacun étant spécifique d’un des trois domaines du vivant : les archées, les bactéries, et les eucaryotes (
Figure 1
). Nous parlerons donc ici des archéovirus, des bactériovirus (traditionnellement appelés bactériophages), et des eucaryovirus. Les eucaryovirus sont particulièrement divers. À l’écriture de ces lignes, 131 familles virales ont en effet été identifiées chez les eucaryotes, contre respectivement 28 chez les bactéries et 16 chez les archées (d’après la ressource Virus du NCBI, en ne tenant compte que de génomes de référence complètement séquencés et rattachés à une famille). Cette différence de représentation est sans doute en partie biaisée par une recherche plus active des virus chez les eucaryotes, mais elle reflète bien leur extraordinaire diversité. Cette diversité se retrouve en premier lieu dans celle de leurs génomes. Tous les types de génomes et de réplication sont en effet représentés chez les eucaryovirus, ce qui n’est pas le cas des virus des deux autres domaines. La majorité des eucaryovirus sont des virus à ARN. Ceux-ci sont relativement rares parmi les bactériovirus et complètement absents chez les archéovirus [
5
]. Les rétrovirus, avec leur génome à ARN et une réplication impliquant une étape utilisant l’ADN, sont strictement spécifiques aux eucaryotes. Cette diversité des eucaryovirus se retrouve également dans leur taille, puisque certains, les
Nucleocytoviricota
, font partie des plus grand virus connus à l’heure actuelle, à la fois par la taille de leurs génomes et par celle de leurs virions.
 | Figure 1.
Représentation schématique des trois domaines du vivant et des sphères virales qui leur sont associées
(voir encart et [
6
]). Les tailles des cercles représentent de façon schématique la proportion relative des virus correspondants dans chacun des trois domaines. Les cercles vides symbolisent des groupes de virus apparentés qui n’ont pas encore été formellement reconnus par l’ICTV comme de nouvelles sphères virales.
|
Les eucaryotes présentent eux aussi de très nombreuses particularités par rapport aux deux domaines cellulaires procaryotes. En effet, si l’origine des eucaryotes est aussi débattue (principalement entre une émergence indépendante à partir d’un ancêtre commun avec les archées, ou directement au sein de ces dernières à partir d’une lignée qui aurait accumulé les différentes caractéristiques eucaryotes), c’est en partie dû aux nombreux traits spécifiques qui les distinguent des archées et des bactéries : noyau, splicéosome
1,
, cytosquelette, etc. Ces traits étant partagés par tous les eucaryotes modernes, ils étaient probablement déjà présents chez LECA, leur dernier ancêtre commun
2
. D’autres particularités complexes ont également émergé dans des groupes d’eucaryotes plus restreints : par exemple, les Viridiplantae et les Métazoaires ont développé indépendamment des systèmes extraordinairement sophistiqués, tels que la floraison, le système nerveux central ou encore un système immunitaire adaptatif particulièrement efficace. Cette complexité est associée à l’existence de mécanismes de différenciation cellulaire qui permettent la présence de multiples formes cellulaires chez un même organisme.
Il semble alors légitime de se demander si l’extraordinaire diversité des eucaryovirus n’a pas quelque chose à voir avec la trajectoire évolutive si particulière des organismes eucaryotes. En d’autres termes, la co-évolution entre eucaryovirus et leurs hôtes pourrait-elle expliquer, au moins en partie, les traits spécifiques des eucaryotes modernes ? |
Origine et diversité des virus eucaryotes
La virosphère eucaryote se caractérise par l’abondance et la diversité des virus à ARN, qui sont inconnus chez les archées et rares chez les bactéries [
5
]. Ces virus possèdent tous une ARN réplicase (ou transcriptase inverse) homologue et sont classés en une même sphère (
Encadré 1
), celle des
Riboviria
, au sein de cinq branches majeures dont trois ne comprennent que des eucaryovirus [
6
]. Les virus à ADN sont également divers, mais la plupart des eucaryovirus à ADN font partie de larges groupes comprenant également des archéovirus et des bactériovirus. En particulier, la majorité des eucaryovirus à ADN double-brin appartiennent à deux sphères virales dites « cosmopolites », les
Varidnaviria
et les
Duplodnaviria
, définies par leurs protéines de capside, et qui sont les seules à comprendre à la fois des archéovirus, des bactériovirus, et des eucaryovirus [
6
].
Alors que les eucaryotes et les archées sont très proches sur le plan moléculaire, étonnamment, les virus de ces deux sphères cosmopolites semblent plutôt marquer une séparation entre eucaryovirus d’un côté, et virus de procaryotes de l’autre (
Figure 2A
). Ainsi, les archéovirus et les bactériovirus de la sphère
Duplodnaviria
font partie d’un même groupe, la classe des
Caudoviricetes
, qui inclue un nombre considérable de familles virales, correspondant toutes à des virus tête-queue particulièrement abondants chez les bactéries et inconnus chez les eucaryotes. En revanche, les eucaryovirus de cette sphère ne sont représentés pour le moment que par une seule famille, celle des
Herpesviridae
, dans la classe
Herviviricetes
. Du côté des
Varidnaviria
, les virus infectant les archées ou les bactéries sont relativement semblables et regroupés dans la classe des
Tectiliviricetes
, alors que les eucaryovirus se positionnent clairement à l’écart [
7
]. Ces derniers comprennent en particulier des virus à ADN double-brin de grande taille, parmi lesquels les virus dit « géants », qui ont tous été regroupés dans le phylum des
Nucleocytoviricota
, jusqu’à récemment encore connus par l’acronyme NCLDV (pour
nucleocytoplasmic large DNA viruses
).
 | Figure 2.
Les deux schémas du haut (A)
représentent les relations évolutives entre les eucaryovirus, les bacteriovirus et les archéovirus des deux sphères de virus cosmopolites, les
Varidnaviria
[
6
,
7
] et les
Duplodnaviria
, montrant que les eucaryovirus se regroupent à part des deux autres ensembles de virus. Au contraire, les eucaryotes se regroupent avec les archées dans l’arbre universel du vivant fondé sur les ARN ribosomiques et/ ou les protéines universelles [
9
]. Les deux schémas du bas
(B, C)
représentent les deux scénarios pour l’origine des eucaryotes et de leurs virus. Les cercles bleus correspondent à la position du dernier ancêtre commun des eucaryotes modernes, LECA. Dans le scénario 2D
(B)
, les eucaryotes émergent à la suite de l’endosymbiose d’une bactérie dans une archée (flèche en gros pointillé), la bactérie devenant la mitochondrie. Les eucaryovirus proviennent du mélange de leurs deux virosphères. Dans le scénario 3D
(C)
, les eucaryotes et les archées dérivent d’un ancêtre commun aujourd’hui disparu. Dans ce scénario, certains eucaryovirus peuvent également dériver de la bactérie à l’origine des mitochondries (flèche en petit pointillé), mais d’autre peuvent provenir de la virosphère qui entourait cet ancêtre. La distinction entre ces deux scénarios est importante dans le cas des virus à ARN. Les cercles oranges représentent les cinq familles de virus à ARN spécifiques des eucaryotes ou d’origine eucaryote [
6
]. Dans le scénario 2D, ces cinq familles sont apparues
de novo
chez les proto-eucaryotes. En revanche, dans le scénario 3D, elles pourraient dériver d’anciens virus à ARN ayant précédé LUCA.
|
Les eucaryovirus étaient probablement déjà différenciés avant l’émergence de LECA, comme le suggère la distribution des différents groupes de virus à ADN et à ARN dans les différentes lignées eucaryotes [
5
]. Dans le scénario évolutif dit « deux domaines », selon lequel les eucaryotes seraient issus de l’association entre une archée, appelée Asgard, et une alpha-protéobactérie, il a été proposé que l’ensemble des eucaryovirus seraient les descendants des virus ayant infecté ces deux procaryotes [
8
] (
Figure 2B
). Dans l’autre scénario majeur, dit « trois domaines », selon lequel les eucaryotes et les archées partagent un ancêtre commun [
9
], certains groupes de virus spécifiques des eucaryotes auraient pu émerger avant cet ancêtre, voire avant LUCA
3
, et être ensuite perdus dans les autres domaines (
Figure 2C
, cercle orange). Ceci pourrait être en particulier le cas des familles de virus à ARN inconnus chez les bactéries, ce qui permet d’imaginer un lien de parenté direct entre les premiers virus à ARN apparus au cours de l’évolution et les virus à ARN des eucaryotes modernes. Quel que soit le scénario envisagé, il suppose une relativement longue période de coévolution entre les ancêtres des eucaryovirus actuels et les proto-eucaryotes avant l’émergence de LECA (
Figure 2B, C
), période durant laquelle de nombreux groupes de virus ont pu jouer un rôle dans la formation de la cellule eucaryote moderne.
|
Nouvelle classification des virus proposée par l’ICTV
L’étude structurale des protéines de capside virale et de celles impliquées dans la réplication des virus a profondément changé la vision de leur évolution et a conduit à revoir leur classification. En effet, il est apparu que ces protéines pouvaient présenter des similarités de structure chez des virus appartenant à des familles distinctes. Cette découverte a conduit l’ICTV (
International Committee on Taxonomy of Viruses
) à abandonner la classification de Baltimore fondée sur la nature du génome viral et à la remplacer par une classification taxonomique fondée sur la conservation structurale des protéines de capside et/ou des protéines de réplication virale [
6
]. Au sein de cette classification, les virus sont ordonnés en 15 taxons. Aux taxons traditionnels (phylums, ordres, familles, genres, etc.), l’ICTV a ajouté deux taxons supérieurs chez les virus : le royaume (
kingdom
) et le
realm
. Le
realm
correspond au taxon le plus élevé chez les virus et permet de distinguer les virus des organismes codant des ribosomes (archées, bactéries et eucaryotes) pour lesquels le taxon le plus élevé est le
domain
. Nous proposons d’utiliser en Français le terme de « sphères », une des traductions possibles de
realm
, et qui se rapproche de la notion de « virosphère » souvent utilisée pour décrire l’ensemble du monde viral.
|
|
Course aux armements et transferts de gènes
Les virus sont des acteurs majeurs de sélection et de variabilité à l’échelle du vivant [
10
]. En effet, leur mode de vie induit une pression de sélection permanente sur leurs hôtes, qui en exercent une en retour sur leurs virus
via
l’émergence de nouveaux mécanismes de défenses : c’est une course aux armements continue que se livrent virus et cellules, course qui a pu jouer un rôle dans la complexité croissante des mécanismes moléculaires lors de la transition des proto-eucaryotes vers les eucaryotes modernes. On peut d’ailleurs penser que de nombreuses « armes » apparues dans un camp ont pu être ensuite récupérées par l’autre camp. La coiffe des ARN messagers pourrait en être un exemple : on considère généralement que les virus coiffent leurs ARN messagers d’un nucléotide modifié à l’extrémité 5’ pour qu’ils ressemblent à ceux des eucaryotes et ainsi « berner » la cellule infectée. Or, il existe chez les eucaryovirus une très grande variété de coiffes qui, pour beaucoup, diffèrent de celles des eucaryotes [
11
]. On pourrait donc se demander si la fonction n’est pas inverse : se distinguer des ARN messagers de leurs hôtes pour pouvoir les dégrader ou les inactiver spécifiquement. Dans ce cas, les gènes codant la coiffe pourraient donc être d’origine virale et avoir été ensuite récupérés par un ancêtre des eucaryotes, qui au fil des mutations ont pu coiffer leurs propres ARN messagers et tromper les virus [
12
].
Les virus peuvent également profondément influencer la trajectoire évolutive de leurs hôtes en provoquant des transferts de gènes. Ces derniers sont favorisés par la proximité immédiate des génomes viraux avec ceux de leurs hôtes au sein de la cellule infectée. Par ailleurs, de nouveaux gènes apparaissent sans doute constamment chez les virus lors de la réplication de leurs génomes, par les mêmes mécanismes moléculaires qui commencent à être bien connus pour les génomes cellulaires [
13
]. Les virus, entités biologiques les plus abondantes sur la Terre, représentent ainsi un réservoir extraordinaire de nouvelles fonctions biologiques. Ils peuvent donc transférer non seulement des gènes cellulaires d’une cellule à une autre, mais également leurs propres gènes, notamment lors d’évènements d’intégration du génome viral dans celui de l’hôte.
De très nombreux virus infectant les trois domaines du vivant sont capables d’intégrer leurs génomes dans celui de leur hôte. Ils sont particulièrement abondants dans les génomes des archées et des bactéries ; il a d’ailleurs été reconnu depuis longtemps que des intégrations de bactériovirus ont joué un rôle majeur dans l’évolution des bactéries [
14
]. L’importance des intégrations virales dans l’évolution des eucaryotes a été reconnue plus récemment. L’attention s’est initialement portée essentiellement sur les intégrations des rétrovirus, particulièrement abondants dans les génomes eucaryotes. Ainsi, Camille Heisserer, Marc-André Selosse et Jean-Michel Drezen discutent, dans ce numéro, du rôle de plusieurs protéines issues des rétrovirus dans l’origine du placenta (fusion cellulaire) ou dans celui du système nerveux central [
49
]. L’intégration des rétrovirus est facilitée par la transcription inverse de l’ARN viral en ADN. Il a toutefois été montré que les virus non-rétroviraux peuvent également s’intégrer dans les génomes eucaryotes. Ces évènements sont rares mais s’accumulent sur le long terme, résultant en la présence de gènes de ces virus dans les génomes eucaryotes [
15
,
16
]. Les virus à ARN ne codent qu’un petit nombre de gènes, mais quelques gènes peuvent être suffisants pour profondément modifier la physiologie des cellules dans lesquelles ils s’expriment. Une expérience menée sur des plants de betterave a ainsi montré que l’expression d’une protéine de 25 kDa du virus à ARN des nervures jaunes nécrotiques de la betterave, entraîne des modifications dans l’expression d’une centaine de gènes chez la plante transformée, ainsi qu’un phénotype particulier caractérisé par l’apparition de racines branchées [
17
].
Différentes familles de virus à ADN peuvent également s’incorporer de façon durable dans les génomes eucaryotes [
19
]. Dans le cas des virus à ADN de grande taille, tels les
Nucleocytoviricota
, ce sont généralement des fragments qui sont retrouvés dans les génomes eucaryotes les plus divers [
20
]. Il a été récemment montré que jusqu’à 10 % environ du contenu en gènes de certaines algues vertes correspondaient à de larges segments de
Nucleocytoviricota
intégrés [
21
]. Plus globalement, les
Nucleocytoviricota
semblent être impliqués dans de nombreux transferts de gènes avec les eucaryotes, et ce, dans les deux directions de transferts [
22
]. L’analyse des transferts dans le sens virus vers cellules a montré que plusieurs recrutements indépendants de glycosyltransférases provenant de
Nucleocytoviricota
ont pu être à l’origine de changement de la morphologie cellulaire au cours de la différenciation des lignées eucaryotes [
22
].
S’il est maintenant clairement établi que les virus ont joué un rôle important au cours de la différenciation des eucaryotes modernes à partir de LECA, il semble logique de penser qu’ils ont également dû jouer un rôle dans l’évolution des proto-eucaryotes qui ont abouti à l’émergence de ce dernier. Ainsi, un rôle possible des virus à ARN dans l’émergence de la reproduction sexuée peut être envisagé. En effet, les protéines virales fusogènes de classe II, impliquées dans la fusion de la membrane des virions de certains virus à ARN avec la membrane cellulaire, pourraient être à l’origine de la protéine HAP2/GCS1 (
Hapless 2
/
generative cell-specific 1
) permettant la fusion des gamètes [
18
]. Mais depuis la découverte des virus géants [
23
], c’est surtout le rôle possible des
Nucleocytoviricota
dans l’eucaryogenèse qui a été particulièrement analysé et discuté [
12
,
13
,
24
–
27
].
|
Un rôle spécifique des
Nucleocytoviricota
dans l’origine des eucaryotes
Le phylum des
Nucleocytoviricota
comprend les plus grands virus connus, autant en termes de virions, dont certains sont plus gros que les plus petites bactéries, que de taille de génomes qui vont d’environ 100 kb (kilobases) à 2,5 Mb (megabases). Ces virus codent de nombreuses protéines de type eucaryote : des protéines informationnelles (intervenant dans la réplication, la transcription, ou la traduction : ADN et ARN polymérases, primases, topoisomérases, facteurs de processivité, d’élongation, coiffe ARN [
25
,
28
,
29
]), mais aussi des protéines structurales (comme les histones [
30
]), des protéines impliquées dans la formation du cytosquelette chez les eucaryotes (actine, myosine [
31
,
32
]), ou encore dans celle des vésicules (protéines SNARE [
33
]). À l’exception des facteurs associés à la traduction, ces protéines ne semblent pas avoir été acquises récemment à partir de leurs hôtes, formant des clades distincts des eucaryotes dans les analyses phylogénétiques.
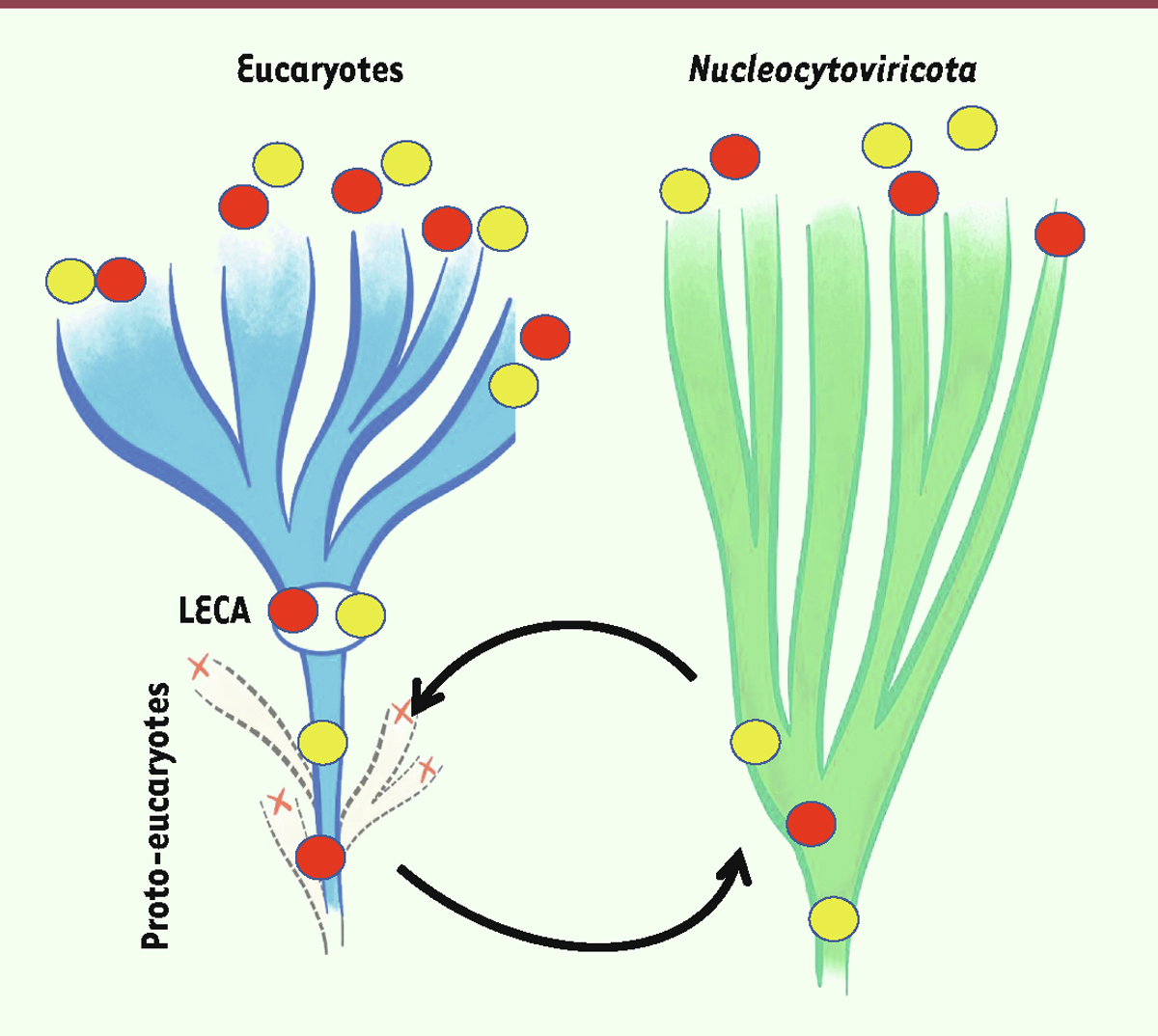
Plusieurs scénarios peuvent expliquer cette observation (
Figure 3
). On peut imaginer qu’un ancêtre des
Nucleocytoviricota
ait récupéré ces protéines d’un proto-eucaryote (transfert de cellule à virus), ou l’inverse (transfert de virus à cellule). Des scénarios plus complexes sont évidemment possibles, comme, par exemple, un premier transfert de proto-eucaryotes vers un
Nucleocytoviricota
, suivi d’une période d’évolution spécifique avant un second transfert d’un virus vers les proto-eucaryotes. D’ailleurs, toutes les protéines de type eucaryote chez les
Nucleocytoviricota
n’ont pas nécessairement suivi la même trajectoire évolutive, plusieurs scénarios hypothétiques coexistant pour différents groupes de protéines.
 | Figure 3.
Représentation schématique des deux principaux scénarios pouvant expliquer la présence de protéines homologues chez les eucaryotes et les Nucleocytoviricota
: transfert d’une protéine cellulaire d’un proto-eucaryote vers un ancêtre des Nucleocytoviricota (cercle rouge) ou transfert d’une protéine virale d’un ancêtre des Nucleocytoviricota vers un proto-eucaryote (cercle jaune). Certaines de ces protéines peuvent avoir été ensuite perdues dans des lignées virales au cours de la diversification des Nucleocytoviricota, expliquant leur distribution hétérogène. Du côté des eucaryotes, s’il s’agit de protéines jouant un rôle important dans la physiologie cellulaire ; celles-ci seront généralement conservées chez tous les eucaryotes si elles étaient déjà présentes chez LECA.
|
Traditionnellement, l’hypothèse de transferts des cellules vers les virus a été privilégiée par la plupart des biologistes. Les auteurs qui soutiennent l’idée d’un rôle majeur des virus dans l’eucaryogenèse ont, au contraire, mis en avant la possibilité de transfert des
Nucléocytoviricota
vers les proto-eucaryotes. Ainsi, plusieurs auteurs ont proposé une origine virale pour les réplicases eucaryotes [
34
–
36
]. Toutefois, des phylogénies approfondies restent à réaliser pour dévoiler le sens du transfert, des virus aux cellules ou inversement. Les réplicases des eucaryotes et des
Nucléocytoviricota
appartiennent en effet à la famille des ADN polymérases B, dont l’histoire évolutive est particulièrement complexe [
37
]. Une origine chez les
Nucleocytoviricota
a également été proposée pour les protéines de la coiffe des ARN messagers [
25
], qui sont identiques à celles des eucaryotes ; là encore, des phylogénies approfondies restent à entreprendre. Récemment, nous avons pu obtenir des phylogénies robustes en faveur d’un transfert des
Nucleocytoviricota
vers les proto-eucaryotes pour l’ADN topoisomérase de la famille IIA (Topo IIA) [
38
]. Toutes les Topo IIA eucaryotes forment en effet un clade qui est localisé à l’intérieur des
Nucleocytoviricota
(
Figure 4
). Les Topo IIA interviennent notamment dans la réplication et la ségrégation de l’ADN chez les eucaryotes, et participent au squelette des chromosomes ; leur introduction dans la lignée proto-eucaryote aurait donc eu des conséquences considérables pour le fonctionnement cellulaire.
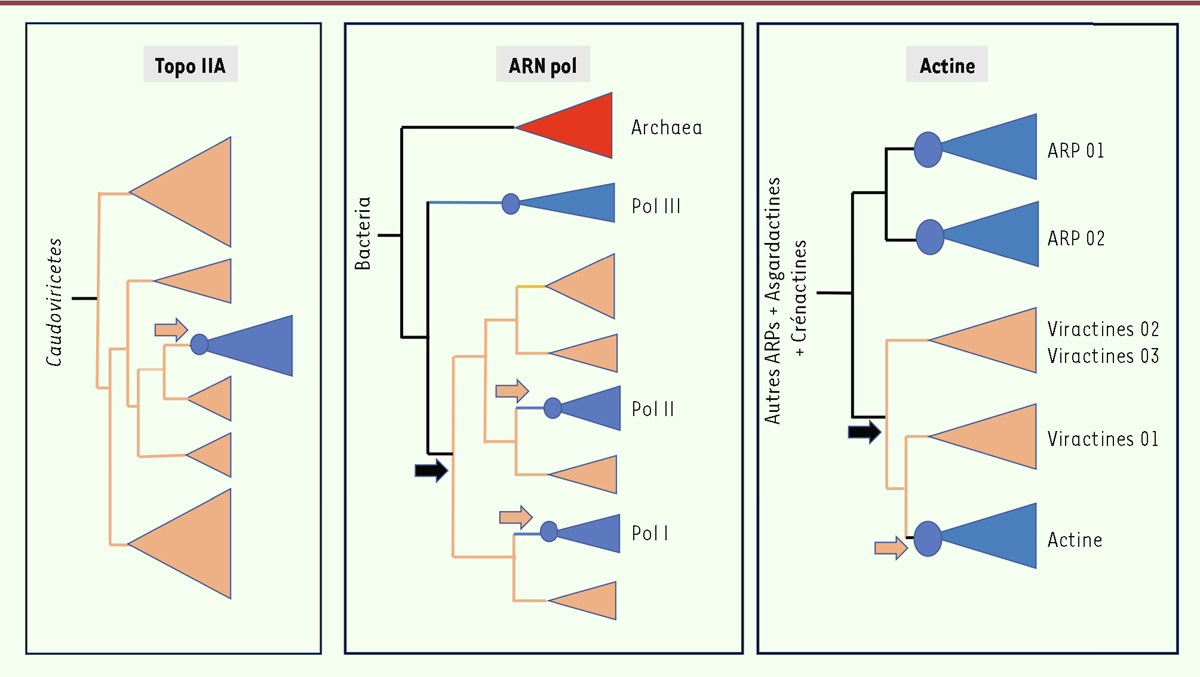
 | Figure 4.
Représentation schématique des phylogénies obtenues pour l’ADN topoisomérase IIA, les ARN polymérases et les actines (et protéines apparentées) chez les eucaryotes et les
Nucleocytoviricota
(d’après [
29
,
32
,
38
]). Les clades des différents groupes de
Nucleocytoviricota
sont en orange et ceux des eucaryotes sont en bleu. Les cercles bleus représentent la position du dernier ancêtre commun aux eucaryotes modernes (LECA). Les flèches noires indiquent les transferts des proto-eucaryotes vers les virus et les flèches orange indiquent les transferts des virus vers des proto-eucaryotes. Les arbres sont enracinés avec les groupes extérieurs les plus proches : les virus Caudoviricetes pour l’arbre Topo IIA, les bactéries pour l’arbre ARN pol (il s’agit d’une protéine universelle), et les protéines homologues pour l’arbre des actines (ARP 03 à 08, Asgardactines, Bathyactines, et Crénactines).
|
Nous avons pu mettre en évidence une situation plus complexe dans le cas des ARN polymérases (ARN pol) [
29
,
37
]. Les deux grandes sous-unités de l’ARN pol des
Nucleocytoviricota
sont homologues à celles des archées, des bactéries, et des eucaryotes. Ces derniers présentent toutefois la particularité d’avoir trois ARN pol nucléaires : l’ARN pol I, principalement associée aux ARN ribosomiques ; l’ARN pol II, responsable des ARN messagers ; et l’ARN pol III, en charge des ARN de transfert. Ces trois ARN pol sont présentes chez tous les eucaryotes « modernes » et étaient donc déjà présentes chez LECA. Par des analyses phylogénétiques poussées, nous avons pu montrer que les deux sous-unités de l’ARN pol II, ainsi probablement qu’une sous-unité de l’ARN pol I, avaient été introduites dans la lignée proto-eucaryote par transfert de gènes à partir de deux lignées de
Nucleocytoviricota
[
37
] (
Figure 4
). Les clades eucaryotes correspondant à ces deux enzymes sont en effet localisés à l’intérieur des
Nucleocytoviricota
, à deux positions différentes. En revanche, le clade eucaryote correspondant à l’ARN pol III est localisé entre les archées et les
Nucleocytoviricota.
Nous avons donc proposé le scénario suivant : un ancêtre des
Nucleocytoviricota
a recruté une ARN pol ancestrale d’un proto-eucaryote peu de temps après la formation de cette lignée cellulaire. Cette ARN pol ancestrale a fini par devenir, chez les eucaryotes, l’ARN pol III, tandis que celle acquise par les virus s’est spécifiée au fil de leur diversification. Plus tard, l’ARN pol spécifique des
Imitervirales
, un ordre des
Nucleocytoviricota
comprenant les virus géants, tels que Mimivirus et Pandoravirus, a été transférée aux proto-eucaryotes, résultant
in fine
en l’ARN pol II eucaryote. Séparément, un autre transfert a impliqué une sous-unité de l’ARN pol d’un autre groupe de
Nucleocytoviricota
. Son association avec la sous-unité d’une ARN pol cellulaire issue d’une duplication de l’ARN pol ancestrale a conduit à l’apparition de l’ARN Pol I [
29
,
37
].
Il est intéressant de noter que chez les eucaryotes « modernes », l’ARN pol II et la Topo IIA interagissent à la fois sur les plans structural et fonctionnel. La Topo IIA, composant structural du complexe ARN pol II holoenzyme, est essentielle pour une synthèse optimale de l’ADN associé aux nucléosomes [
39
]. Elle facilite aussi le recrutement de l’ARN pol II aux promoteurs chez les levures [
40
]. Il est donc possible que ces deux enzymes aient été transférées ensemble d’un
Nucleocytoviricota
vers les proto-eucaryotes.
Récemment, ce sont des gènes codant deux protéines clés du cytosquelette eucaryote, l’actine et la myosine, qui ont été identifiées dans les génomes de plusieurs
Imitervirales
[
31
,
32
]. Des transcrits de leurs gènes ont d’ailleurs été détectés dans plusieurs viromes océaniques, suggérant leur expression lors d’infections virales. Dans le cas des actines virales, que nous avons appelées viractines, un scénario similaire à celui des ARN pol est soutenu par les analyses phylogénétiques : un premier transfert d’une actine ancestrale d’un proto-eucaryote vers un virus, suivi d’un retro-transfert après une période de diversification (
Figure 4
). Ce dernier retro-transfert, de virus vers proto-eucaryotes, pourrait être à l’origine de l’actine majeure présente chez les eucaryotes « modernes », témoignant une fois de plus d’un lien évolutif particulièrement étroit entre les virus du phylum
Nucleocytoviricota
et les proto-eucaryotes.
|
En 2001, avant la découverte des virus géants, Masaharu Takemura et Philip Bell avaient indépendamment proposé l’hypothèse selon laquelle le noyau des cellules provenait d’un virus apparenté à la famille des
Poxviridae
, dont le virus de la vaccine fait partie [
25
,
26
]. Leur hypothèse, appelée l’eucaryogenèse virale, était fondée sur les similarités de structure et de mécanismes entre le noyau eucaryote et les usines virales produites par l’infection du virus de la vaccine dans le cytoplasme des cellules infectées pour protéger la réplication virale. Le premier virus géant, Mimivirus, était découvert deux ans plus tard [
23
], et ce dernier s’est justement révélé être apparenté aux
Poxviridae
, tous deux faisant partie des
Nucleocytoviricota
. La plupart des virus infectant les eucaryotes, y compris des virus à ARN, produisent des usines virales [
41
], mais celles des
Nucleocytoviricota
sont particulièrement élaborées. Elles se forment souvent à partir des membranes du réticulum endoplasmique, voire de la membrane nucléaire, évoquant l’idée d’un lien évolutif [
10
,
13
].
Deux scénarios principaux sont considérés dans le cadre de l’eucaryogenèse virale : l’infection d’un proto-eucaryote sans noyau (donc phénotypiquement de type procaryote) par un
Nucleocytoviricota
ancestral suivi de la transformation de l’usine virale en noyau ; ou la récupération par le proto-eucaryote infecté des gènes viraux codant les protéines impliquées dans la formation de l’usine virale, lui permettant de constituer sa propre « usine à chromosomes ». Compte-tenu de la complexité du processus d’eucaryogenèse et la durée pendant laquelle les
Nucleocytoviricota
et proto-eucaryotes ont coévolué, on peut imaginer que plusieurs virus aient joué un rôle dans cet évènement certainement progressif.
Deux découvertes récentes ont également renforcé le lien apparent entre virus et noyau eucaryote. En 2017, Joe Poligliano et ses collaborateurs ont ainsi découvert une famille de
Caudoviricetes
de grande taille à l’origine de la formation d’un noyau viral dans la bactérie qu’ils infectent, par l’expression d’une seule protéine formant une membrane nucléaire [
42
]. La nouvelle structure permet au virus de répliquer son génome à l’abri des systèmes CRISPR. Ce noyau viral est par ailleurs positionné au centre de la bactérie infectée par des filaments formés par une tubuline virale homologue des tubulines eucaryotes, rappelant le positionnement des chromosomes eucaryotes par les tubulines du fuseau mitotique [
42
]. L’ADN viral est ensuite empaqueté dans les capsides virales qui se logent à la surface de ce « noyau »
via
un mécanisme encore inconnu. Il aura au préalable fallu que les ARN messagers viraux soient transférés du noyau viral vers le cytoplasme pour y être traduits, ces virus restants en effet sensibles aux systèmes CRISPR visant l’ARN [
43
]. On peut donc supposer l’existence de pores nucléaires permettant à ces ARN messagers de traverser la membrane du noyau viral.
Cette possibilité est d’autant plus envisageable que des pores « nucléaires » viraux ont pu être mis en évidence, mais dans un autre contexte : celui des virus à ARN, en particulier le SARS-CoV-2 (
severe acute respiratory syndrome coronavirus 2
). Les usines virales produites par ce virus se présentent sous la forme de vésicules entourées d’une double membrane lipidique issue des membranes intracellulaires [
44
]. Une équipe américaine a réussi à obtenir des images particulièrement spectaculaires de pores à la surface des vésicules permettant à l’ARN viral de quitter ces usines virales [
44
]. Une seule protéine virale, nsp3 (
non-structural protein 3
), une protéine de 222 kDa, est responsable du corps central de ces pores qui ressemblent de façon frappante aux pores nucléaires des eucaryotes.
Ces pores viraux, ainsi que le noyau viral de certains bactériovirus, ne peuvent évidemment pas être considérés comme des ancêtres possibles des noyaux eucaryotes ; ils démontrent en revanche la capacité des virus, dans leur globalité, à former des structures au fonctionnement proche des noyaux. L’idée d’un rôle majeur des virus dans l’apparition du noyau eucaryote est donc tout à fait possible. |
Le concept de cellule virale (
virocell
) et l’origine de la différenciation cellulaire
Plusieurs lignées d’eucaryotes sont caractérisées par l’abondance de formes multicellulaires complexes, ce qui a contribué fortement à leur expansion et à leur diversification. La formation des organismes multicellulaires passe par la transformation d’une cellule initiale en de multiples formes cellulaires par un phénomène de différenciation qui existe également chez certaines bactéries, mais qui a pris, chez les eucaryotes, une importance toute particulière. Un parallèle pourrait être tracé entre les transformations des cellules lors de leur différenciation et celles observées au cours des infections virales. Les transformations induites par l’infection peuvent en effet conduire à des changements spectaculaires dans la morphologie et/ou dans la physiologie des cellules infectées, comme en cours de différenciation cellulaire. Par exemple, le virus de la vaccine induit la mobilité des cellules épithéliales qu’il infecte, ce qui va conduire à la désorganisation des tissus et faciliter la propagation des virions dans l’organisme [
45
]. Plus récemment, il a été montré que l’infection par le SARS-CoV2 induit la fusion des cellules infectées et la formation de syncytium [
46
]. Chez les archées, certaines infections virales peuvent entraîner un gigantisme cellulaire suite à la manipulation par le virus des protéines du cycle cellulaire [
47
].
Le concept de cellule virale (ou
virocell
) a été introduit pour rendre compte de cette transformation d’une cellule non-infectée en une entité clairement différente lors de l’infection [
48
]. Le métabolisme, comme le mode d’interaction avec les autres cellules de l’organisme (pour les organismes multicellulaires) peuvent être très différents de ce qu’ils étaient avant l’infection. En effet, comme nous l’avons évoqué, les virus codent de nombreuses protéines régulatrices qui peuvent modifier la trajectoire phénotypique des cellules infectées. Ce statut de cellule virale ne se solde par ailleurs pas systématiquement par la mort cellulaire, plusieurs débouchés existant pour une infection. On peut se demander si les eucaryotes n’auraient pas appris des virus comment manipuler leurs propres cellules en fonction des besoins. Dans cette hypothèse, les eucaryotes auraient recruté des protéines régulatrices virales pour construire des mécanismes de différenciation cellulaire de plus en plus complexes.
Il sera intéressant de tester cette dernière hypothèse en cherchant des homologies entre protéines virales et protéines régulatrices intervenant dans la différenciation cellulaire, ou encore en déterminant si des virus ont pu jouer un rôle dans les quelques exemples connus de différenciation chez les bactéries. |
L’existence d’un rôle qu’ont pu jouer les interactions entre cellules et virus, déjà certainement intenses avant même l’apparition de LUCA, dans la formation des domaines cellulaires modernes ne fait plus guère de doute : l’ampleur de ce rôle dans l’émergence des eucaryotes modernes demeure toutefois encore mal connue. Il faut notamment expliquer comment ces interactions n’ont pas abouti à la formation de cellules aussi complexes chez les bactéries et les archées. Nous avons tenté dans cette synthèse, d’établir un parallèle entre la spécificité des eucaryotes et ce qui distingue leur virosphère de celles des deux autres domaines du vivant, bactéries et archées. En particulier, nous avons avancé l’idée d’un lien entre la différenciation cellulaire chez les eucaryotes et la capacité des virus à transformer les cellules infectées en cellules virales. Poursuivre l’exploration du monde viral reste bien évidemment une priorité. La fréquence des découvertes en matière de coévolution entre virus et cellules montre que cette exploration réserve sans doute encore bien des surprises. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Raoult
D
,
Forterre
P
.
Redefining viruses: Lessons from Mimivirus.
.
Nat Rev Microbiol.
2008;
;
6
:
:315.
–
319
.
2.
Forterre
,
P
.
To Be or Not to Be Alive: How Recent Discoveries Challenge the Traditional Definitions of Viruses and Life.
,
Stud Hist Philos Biol Biomed Sci.
2016
;
pii : S1369–8486(16)30008–5.
.
3.
Nasir
A
,
Romero-Severson
E
,
Claverie
JM
.
Investigating the Concept and Origin of Viruses.
.
Trends Microbiol.
2020;
;
28
:
:959.
–
67
.
4.
Koonin
EV
,
Dolja
VV
,
Krupovic
M
,
et al
.
Viruses Defined by the Position of the Virosphere within the Replicator Space.
.
Microbiol Mol Biol Rev.
2021;
;
85
(
(4)
) :
:e0019320.
.
5.
Nasir
A
,
Forterre
P
,
Kim
KM
,
et al.
The distribution and impact of viral lineages in domains of life.
.
Front Microbiol.
2014;
;
5
:
:194.
.
6.
Koonin
EV
,
Dolja
VV
,
Krupovic
M
,
et al.
Global organization and proposed megataxonomy of the virus world.
.
Microbiol Mol Biol Rev.
2020;
;
84
(
(2)
) :
:e00061.
–
19
.
7.
Woo
AC
,
Gaia
M
,
Guglielmini
J
,
et al
.
Phylogeny of the Varidnaviria morphogenesis module: congruence and incongruence with the tree of life and viral taxonomy.
.
Front Microbiol.
2021;
;
12
:
:704052.
.
8.
Koonin
EV
,
Dolja
VV
,
Krupovic
M
.
Origins and evolution of viruses of eukaryotes: The ultimate modularity.
.
Virology.
2015;
;
479–80
:
:2.
–
25
.
9.
Da Cunha
V
,
Gaia
M
,
Forterre
P
.
The expending Asgard archaea and their elusive relationships with Eukarya.
.
mLife.
2022;
;
1
:
:3.
–
12
.
10.
Forterre
P
,
Prangishvili
D
.
The great billion-year war between ribosome – and capsid-encoding organisms (cells and viruses) as the major source of evolutionary novelties.
.
Ann N Y Acad Sci.
2009;
;
1178
:
:65.
–
77
.
11.
Decroly
E
,
Ferron
F
,
Lescar
J
,
et al.
Conventional and unconventional mechanisms for capping viral mRNA.
.
Nat Rev Microbiol.
2012;
;
10
:
:51.
–
65
.
12.
Bell
PJL
.
Evidence supporting a viral origin of the eukaryotic nucleus.
.
Virus Res.
2020;
;
289
:
:198168.
.
13.
Forterre
P
,
Gaïa
M
.
Giant viruses and the origin of modern eukaryotes.
.
Curr Opin Microbiol.
2016;
;
31
:
:44.
–
49
.
14.
Ochman
H
,
Lawrence
JG
,
Groisman
EA
.
Lateral gene transfer and the nature of bacterial innovation.
.
Nature.
2000;
;
405
:
:299.
–
304
.
15.
Koonin
EV
.
Taming of the shrewd: novel eukaryotic genes from RNA viruses.
.
BMC Biol.
2010;
;
8
:
:2.
.
16.
Arneth
B
.
Leftovers of viruses in human physiology.
.
Brain Struct Funct.
2021;
;
226
:
:1649.
–
58
.
17.
Peltier
C
,
Schmidlin
L
,
Klein
E
,
et al.
Expression of the Beet necrotic yellow vein virus p25 protein induces hormonal changes and a root branching phenotype in Arabidopsis thaliana.
.
Transgenic Res.
2011;
;
20
:
:443.
–
466
.
18.
Valansi
C
,
Moi
D
,
Leikina
E
,
et al.
Arabidopsis HAP2/GCS1 is a gamete fusion protein homologous to somatic and viral fusogens.
.
J Cell Biol.
2017;
;
216
:
:571.
–
581
.
19.
Barreat
JCN
,
Katzourakis
A
.
Paleovirology of the DNA viruses of eukaryotes.
.
Trends in Microbiol.
2022;
;
30
:
:281.
–
92
.
20.
Filée
J.
Multiple occurrences of mimivirus core genes acquired by eukaryotic genomes.
.
Virology.
2014;
;
466–7
:
:53.
–
59
.
21.
Moniruzzaman
M
,
Weinheimer
AR
,
Martinez-Gutierrez
CA
,
et al.
,
Widespread endogenization of giant viruses shapes genomes of green algae.
.
Nature.
2020;
;
588
:
:141.
–
5
.
22.
Irwin
NAT
,
Pittis
AA
,
Richard
TA
,
et al
.
Systematic evaluation of horizontal gene transfer between eukaryotes and viruses.
.
Nature microbiol.
2022;
;
7
:
:327.
–
36
.
23.
La Scola
B
,
Audic
S
,
Robert
C
,
et al.
A giant virus in amoebae. Science.
2003;
;
299
:
:2033.
.
24.
Takemura
M.
Poxviruses and the origin of the eukaryotic nucleus.
.
J Mol Evol.
2001;
;
52
:
:419.
–
425
.
25.
Bell
PJ
.
Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus?
.
J Mol Evol.
2001;
;
53
:
:251.
–
256
.
26.
Takemura
M
.
Medusavirus Ancestor in a Proto-Eukaryotic Cell: Updating the Hypothesis for the Viral Origin of the Nucleus.
.
Front Microbiol.
2020;
;
11
:
:571831.
.
27.
Forterre
P
,
Gaia
M
.
Giant viruses and the origin of eukaryotic RNA polymerases.
.
Med Sci (Paris).
2021;
;
37
:
:230.
–
3
.
28.
Koonin
EV
,
Yutin
N
.
Evolution of the Large Nucleocytoplasmic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism.
.
Adv Virus Res.
2019;
;
103
:
:167.
–
202
.
29.
Guglielmini
J
,
Woo
AC
,
Krupovic
M
,
et al.
Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes.
.
Proc Natl Acad Sci USA.
2019;
;
116
:
:19585.
–
19592
.
30.
Liu
Y
,
Bisio
H
,
Toner
CM
,
et al
.
Virus-encoded histone doublets are essential and form nucleosome-like structures.
.
Cell.
2021;
;
184
:
:4237.
–50.
e19
.
31.
Kijima
S
,
Delmont
TO
,
Miyazaki
U
,
et al
.
Discovery of Viral Myosin Genes With Complex Evolutionary History Within Plankton.
.
Front Microbiol.
2021;
;
12
:
:683294.
.
32.
Da Cunha
V
,
Gaia
M
,
Ogata
H
,
et al
.
Giant Viruses Encode Actin-Related Proteins.
.
Mol Biol Evol.
2022;
;
39
(
(2)
) :
:msac022.
.
33.
Khalifeh
D
,
Neveu
E
,
Fasshauer
D
.
Megaviruses contain various genes encoding for eukaryotic vesicle trafficking factors.
.
Traffic.
2022;
;
23
:
:414.
–
25
.
34.
Villarreal
LP
,
DeFilippis
VR
.
A hypothesis for DNA viruses as the origin of eukaryotic replication proteins.
.
J Virol.
2000;
;
74
:
:7079.
–
7084
.
35.
Forterre
P.
Why are there so many diverse replication machineries?
.
J Mol Biol.
2013;
;
425
:
:4714.
–
4726
.
36.
Takemura
M
,
Yokobori
S
,
Ogata
H
.
Evolution of Eukaryotic DNA Polymerases via Interaction Between Cells and Large DNA Viruses.
.
J Mol Evol.
2015;
;
81
:
:24.
–
33
.
37.
Kazlauskas
D
,
Krupovic
M
,
Guglielmini
J
,
et al
.
Diversity and evolution of B-family DNA polymerases.
.
Nucleic Acids Res.
2020;
;
48
:
:10142.
–
56
.
38.
Guglielmini
J
,
Gaia
M
,
Da Cunha
V
,
et al
.
Viral origin of eukaryotic type IIA DNA topoisomerases.
.
Virus evolution.
2022
;
sous presse.
.
39.
Mondal
N
,
Parvin
JD
.
DNA topoisomerase II alpha is required for RNA polymerase II transcription on chromatin templates.
.
Nature.
2001;
;
413
:
:435.
–
438
.
40.
Sperling
AS
,
Jeong
KS
,
Kitada
T
,
et al.
Topoisomerase II binds nucleosome-free DNA and acts redundantly with topoisomerase I to enhance recruitment of RNA Pol II in budding yeast.
.
Proc Nat Acad Sci USA.
2011;
;
108
:
:12693.
–
12698
.
41.
Netherton
CL
,
Wileman
T
.
Virus factories, double membrane vesicles and viroplasm generated in animal cells.
.
Curr Opin Virol.
2011;
;
1
:
:381.
–
387
.
42.
Chaikeeratisak
V
,
Nguyen
K
,
Khanna
K
,
et al.
Assembly of a nucleus-like structure during viral replication in bacteria.
.
Science.
2017;
;
355
:
:194.
–
197
.
43.
Mendoza
SD
,
Nieweglowska
ES
,
Govindarajan
S
,
et al
.
A bacteriophage nucleus-like compartment shields DNA from CRISPR nucleases.
.
Nature.
2020;
;
577
:
:244.
–
8
.
44.
Wolff
G
,
Limpens
RWAL
,
Zevenhoven-Dobbe
JC
,
et al
.
A molecular pore spans the double membrane of the coronavirus replication organelle.
Science.
2020;
;
369
:
:1395.
–
8
.
45.
Sanderson
M
,
Way
M
,
Smith
GL
.
Virus-induced cell motility.
.
J Virol.
1998;
;
72
:
:1235.
–
1243
.
46.
Buchrieser
J
,
Dufloo
J
,
Hubert
M
,
et al
.
Syncytia formation by SARS-CoV-2-infected cells.
.
EMBO J.
2020;
;
39
:
:e106267.
.
47.
Liu
J
,
Cvirkaite-Krupovic
V
,
Baquero
DP
,
et al
.
Virus-induced cell gigantism and asymmetric cell division in archaea.
.
Proc Natl Acad Sci USA.
2021;
;
118
:
:e2022578118.
.
48.
Forterre
P.
The virocell concept and environmental microbiology.
.
ISME J.
2013;
;
7
:
:233.
–
236
.
49.
Heisserer
C
,
Selosse
MA
,
Drezen
JM
.
Des virus bénéfiques pour les plantes et les animaux.
.
Med Sci (Paris).
2022;
;
38
:
???
.
|