| |
| Med Sci (Paris). 39(4): 392–394. doi: 10.1051/medsci/2023046.RCC1, un nouveau partenaire stabilisant le génome du virus d’Epstein-Barr Lucie Cressot,1* Célia Galleri-Paris,1** Marine Tronchon,1*** and Samantha Vonau1§ 1École normale supérieure de Lyon, département de biologie, Master Biosciences
,
Lyon
,
France MeSH keywords: Humains, Herpèsvirus humain de type 4, Infections à virus Epstein-Barr, Protéines nucléaires, Protéines du cycle cellulaire, Facteurs d'échange de nucléotides guanyliques |
Le virus d’Epstein-Barr (EBV) est un herpèsvirus de très haute prévalence : il est estimé que 90 % de la population mondiale est infectée par l’EBV [
1
]. Le premier contact se fait le plus souvent dès le plus jeune âge, le virus se transmettant
via
les sécrétions respiratoires et la salive [
1
]. Lors d’une infection primaire, le virus peut causer une pharyngite ou de la fièvre, les symptômes variant en fonction de l’âge auquel a lieu l’infection. Le virus reste ensuite à l’état latent, en particulier dans les cellules épithéliales et dans les lymphocytes B. L’EBV est un virus oncogène qui favorise l’apparition de divers cancers, comme le lymphome de Burkitt.
Après infection, le virus persiste dans les cellules sous la forme d’une molécule d’ADN circulaire, appelée épisome. Cet épisome a la capacité de s’associer avec les chromosomes de la cellule hôte, et d’ainsi utiliser la machinerie cellulaire de réplication et de ségrégation à ses propres fins [
2
]. De cette manière, au cours des divisions cellulaires, l’épisome est répliqué en même temps que le génome cellulaire. La ségrégation de l’épisome lors de la division permet ensuite d’obtenir un unique épisome dans chaque cellule-fille. Cependant, les mécanismes moléculaires de l’interaction de l’épisome avec les chromosomes de la cellule-hôte ne sont pas encore tout à fait établis. Plusieurs protéines virales sont exprimées lors des phases de latence du virus, en particulier, la protéine EBNA1 (
Epstein-Barr virus nuclear antigen 1
) [
3
]. EBNA1 s’associe à l’épisome et permet sa réplication et sa ségrégation [
4
], mais le rôle joué dans la liaison au chromosome de la cellule hôte était jusqu’à maintenant peu connu. L’étude de Deschamps
et al.
[
4
] s’est intéressée à l’identification de protéines cellulaires interagissant avec EBNA1 et intervenant dans la réplication et la ségrégation de l’épisome d’EBV afin de comprendre le maintien du génome viral dans les cellules lors de la multiplication cellulaire. Deschamps
et al.
ont ainsi identifié RCC1 (
regulator of chromosome condensation 1
) comme un nouveau partenaire d’EBNA1 permettant de stabiliser le génome d’EBV. Les auteurs ont également caractérisé les régions impliquées dans l’interaction entre les deux protéines et analysé la dynamique de cette interaction au cours du cycle cellulaire.
|
EBNA1 interagit de manière directe et indirecte avec le chromosome mitotique
EBNA1 est capable de se lier à la fois à l’épisome viral et au génome cellulaire [
5
]. EBNA1 se lie à l’épisome d’EBV par son domaine de liaison à l’ADN situé à son extrémité C-terminale, tandis qu’elle se lie au chromosome métaphasique par son extrémité N-terminale au niveau de ses domaines CBS (
chromosome-binding site
) [
6
]
(
Figure 1
)
. Ainsi, EBNA1 permet d’associer l’épisome viral au chromosome métaphasique de manière directe.
 | Figure 1.
Structure des protéines EBNA1 et RCC1.
EBNA1 est composée d’un domaine de liaison à l’ADN (DBD :
DNA-binding domain
) et de domaines de liaison au chromosome (CBS :
chromosome-binding sites
). Ces derniers permettent aussi l’interaction avec RCC1. RCC1 est composée de sept feuillets-β, et d’extrémités C- et N-terminales. Son extrémité N-terminale permet l’interaction avec EBNA1.
|
En plus de sa capacité de liaison directe à l’ADN, des études ont montré qu’EBNA1 était également capable de se fixer indirectement à l’ADN,
via
des interactions avec des protéines cellulaires. Différentes protéines ont été identifiées, dont hEBP2 (
human Epstein-Barr-binding protein 2
) et HMGB2 (
high mobility group box 2
). Cependant, EBNA1 n’interagit pas avec ces protéines durant le cycle cellulaire et leur délétion n’altère pas le maintien de l’épisome dans la cellule. Les auteurs ont donc cherché à identifier une autre protéine capable de se lier à la fois à EBNA1 et au génome cellulaire.
|
EBNA1 et RCC1 interagissent directement
Pour cela, les auteurs ont utilisé une technique de criblage permettant d’identifier les protéines interagissant avec EBNA1. Parmi les protéines testées, les auteurs ont identifié RCC1 (
regulator of chromosome condensation 1
). Capable de se lier à la fois à la chromatine [
7
] et à EBNA1, RCC1 est ainsi un bon candidat pour expliquer le maintien du génome d’EBV sur les chromosomes mitotiques.
Dans un premier temps, Deschamps
et al.
ont confirmé que EBNA1 et RCC1 interagissaient directement. Par des expériences de GST(
glutathione S-transferase
)-
pulldown
, ils ont montré que la protéine recombinante EBNA1 pouvait être capturée spécifiquement par RCC1 à la surface de microbilles, ce qui suggère une liaison de EBNA1 à RCC1, sans protéine intermédiaire. Par la suite, Deschamps
et al.
ont caractérisé les domaines de liaison intervenant dans cette interaction.
|
EBNA1 interagit avec RCC1 par ses domaines CBS-3/-1 et CBS-2
EBNA1 interagit avec les chromosomes mitotiques par l’intermédiaire de trois régions connues : CBS-1, CBS-2 et CBS-3 (
chromosome-binding sites
-1, -2 et -3) [
6
]. Les domaines CBS-3 et CBS-1, proches l’un de l’autre et situés dans la région N-terminale de EBNA1, ont été analysés conjointement
(
Figure 1
)
. Par la même technique d’analyse des interactions protéine-protéine, les auteurs ont montré en utilisant différents mutants EBNA1 dans lesquels les domaines CBS-3/-1 ou CBS-2 ont été supprimés, que la présence d’une seule de ces deux régions est suffisante au maintien de l’interaction entre EBNA1 et RCC1. En revanche, si les deux régions sont supprimées, EBNA1 et RCC1 n’interagissent plus. Les CBS-1/-3 et CBS-2 sont donc nécessaires pour que l’interaction de EBNA1 avec RCC1 puisse avoir lieu.
|
RCC1 interagit avec EBNA1 par l’intermédiaire de ses vingt derniers acides aminés de son extrémité N-terminale
Les auteurs se sont ensuite intéressés aux domaines de liaison à EBNA1 présents sur RCC1. RCC1 est composée d’une hélice-β à sept feuillets et d’extrémités C- et N- terminales non structurées [
8
]
(
Figure 1
)
. L’extrémité N-terminale permet les interactions de RCC1 avec la chromatine et avec d’autres protéines [
8
] et est la seule partie de RCC1 qui soit modifiable sans avoir d’impact sur sa structure et son intégrité. De nouvelles expériences de GST-
pulldown
permettant d’analyser l’interaction entre EBNA1 et une protéine RCC1 dont l’extrémité N-terminale a été supprimée ont été réalisées. Grâce à ces expériences, Deschamps
et al.
ont non seulement montré que l’extrémité N-terminale de RCC1 interagit fortement avec EBNA1, mais aussi que cette interaction se fait grâce aux vingt premiers acide-aminés de RCC1
(
Figure 1
)
.
|
L’interaction entre EBNA1 et RCC1 est dynamique au cours du cycle cellulaire
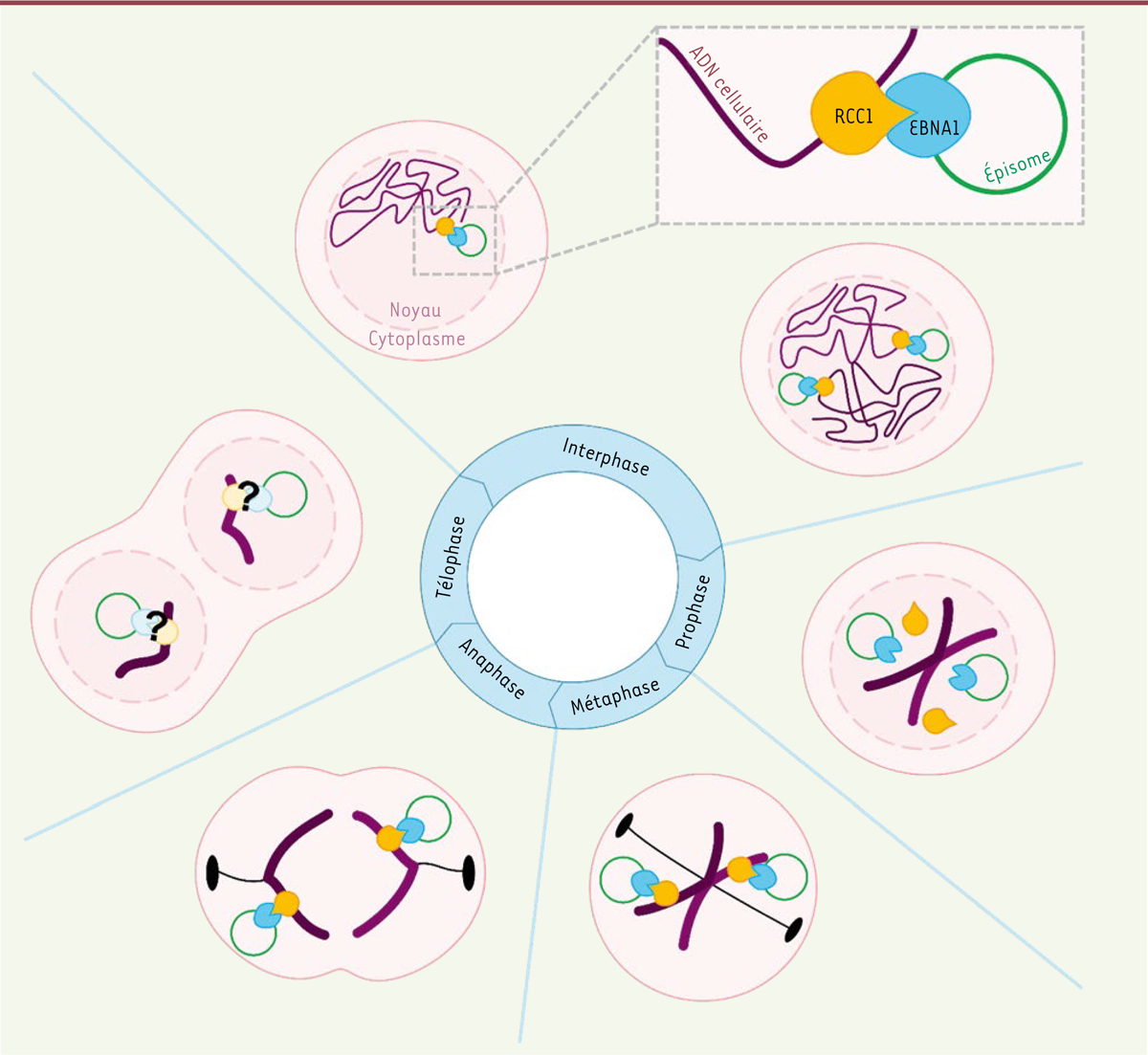
Pour finir, une analyse par imagerie de type FRET (
Förster resonance energy transfer
) a permis d’étudier l’évolution de l’interaction entre EBNA1 et RCC1 au cours du cycle cellulaire. En comparant les données obtenues pendant les différentes phases du cycle cellulaire, les auteurs ont montré que RCC1 et EBNA1 interagissent fortement pendant l’interphase et la métaphase, mais peu pendant la prophase
(
Figure 2
)
. Deschamps
et al.
ont ainsi montré que l’interaction entre EBNA1 et RCC1 est dynamique au cours du cycle cellulaire et que les phases d’interactions concordent avec le maintien du génome de l’EBV au sein des cellules.
 | Figure 2.
Schéma des possibles interactions entre EBNA1 et un chromosome pendant la mitose.
Après entrée de l’épisome dans le noyau, EBNA1 va permettre à l’épisome d’être lié au génome de la cellule hôte. Deschamps
et al.
ont montré que, durant l’interphase, RCC1 et EBNA1 interagissent, ce qui permettrait la stabilisation de l’épisome et sa réplication en même temps que la réplication du chromosome de la cellule hôte. Au moment de la prophase, les deux protéines ne semblent pas interagir, alors que lors de la métaphase, RCC1 lie l’épisome au chromosome métaphasique grâce à sa liaison avec EBNA1. Lors de l’anaphase et de la télophase, l’interaction n’a pas été étudiée. Néanmoins, EBNA1 permettant le maintien de l’épisome dans les cellules en division, RCC1 pourrait faire partie des protéines aidant à ce mécanisme. On pourrait donc imaginer que EBNA1 et RCC1 interagissent durant l’anaphase et la télophase (cette figure a été partiellement réalisée à l’aide de « Servier Medical Art » fourni par Servier, sous licence Creative Commons Attribution 3.0).
|
Ainsi, ces résultats suggèrent que RCC1 et EBNA1 interagissent pendant l’interphase et favorisent la réplication de l’épisome en même temps que la réplication du génome de la cellule-hôte par recrutement de la machinerie de l’hôte. RCC1 et EBNA1 interagissent aussi pendant la métaphase. Cela permet une bonne ségrégation de l’épisome au sein des cellules, résultant en un unique épisome par cellule-fille après division. |
Cette étude démontre que l’interaction entre RCC1 et EBNA1 est importante pour maintenir l’épisome dans la cellule-hôte et pour stabiliser l’interaction entre EBNA1 et le chromosome métaphasique avant la séparation des chromatides, facilitant ainsi la ségrégation de l’épisome au cours des divisions cellulaires. Ainsi, l’interaction entre RCC1 et EBNA1 pourrait être une nouvelle cible thérapeutique. En effet, limiter le maintien de l’épisome dans la cellule hôte permettrait de cibler la phase latente des infections à EBV. |
Les auteures déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Nowalk
A
,
Green
M
.
Epstein-Barr Virus.
.
Microbiology Spectrum.
2016;
;
4
:
4.3.47.
.
2.
Nanbo
A
,
Sugden
A
,
Sugden
B
.
The coupling of synthesis and partitioning of EBV’s plasmid replicon is revealed in live cells.
.
EMBO J.
2007;
;
26
:
:4252.
–
4262
.
3.
Epstein
MA
,
Rickinson
AB
,
Weiss
RA
,
et al.
Biology and disease associations of Epstein-Barr virus.
.
Philos Trans R Soc Lond B Biol Sci.
2001;
;
356
:
:461.
–
473
.
4.
Deschamps
T
,
Bazot
Q
,
Leske
DM
,
et al.
Epstein-Barr virus nuclear antigen 1 interacts with regulator of chromosome condensation 1 dynamically throughout the cell cycle.
.
J Gen Virol.
2017;
;
98
:
:251.
–
265
.
5.
Rawlins
DR
,
Milman
G
,
Hayward
SD
,
et al.
Sequence-specific DNA binding of the Epstein-Barr virus nuclear antigen (EBNA-1) to clustered sites in the plasmid maintenance region.
.
Cell.
1985;
;
42
:
:859.
–
868
.
6.
Marechal
V
,
Dehee
A
,
Chikhi-Brachet
R
,
et al.
Mapping EBNA-1 Domains Involved in Binding to Metaphase Chromosomes.
.
J Virol.
1999;
;
73
:
:4385.
–
4392
.
7.
Makde
RD
,
England
JR
,
Yennawar
HP
,
et al.
Structure of RCC1 chromatin factor bound to the nucleosome core particle.
.
Nature.
2010;
;
467
:
:562.
–
566
.
8.
Ren
X
,
Jiang
K
,
Zhang
F
.
The Multifaceted Roles of RCC1 in Tumorigenesis.
.
Front Mol Biosci.
2020;
;
7
:
:225.
.
|