| |
| Med Sci (Paris). 39(6-7): 496–499. doi: 10.1051/medsci/2023070.Paléogénomique et évolution du système immunitaire humain au cours des dix derniers millénaires Gaspard Kerner1* and Lluís Quintana-Murci1,2** 1Institut Pasteur, université Paris Cité, CNRS UMR2000, unité de génétique évolutive humaine
,
Paris
,
France 2Collège de France, Chaire génomique humaine et évolution
,
Paris
,
France MeSH keywords: Humains, Génomique, Paléontologie |
L’essor de nouvelles techniques de séquençage de l’ADN fossile est à l’origine d’une discipline émergente connue sous le nom de paléogénomique, et a profondément bouleversé les études de génétique humaine, génétique des populations et évolution. Ces techniques permettent de répondre à des questions majeures en archéologie ou en paléoanthropologie, comme celles concernant l’évolution et l’expansion de l’espèce humaine ainsi que les métissages avec des populations humaines aujourd’hui disparues, telles que les Néandertaliens. Elles ont également permis de découvrir de nouveaux mécanismes sous-jacents à la domestication des animaux ou à l’évolution des espèces sauvages en réponse aux changements climatiques [
1
–
3
]. Mais la paléogénomique peut aussi éclairer la recherche médicale en retraçant certains avatars de santé des populations humaines du passé, notamment des épidémies, par l’analyse d’échantillons anciens.
|
Les microorganismes pathogènes ont façonné l’évolution des génomes humains
Dans les années 1950, le généticien John Burdon Sanderson Haldane explique le maintien d’une mutation du gène de l’hémoglobine responsable d’une anomalie grave des globules rouges communément observée en Afrique, la drépanocytose, par la protection qu’elle confère contre le paludisme [
4
]. Cette découverte identifie l’infection par des microorganismes pathogènes comme une des pressions sélectives, les plus fortes subies par les populations humaines, les obligeant à une adaptation coûteuse en termes de santé. De nombreuses études de génétique des populations ont par la suite confirmé cette hypothèse en identifiant des centaines de gènes impliqués dans l’immunité et soumis à la sélection naturelle au cours de l’histoire de l’humanité [
5
,
6
]. Cependant, ces études, fondées sur l’analyse de l’ADN « moderne », n’offrent qu’une vision partielle de la « course aux armements » qui caractérise la coévolution entre populations humaines et agents infectieux. Par exemple, on connaît mal les époques durant lesquelles les pressions sélectives exercées par les microorganismes pathogènes ont été les plus fortes, et qui devraient nous renseigner sur les épidémies du passé les plus meurtrières, ou bien sur les conséquences de ces pressions de sélection sur le risque actuel de développer d’autres maladies.
|
La paléogénomique comme mesure de la sélection naturelle en temps réel
La paléogénomique peut permettre de répondre à ces questions en étudiant par exemple l’évolution de la diversité génétique d’une population au cours du temps. On peut ainsi identifier des changements génétiques ayant eu lieu au temps d’un évènement épidémique majeur, tel que la peste, et détecter par conséquent des variants génétiques ayant contribué à la survie de certains individus [
7
,
14
] (
→
).
(→) Voir la Nouvelle de C.E. Demeure
et al
.,
m/s
n° 4, avril 2023, page 19
Dans une étude récente [
8
], nous avons eu recours à une grande collection de génomes anciens couvrant les dix derniers millénaires de l’histoire européenne pour tenter de mieux comprendre le rôle des maladies infectieuses du passé dans le façonnement des gènes de l’immunité et l’impact conséquent sur la santé humaine actuelle. Ces échantillons ont été majoritairement collectés sur le territoire européen, ce qui témoigne, d’une part, d’un biais archéologique lié à la conservation des échantillons, qui est d’autant meilleure dans les climats froids et secs, mais aussi d’un biais plus général favorisant l’investigation de personnes d’origine européenne, notamment dans les études génomiques. Nous avons ainsi comparé la diversité de 2 376 génomes anciens récupérés sur le territoire européen à celle de 503 génomes modernes d’individus d’origine européenne. Sur cette base, nous avons estimé les « trajectoires de fréquence » – c’est-à-dire les changements de fréquence allélique d’un variant génétique au cours du temps au sein d’une population – de 1 233 013 sites polymorphes du génome humain à travers les périodes du Néolithique (ici, entre 8 500 et 5 000 ans auparavant), de l’âge du Bronze (5 000 - 2 500 ans auparavant), de l’âge du Fer (2 500 - 1 250 ans auparavant), du Moyen-âge (1 250 - 750 ans auparavant) et du temps présent. En utilisant des données simulées à partir d’un modèle démographique conforme à l’histoire européenne et la méthode statistique ABC (
approximate Bayesian computation
) [
9
], nous avons repéré les sites polymorphes soumis à la sélection naturelle et estimé, pour chacun de ces sites, l’intensité et le temps du début de cette sélection. Cette méthode est fondée sur la comparaison de trajectoires de fréquences attendues dans un modèle de neutralité évolutive (aucun avantage sélectif) à celles observées, et permet donc de révéler des allèles dont la fréquence a augmenté au cours du temps par sélection positive (allèle « avantageux ») ou, au contraire, diminué par sélection négative (allèle « désavantageux »).
|
Le fardeau des maladies infectieuses s’est alourdi à partir de l’âge du Bronze
Nous avons identifié 89 gènes porteurs de mutations présentant des signes de forte sélection positive, ce qui suggère que ces mutations ont conféré un avantage évolutif considérable aux individus du passé. Ces gènes incluent notamment le gène de la lactase, qui permet aux populations humaines de digérer le lait même à l’âge adulte (
Figure 1A
), ou encore des gènes impliqués dans la pigmentation, à l’origine de la couleur de peau plus claire chez les Européens. Toutefois, il est intéressant de noter que cet ensemble de 89 gènes est enrichi en gènes impliqués dans l’immunité innée, la première ligne de défense de l’hôte contre les microorganismes pathogènes. Tel est le cas du complexe de gènes
OAS
(
2’-5’-oligoadenylate synthetase
), qui agit dans la réponse antivirale en limitant la réplication du virus, ou encore du « gène
ABO
» (codant une glycosyl-transférase), dont le produit est à l’origine du système des groupes sanguins A, B et O. La datation des évènements de sélection positive agissant sur ces sites du génome, qui correspond généralement au moment de plus grand accroissement de la fréquence du variant génétique avantageux, a révélé un biais important en faveur de temps relativement récents. Plus de 80 % des évènements de sélection positive détectés ont en effet un « âge de sélection » estimé après le début de l’âge du Bronze, c’est-à-dire au cours des 4 500 dernières années, ce qui peut être expliqué par une plus forte croissance démographique des populations européennes à partir de cette période, mais aussi, ce qui n’est pas incompatible, par une plus forte présence de microorganismes pathogènes meurtriers depuis le début de l’âge du Bronze.
 | Figure 1.
Trajectoires temporelles de fréquence allélique pour des allèles soumis à une sélection positive ou négative.
Trajectoires de fréquence allélique à travers cinq périodes historiques, représentées par des intervalles de temps en « années auparavant » (
ya : years ago
) sur l’axe des abscisses, pour
(A)
un variant du gène de la lactase (rs4988235,
LCT
) soumis à une sélection positive, et
(B)
un variant du gène
TYK2
(rs34536443) soumis à une sélection négative, probablement, car il est associé à un risque accru de développer une tuberculose.
T
indique l’âge du début de la pression de sélection positive ou négative, et la valeur de
s
(+ ou -) reflète son intensité. Pour chaque point de fréquence allélique correspondant à chaque intervalle de temps est indiqué le nombre d’échantillons anciens (nombre d’individus) utilisés pour estimer cette fréquence, et le polygone gris indique l’intervalle de confiance à 95 % de l’estimation.
|
La sélection naturelle peut également opérer en éliminant des variants génétiques qui ont un effet délétère sur des agents essentiels de l’immunité. Ce processus de « sélection négative » peut notamment se produire au cours d’une épidémie, avec l’élimination de mutations associées à un risque accru de développer la maladie infectieuse. Nous avons ainsi pu identifier six variants génétiques localisés dans des gènes de l’immunité, dont la fréquence a significativement diminué au cours des derniers millénaires. Par exemple, nous avons identifié un variant du gène
TYK2
(
tyrosine kinase 2
) qui, à l’état homozygote, multiplie par dix le risque de développer une tuberculose à la suite d’une infection par
Mycobacterium tuberculosis
(
Figure 1B
) [
10
]. Selon notre estimation, la sélection négative a agi sur ce variant seulement au cours des 2 000 dernières années [
11
] : le fardeau imposé par la tuberculose à la santé des populations européennes semble donc plus récent qu’on ne le pensait jusqu’à présent. Trois autres de ces six mutations soumises à une sélection négative sont situées dans les gènes
LBP
(
LPS-binding protein
),
IL23R
(
interleukin 23 receptor
) et
TLR3
(
Toll-like receptor 3
), qui sont impliqués dans trois voies de signalisation majeures du système immunitaire associées à la défense de l’hôte contre des agents pathogènes respectivement bactériens, mycobactériens et viraux. Nous avons montré que ces trois mutations, qui entraînent toutes un changement d’acide aminé dans la séquence de la protéine codée, ont un effet délétère sur cette protéine ou sur la voie de signalisation correspondante, ce qui suggère que les porteurs de ces variants peuvent présenter un risque accru de développer une forme grave de maladie infectieuse.
|
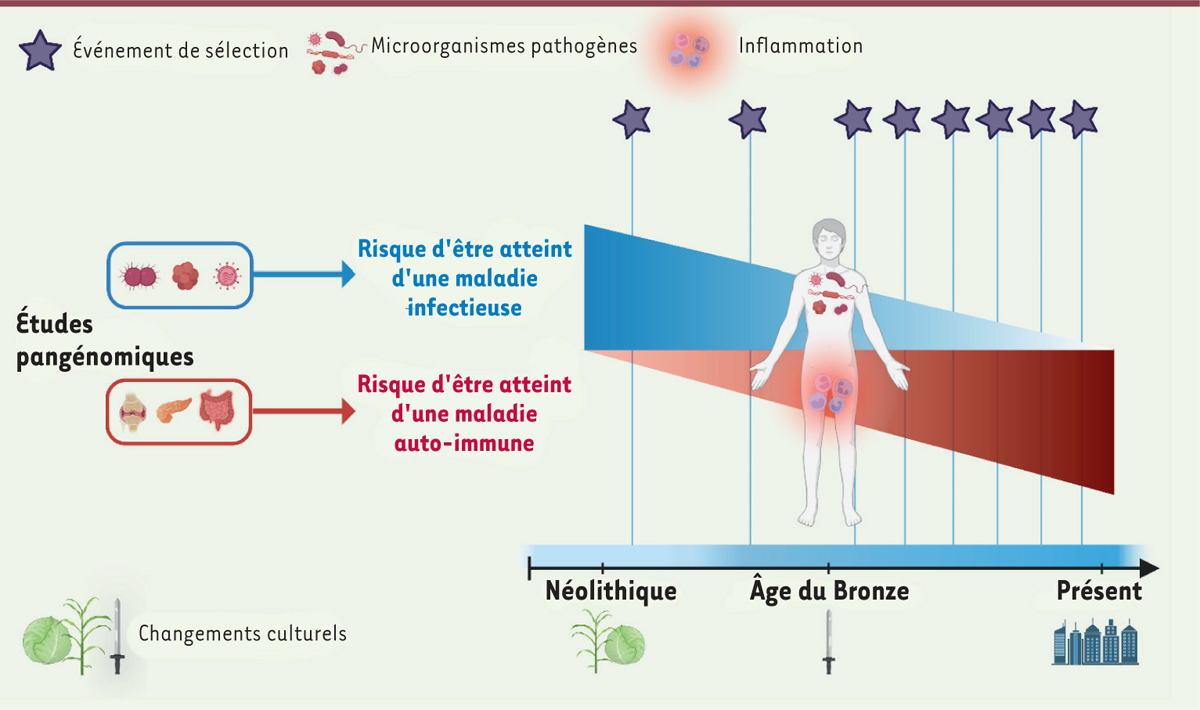
La sélection naturelle :une arme à double tranchant
La sélection naturelle n’est pas toujours sans conséquence négative pour l’espèce concernée. En effet, la plupart des gènes ont un effet pléiotropique, c’est-à-dire que la protéine qu’ils codent influe sur plusieurs traits phénotypiques. Une mutation génétique peut donc s’avérer bénéfique pour un trait phénotypique, mais désavantageuse pour un autre (pléiotropie antagoniste). Ainsi, un nombre croissant de mutations associées au risque de contracter certaines maladies infectieuses sont également associées au risque de développer des maladies auto-immunes ou inflammatoires chroniques, telles que le diabète de type I ou la maladie de Crohn, souvent avec des effets opposés, avantageux dans un cas et désavantageux dans l’autre [
12
,
13
]. En étudiant la façon dont la sélection naturelle a ciblé les variants génétiques associés à la susceptibilité aux maladies infectieuses d’une part, et aux maladies auto-immunes ou inflammatoires chroniques d’autre part, nous avons essayé de reconstruire l’évolution du risque d’être atteint de ces maladies au cours des dix derniers millénaires, jusqu’à la période actuelle. Tout d’abord, nous avons constaté que ce risque a considérablement diminué pour les maladies infectieuses, alors qu’il a augmenté pour les maladies auto-immunes et inflammatoires chroniques (
Figure 2
). Nous avons par ailleurs noté que le nombre de variants génétiques associés (dans un sens ou dans l’autre) à la fois au risque de maladie infectieuse et à celui de maladie inflammatoire chronique était plus important que ne le prédisait le simple hasard. Finalement, nous avons remarqué qu’un nombre significatif de ces variants étaient soumis à une sélection. Notre analyse apporte donc des preuves d’une contribution considérable de la pléiotropie antagoniste à l’émergence des maladies chroniques modernes par l’effet de la sélection positive de variants génétiques conférant une protection contre des maladies infectieuses, et cela dès la fin de la période néolithique, donc, contrairement à ce que l’on pensait précédemment, bien avant l’introduction des vaccins et des antibiotiques.
 | Figure 2.
Illustration de la pléiotropie antagoniste à l’origine d’un risque accru de maladies inflammatoires chroniques ou auto-immunes chez les Européens d’aujourd’hui.
Schéma représentant l’évolution des risques d’être atteint d’une maladie infectieuse (en bleu) ou auto-immune (en rouge) depuis le Néolithique jusqu’au temps présent (axe horizontal). L’estimation des risques est obtenue à partir d’études pan-génomiques pour divers traits phénotypiques associés soit aux maladies infectieuses soit aux maladies auto-immunes. Depuis le Néolithique, plusieurs évènements de sélection positive ont eu lieu, dont la fréquence a augmenté après le début de l’âge du Bronze. Ces évènements ont ciblé des variants génétiques protecteurs contre les maladies infectieuses mais aussi perturbateurs du système immunitaire, expliquant la diminution du risque infectieux au cours du temps (triangle bleu) et l’augmentation du risque de maladie auto-immune (triangle rouge) (figure créée avec le logiciel BioRender.com).
|
|
Le futur de la paléogénomique dans la recherche médicale L’amélioration constante des techniques d’extraction d’ADN de haute qualité provenant d’échantillons fossiles permet d’envisager une contribution croissante de la paléogénomique à la recherche médicale. Cependant, en raison de la nature souvent dégradée de l’ADN ancien, la plupart des génomes humains anciens disponibles ne permettent d’identifier qu’environ 1,2 million de variants génétiques relativement fréquents. L’utilisation de techniques alternatives de séquençage de l’ADN, à l’échelle du génome entier, devrait faciliter la détection de variants qui, sous l’effet d’une forte sélection négative, sont devenus rares, voire ont disparu dans les populations actuelles. Ainsi pourront être identifiés de nouveaux gènes dont certains variants alléliques ont un effet délétère sur la santé, ce qui nous permettra de progresser dans la compréhension des facteurs génétiques impliqués dans les disparités actuelles de santé, à l’échelle des individus ou des populations humaines. |
Cette étude a été financée par l’Institut Pasteur, le Collège de France, le CNRS, l’Agence nationale de la recherche (LIFECHANGE-ANR-CE-1712-0018-02, CNSVIRGEN-ANR-19-CE15-0009-02, et MORTUI-ANR-19-CE35-0005), les Laboratoires d’excellence “Integrative biology of emerging infectious diseases” (ANR-10-LABX-62-IBEID) et “Milieu intérieur” (ANR-10-LABX-69-01), la Fondation pour la recherche médicale (FRM DEQ20180339214), la Fondation Allianz-Institut de France, et la Fondation de France (00106080). Gaspard Kerner a bénéficié d’une bourse Pasteur-Roux-Cantarini.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Dannemann
M
,
Racimo
F
.
Something old, something borrowed : admixture and adaptation in human evolution.
.
Curr Opin Genet Dev.
2018;
;
53
:
:1.
–
8
.
2.
Gittelman
RM
,
Schraiber
JG
,
Vernot
B
,
et al.
Archaic hominin admixture facilitated adaptation to out-of-Africa environments.
.
Curr Biol.
2016;
;
26
:
:3375.
–
3382
.
3.
Skoglund
P
,
Mathieson
I
.
Ancient genomics of modern humans : The first decade.
.
Annu Rev Genom Hum Genet.
2018;
;
19
:
:381.
–
404
.
4.
Allison
AC
.
Protection afforded by sickle-cell trait against subtertian malareal infection.
.
Br Med J.
1954;
;
1
:
:290.
–
294
.
5.
Barreiro
LB
,
Quintana-Murci
L
.
From evolutionary genetics to human immunology : how selection shapes host defence genes.
.
Nat Rev Genet.
2010;
;
11
:
:17.
–
30
.
6.
Quintana-Murci
L.
Human immunology through the lens of evolutionary genetics.
.
Cell.
2019;
;
177
:
:184.
–
199
.
7.
Klunk
J
,
Vilgalys
TP
,
Demeure
CE
,
et al
.
Evolution of immune genes is associated with the Black Death.
.
Nature.
2022;
;
611
:
:312.
–
9
.
8.
Kerner
G
,
Neehus
AL
,
Philippot
Q
,
et al
.
Genetic adaptation to pathogens and increased risk of inflammatory disorders in post-Neolithic Europe.
.
Cell Genom.
2023;
;
3
:
:100248.
.
9.
Beaumont
MA
,
Rannala
B
.
The Bayesian revolution in genetics.
.
Nat Rev Genet.
2004;
;
5
:
:251.
–
261
.
10.
Kerner
G
,
Ramirez-Alejo
N
,
Seeleuthner
Y
,
et al.
Homozygosity for TYK2 P1104A underlies tuberculosis in about 1 % of patients in a cohort of European ancestry.
.
Proc Natl Acad Sci USA.
2019;
;
116
:
:10430.
–
10434
.
11.
Kerner
G
,
Laval
G
,
Patin
E
,
et al
.
Human ancient DNA analyses reveal the high burden of tuberculosis in Europeans over the last 2,000 years.
.
Am J Hum Genet.
2021;
;
108
:
:517.
–
24
.
12.
Fumagalli
M
,
Pozzoli
U
,
Cagliani
R
,
et al.
Parasites represent a major selective force for interleukin genes and shape the genetic predisposition to autoimmune conditions.
.
J Exp Med.
2009;
;
206
:
:1395.
–
1408
.
13.
Langlais
D
,
Fodil
N
,
Gros
P
.
Genetics of infectious and inflammatory diseases : Overlapping discoveries from association and exome-squencing studies.
.
Annu Rev Immunol.
2017;
;
35
:
:1.
–
30
.
14.
Demeure
CE
,
Poinar
H
,
Barreiro
L
,
Pizarro-Cerdá
J
.
La peste noire, la sélection naturelle et notre sensibilité aux maladies auto-immunes ou inflammatoires.
.
Med Sci (Paris).
2023;
;
39
:
:19.
–
21
.
|