L’enveloppe cellulaire est un composant essentiel des bactéries. Elle est fondamentale pour le maintien de la forme cellulaire, la résistance aux conditions difficiles, voire extrêmes, et la détection des signaux environnementaux. Porteuse de nombreux systèmes de défense et d’attaque, l’enveloppe participe aussi à la pathogénicité bactérienne.
Il existe deux profils majoritaires d’enveloppes bactériennes [ 1 ]. Chez les bactéries à Gram négatif comme Escherichia coli, l’enveloppe est composée d’une membrane plasmique, d’une mince couche de peptidoglycane (PG) contenue dans l’espace périplasmique et d’une membrane externe. Chez les bactéries à Gram positif comme Bacillus subtilis , l’enveloppe est composée d’une membrane plasmique et d’une couche épaisse de PG, qui est alors le constituant majeur de la paroi ( Figure 1A ).
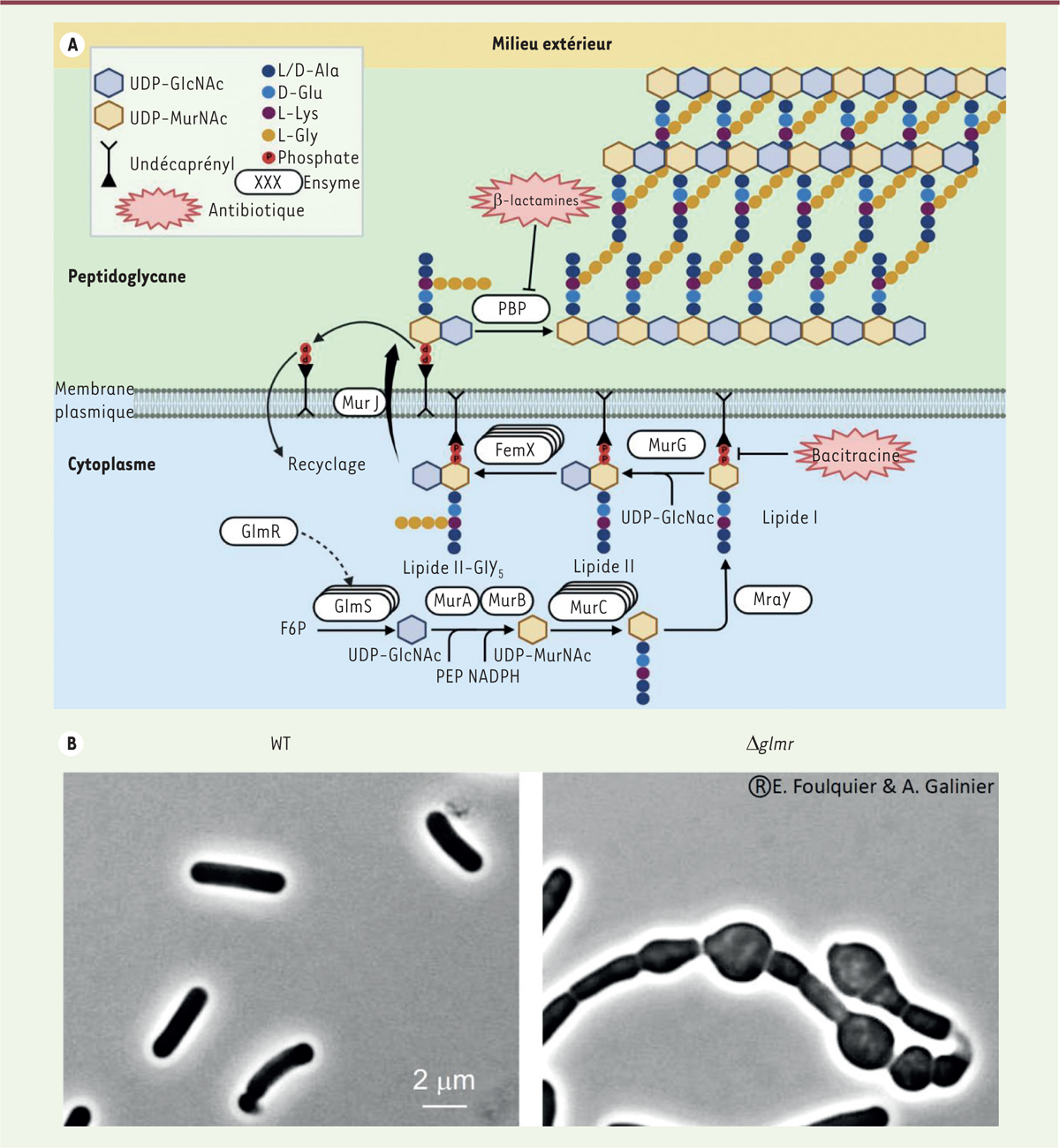
| Figure 1.
La biosynthèse du peptidoglycane chez Bacillus subtilis. (A)
La formation du PG débute dans le cytoplasme avec la synthèse des précurseurs. L’UDP-GlcNac [hexagone bleu] et l’UDP-MurNac [hexagone jaune] sont les deux principaux précurseurs. La GlmR interagit d’abord avec la GlmS [flèche discontinue] et stimule la synthèse de l’UDP-GlcNac. Ensuite, l’UDP-MurNAc est lié à une chaîne peptidique après plusieurs réactions au cours desquelles différentes enzymes interviennent. La synthèse se poursuit au niveau de la membrane plasmique avec la formation du lipide I par fusion du phospho-MurNAc-pentapeptide avec l’undécaprényl phosphate lié à la membrane. La fusion de l’UDP-GlcNAc au lipide I forme le lipide II. Ce dernier est ensuite transloqué par une flippase du côté extérieur de la membrane. Il est pris en charge par les
penicilin-binding proteins
(PBP), qui permettent son insertion dans le PG mature ainsi que le clivage de l’undécaprényl phosphate qui est ensuite recyclé. Chaque ovale blanc représente une enzyme impliquée dans une étape de la biosynthèse. Certains antibiotiques comme les β-lactamines inhibent l’action des PBP, ce qui bloque l’incorporation du lipide II au peptidoglycane préexistant. La bacitracine, quant à elle, bloque la translocation des précurseurs en empêchant leur fixation à l’undécaprényl phosphate (figure réalisée avec Biorender).
(B)
Images de microscopie à contraste de phase de bactéries
B. subtilis
sauvages (WT, à gauche) ou dépourvues du gène
glmR
(Δ
glmR,
à droite), cultivées en présence de gluconate [
4
].
|
Permettant de maintenir l’intégrité cellulaire en formant une réelle barrière protectrice, le PG est un polymère glyco-peptidique qui forme un réseau tridimensionnel au sein de l’enveloppe. En effet, ce dernier est composé d’un enchevêtrement de chaînes glycanes complexes reliées par des ponts peptidiques. De nombreuses étapes sont nécessaires pour obtenir la structure finale du peptidoglycane à partir de précurseurs cytoplasmiques tels que l’uridine diphosphate N-acétylglucosamibe (UDP-GlcNAc) et l’acide uridine-diphosphate-N-acétylmuramique (UDP-MurNac), des nucléotides couplés à des oses. Ces précurseurs sont exportés à l’extérieur de la membrane pour être assemblés par les « penicillin-binding proteins » (PBP) et reliés entre eux par des ponts peptidiques. Les PBP sont donc des enzymes essentielles assurant le maillage du PG. Elles sont majoritairement ciblées par des antibiotiques tels que la pénicilline qui appartient à la famille des bêta-lactamines. Puisqu’il est conservé chez la majorité des bactéries [ 2 ], le PG est une bonne cible thérapeutique pour le développement de nouvelles molécules antibiotiques à large spectre.
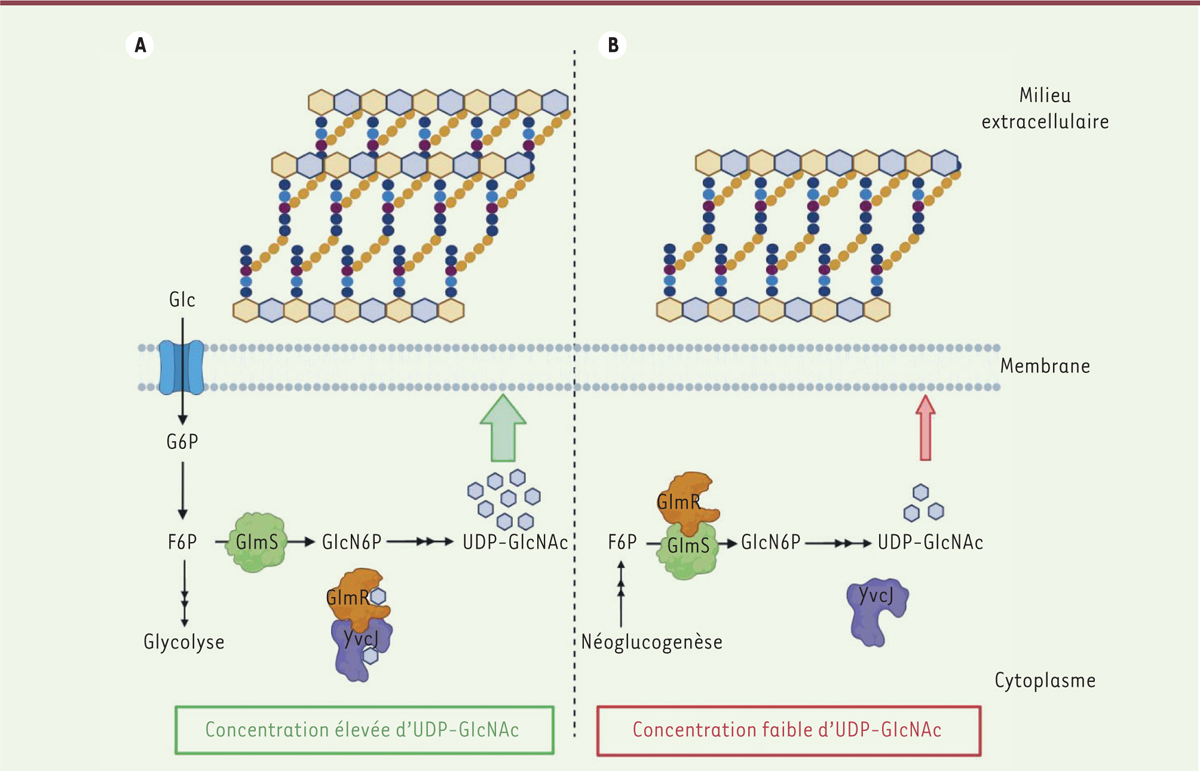
Étant une structure complexe, la paroi implique une synthèse faisant intervenir plusieurs précurseurs et nécessite un apport conséquent en énergie. Au cours de cette synthèse, des intermédiaires moléculaires qui proviennent du métabolisme du carbone, comme l’UDP-GlcNac et l’UDP-MurNac, sont utilisés pour la synthèse de composants lipidiques précurseurs du PG, après leur fixation sur l’undécaprényl diphosphate, donnant successivement les lipides I et II ( Figure 1A ). Ainsi, les mécanismes de synthèse et de dégradation du PG doivent être finement régulés pendant le cycle cellulaire bactérien en fonction de la biodisponibilité en carbone et sont donc étroitement liés au métabolisme central. L’analyse des mécanismes permettant la synchronisation de la synthèse du PG au métabolisme cellulaire global est donc cruciale pour une meilleure compréhension de la formation de cette structure essentielle des bactéries. Récemment, la protéine GlmR (glucosamine-6-phosphate regulator) a été identifiée comme une protéine clé modulant la synthèse du PG en fonction de la nature des substrats carbonés présents dans le milieu [ 3 ]. La GlmR est particulièrement étudiée chez Bacillus subtilis , une bactérie ubiquitaire aérobie stricte en forme de bacille. Cette bactérie est non pathogène, facile à cultiver et à manipuler, ce qui en fait un modèle de choix pour l’étude des bactéries à Gram positif [ 3 ]. La GlmR est également présente chez de nombreuses autres bactéries, notamment les bactéries pathogènes Listeria monocytogenes et Staphylococcus aureus . Récemment, le rôle de la GlmR a été élucidé et un modèle de son mode d’action a été proposé [ 4 ].