Le cancer du sein touche environ deux millions de femmes chaque année dans le monde. Il est une cause majeure de décès liés au cancer. Il s’agit d’une maladie hétérogène, qui comporte différentes formes moléculaires nécessitant des approches thérapeutiques spécifiques. Si le taux global de survie atteint 70 à 80 % pour les stades précoces, les formes avancées de la maladie restent incurables, avec une durée de survie limitée malgré les traitements existants.
L’existence d’un défaut de la recombinaison homologue ( homologous recombination , HR) de l’ADN dans les cellules tumorales entraîne une forte instabilité génomique, et rend ces cellules sensibles aux inhibiteurs de la poly(ADP-ribose) polymérase (PARPi) in vitro [ 1 , 2 ]. Ce traitement a montré une amélioration de la survie sans progression pour les cancers du sein métastatiques et comportant une mutation d’un gène BRCA ( breast cancer ) [ 3 , 4 ]. Connaître le statut HR des cancers du sein devient donc indispensable pour orienter le traitement des femmes concernées. Cependant, les tests moléculaires permettant de déterminer ce statut sont coûteux, et la logistique pour les réaliser chez toutes les patientes demeure un véritable défi.
Les lames histologiques des cancers du sein sont utilisées en routine médicale pour poser le diagnostic et définir les marqueurs pronostiques et théranostiques 1 nécessaires à la prise en charge des patientes. De plus, leur numérisation devient possible à faible coût. Dans ce contexte, nous avons cherché à prédire le statut HR à partir des images numériques de lames histologiques de cancer du sein, en utilisant des algorithmes d’apprentissage profond ( deep learning ). Ces algorithmes « apprennent » automatiquement, à partir de paires constituées d’images histologiques et du statut HR, des règles de prédiction, efficaces mais difficilement interprétables : ils sont donc souvent qualifiés de « boîte noire ». Pour cette raison, au-delà des étapes de développement et de validation rigoureuse de ces algorithmes, nous avons introduit une méthode permettant leur interprétation. Cette méthode nous permet d’extraire les régions de tissus ayant eu le plus d’influence sur la décision de l’algorithme.
Pour développer cette méthode et en valider l’utilisation médicale, nous avons analysé les lames numérisées de cancers du sein de 714 patientes traitées par chirurgie première. Nous avons par ailleurs établi le statut HR de la tumeur (HRD ou HRP) en utilisant deux approches différentes : soit en recherchant la principale cause connue de HRD par séquençage des gènes BRCA dans les cellules du sang ou dans celles de la tumeur 2 , soit en analysant les conséquences d’une HRD grâce au séquençage complet du génome pour identifier les réarrangements de grande taille ( large state transition , LST) de l‘ADN [ 5 ]. Les images numériques des lames de cancer du sein sont de grande taille (plusieurs gigaoctets), contenant des milliards de pixels et jusqu’à plusieurs dizaines de milliers de cellules. Une des premières étapes de l’analyse est donc de les diviser en éléments utilisables par nos cartes graphiques : les tuiles (ou instances), qui sont de petites images carrées de 224 pixels de côté. Chaque tuile est ensuite transformée en un unique vecteur de représentation en utilisant le modèle auto-supervisé MoCo [ 6 ] sur 16 millions de tuiles extraites de notre jeu de données. Ces tuiles sont ensuite utilisées pour entraîner un algorithme capable de trier les tuiles importantes, d’agréger leurs informations en une représentation globale de l’image de la tumeur, et de classifier cette représentation en fonction du statut HR de la tumeur. En utilisant ces représentations compactes des tuiles (256 valeurs) plutôt que les images elles-mêmes, nous avons considérablement réduit le temps de calcul. Ce type d’algorithme, qui tire parti de l’information inégalement répartie dans les différentes tuiles pour prédire une caractéristique globale, fait partie des algorithmes « d’apprentissage à instances multiples ». Les performances du modèle ont été évaluées par validation croisée, et les résultats sur la base de données entière sont très prometteurs, avec une aire sous la courbe ROC (AUROC) 3 de 86 %.
Nous avons identifié et quantifié l’impact de divers biais, tant techniques que biologiques, sur les prédictions des algorithmes. Nous proposons en outre une méthode d’apprentissage permettant de réduire l’impact de tels biais. Malgré cette méthode, leur influence pourrait néanmoins persister [ 7 ]. Pour cette raison, nous avons réalisé l’étude d’interprétation morphologique des modèles sur un sous-groupe de 251 patientes présentant un cancer du sein au profil moléculaire « luminal » ( i.e. , dont les cellules tumorales expriment le récepteur des œstrogènes), où ces biais sont absents, dans le but d’extraire un signal correspondant à la HRD le plus pur possible. Après un ré-entraînement des modèles sur ce jeu de données, nous avons obtenu une performance AUROC de 83 %. Nous avons ensuite examiné deux des blocs des modèles, l’attention et la décision, pour comprendre leurs prédictions : quelles tuiles les modèles entraînés utilisent-ils le plus, et comment influencent-elles leur décision de classement en HRD ou HRP ?
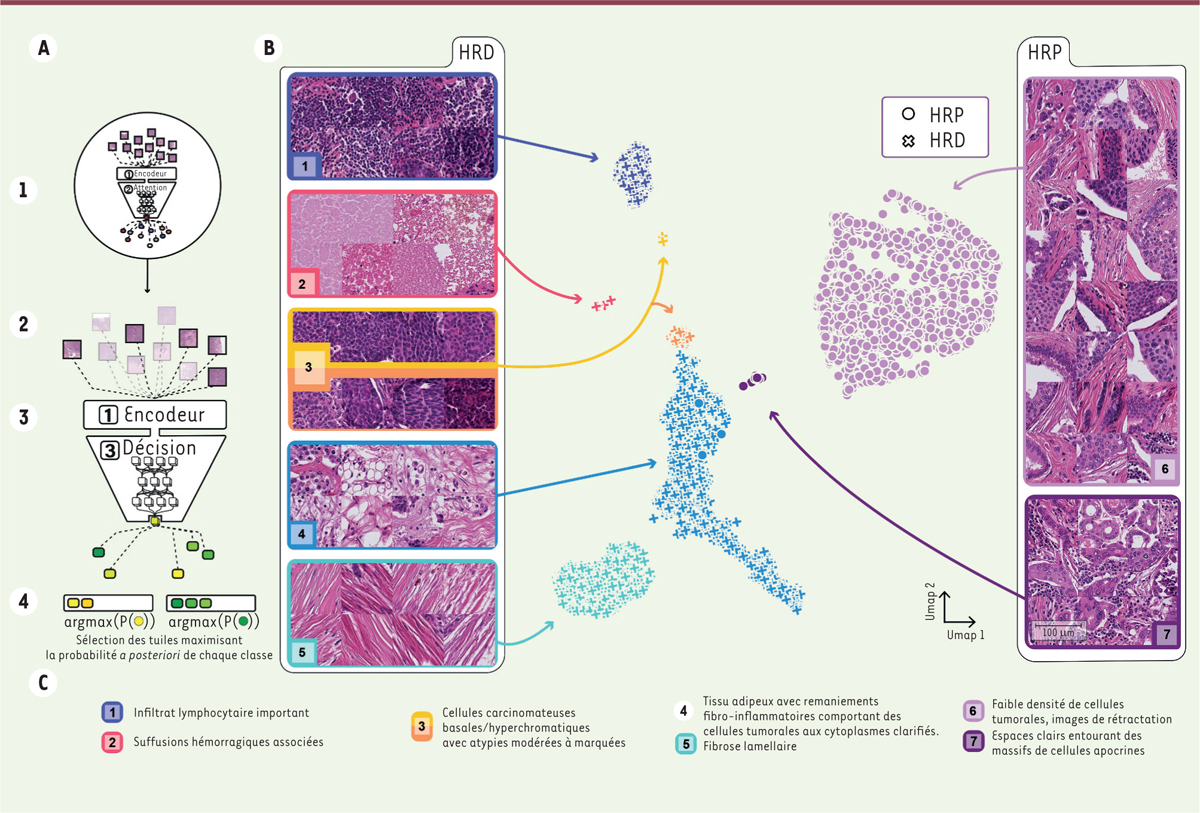
En accord avec la littérature médicale, nous avons constaté que le statut HRD est associé à un important infiltrat inflammatoire du stroma (tissu conjonctif) tumoral, l’existence de plages de nécrose et de suffusion hémorragique, une forte densité cellulaire, et des atypies nucléaires ( Figure 1 ). De plus, une fibrose lamellaire dense du stroma tumoral semble également associée à ce statut, ce qui suggère un rôle important des fibroblastes associés au cancer dans les tumeurs luminales classées HRD. Enfin, il existait une corrélation négative entre le statut HRD et l’existence d’images de rétractation entre les massifs de cellules tumorales et le stroma tumoral. Ces résultats, aussi bien du point de vue de la classification du statut HR des tumeurs que de l’extraction des motifs morphologiques associés, ont pu être validés sur le jeu de données publiques de The cancer genome atlas .
| Figure 1.
A. Résumé de l’algorithme d’interprétation des coupes histologiques de cancer du sein.
Chaque tuile du jeu de données est d’abord filtrée par le module d’attention. Les tuiles filtrées passent par le module de décision, et sont classées selon leur score d’appartenance à une classe (
homologous recombination deficiency
, HRD, ou
proficiency
, HRP). Celles ayant les plus hauts scores sont sélectionnées comme représentatives.
B. Projection UMAP (uniform manifold approximation and projection) des représentations de tuiles sélectionnées par l’algorithme d’interprétation.
UMAP est une méthode de réduction de la dimensionalité. Les groupes sont automatiquement calculés par la méthode de groupement HDBScan. Chaque groupe se voit attribué une couleur et un numéro spécifique. Les tuiles présentes sur la figure sont aléatoirement sélectionnées parmi celles du groupe correspondant.
C. Interprétation des différents groupes (ou motifs) présents en B.
Pour chaque groupe présent en B, représenté par une couleur et un numéro, est mise en correspondance son interprétation morphologique (figure adaptée de [
7
]).
|
Nous avons donc montré que la prédiction du statut HR des cancers du sein, une variable actuellement essentielle pour guider la thérapie, peut être réalisée à partir de la numérisation de simples lames histologiques de la tumeur. La prochaine étape consistera à intégrer cette prédiction dans des essais cliniques afin de la valider et de la rendre utilisable en routine. Enfin, cette étude démontre qu’au delà de leur intérêt médical immédiat, les algorithmes d’apprentissage ouvrent également la voie à de nouvelles hypothèses biologiques concernant la relation entre génotype et phénotype.