| |
| Med Sci (Paris). 39(12): 981–983. doi: 10.1051/medsci/2023180.Les secrets des variants Chroniques génomiques Bertrand Jordan1* 1Biologiste, généticien et immunologiste, Président d’Aprogène (Association pour la promotion de la Génomique)
,
13007Marseille
,
France MeSH keywords: Humains, Protéome, Génétique médicale |
La mise au point du système
AlphaFold
[
1
,
2
] (
→
) en 2021 a permis pour la première fois la prédiction de la structure tridimensionnelle de protéines à partir de leur séquence, avec une fiabilité et un niveau de détail quasiment équivalents à ce qu’apporte la (laborieuse) détermination expérimentale de ces structures. Cela représente un Graal vers lequel tendaient de nombreuses équipes depuis plus de cinquante ans ; il a longtemps paru hors de portée, et c’est la combinaison des techniques d’intelligence artificielle avec l’exploitation de vastes banques de données qui a finalement permis ce succès.
AlphaFold
est l’œuvre d’une trentaine de scientifiques rassemblés au sein de l’entreprise
DeepMind
, émanation de
Google
, spécialisée dans le développement et les applications de l’intelligence artificielle. Tout récemment,
DeepMind
a annoncé une nouvelle avancée avec le système
AlphaMissense
[
3
], présenté comme capable de prédire le caractère pathogène ou bénin de n’importe quelle substitution au sein de l’une des vingt mille séquences codantes que comporte notre génome. C’est une avancée importante qui va avoir des conséquences dans de multiples domaines et notamment en génétique médicale.
(→) Voir la Chronique génomique de B. Jordan,
m/s
n° 2, février 2021, page 197
|
Il y a variant et variant
Chacune des trois milliards de bases de notre ADN est susceptible de subir une mutation. L’immense majorité n’a aucune conséquence fonctionnelle, mais certaines des altérations qui touchent nos gènes (ou leurs séquences de régulation) peuvent modifier le fonctionnement de l’organisme et parfois provoquer des maladies. Si l’on se limite aux séquences codant des protéines, la création d’un codon « stop » induit presque toujours la production d’une protéine non fonctionnelle ; mais le plus souvent, l’altération va être de type « faux sens » (
missense
), c’est-à-dire qu’elle va changer un acide aminé en un autre sans pour autant interrompre la chaîne d’acides aminés. Du coup, la protéine, selon les cas, peut rester fonctionnelle ou, au contraire, être inactivée. Lors du séquençage de l’ADN d’un malade ou d’une tumeur, on découvre souvent un tel variant et se pose alors la question de savoir s’il est pathogène (auquel cas on a peut-être découvert la cause de l’affection) ou bénin. Au mieux, s’il a déjà été étudié, une base de données cliniques comme
ClinVa
1,
va donner une réponse ; il peut s’agir d’un variant déjà répertorié mais de signification inconnue (VUS, pour
variant of unknown significance
), ou d’un variant jusque-là inconnu. Pour fixer les idées, sur les près de vingt mille séquences codantes que renferme notre génome, il y a 71 millions de substitutions
missense
possibles dont quatre millions ont été observées et environ cent mille seulement classées comme pathogènes ou bénignes – tout le reste appartient à la catégorie des VUS [
3
]. Il serait donc fabuleux de disposer d’un outil permettant d’indiquer si une substitution donnée rend, ou pas, la protéine inactive, et,
a priori
,
AlphaFold
semble offrir la possibilité d’une solution.
|
La voie paraît toute tracée :
AlphaFold
prédit la structure de la protéine « standard », on change un acide aminé selon la mutation observée et on exécute de nouveau
AlphaFold
, ce qui donne une nouvelle structure dont on va examiner les différences pour évaluer si le changement risque d’être impactant. Sauf que…
AlphaFold
n’est pas capable de prédire les changements induits par la substitution d’un acide aminé [
4
] ! C’est que ce système est fondé sur l’analyse de similitudes entre protéines et sur les structures connues, en exploitant de grandes bases de données par intelligence artificielle, et non sur un véritable calcul des structures tridimensionnelles à partir de principes premiers [
1
]. Et les bases de données qu’exploite le système n’incluent pas des structures de protéines ayant subi une substitution.… Le système
AlphaMissense
pourrait s’appuyer sur les données cliniques déjà répertoriées (type
ClinVar
) – c’est ce que font plusieurs des systèmes de prédiction déjà utilisés –, mais ce serait introduire un biais important et en quelque sorte un raisonnement circulaire.
DeepMind
a donc pris une approche différente, fondée sur l’analyse de la conservation des séquences et l’utilisation des contextes de structure apportés par
AlphaFold
, sans utilisation des données cliniques existantes. L’évaluation du caractère pathogène des variations repose en particulier sur la prise en compte des fréquences alléliques observées dans la population, en partant du principe que les variants très rares ou non observés ont toutes les chances d’être pathogènes (tendant donc à être éliminés par la sélection), et en examinant si les substitutions évaluées sont compatibles avec les règles de structure indiquées par
AlphaFold
. Le résultat est un « indice de pathogénicité » compris entre 0 (variant bénin) et 1 (variant certainement pathogène). On peut alors tester le système sur un jeu de variants répertoriés dans
ClinVar
pour avoir une idée de sa validité.
|
L’examen des performances d’
AlphaMissense
peut se faire de différentes manières, nous en verrons deux ici. On peut partir d’un jeu équilibré de variants
ClinVar
comprenant 9 462 variants pathogènes et autant de variants bénins, définis à partir de données cliniques.
AlphaMissense
va alors calculer un score de pathogénicité pour chacun d’eux ; la
Figure 1
montre la répartition des variants pathogènes (en rouge) et bénins (en bleu) par rapport à ce score. On voit que les variants cliniquement connus comme bénins ont presque tous un score de pathogénicité inférieur à 0,2, et ceux qui sont pathogènes, un score supérieur à 0,8. La classification donnée par
AlphaMissense
retrouve donc bien les données cliniques pour un jeu important de variants cliniquement caractérisés.
 | Figure 1.
Analyse par AlphaMissense de 18 924 variants bénins (en bleu) ou pathogènes (en rouge) selon les données de la base ClinVar.
On voit que le score selon
AlphaMissense
correspond bien aux données cliniques. La courbe en diagonale indique la proportion de variants pathogènes en fonction du score
AlphaMissense
(extrait partiel et remanié de la figure 2D de [
3
]).
|
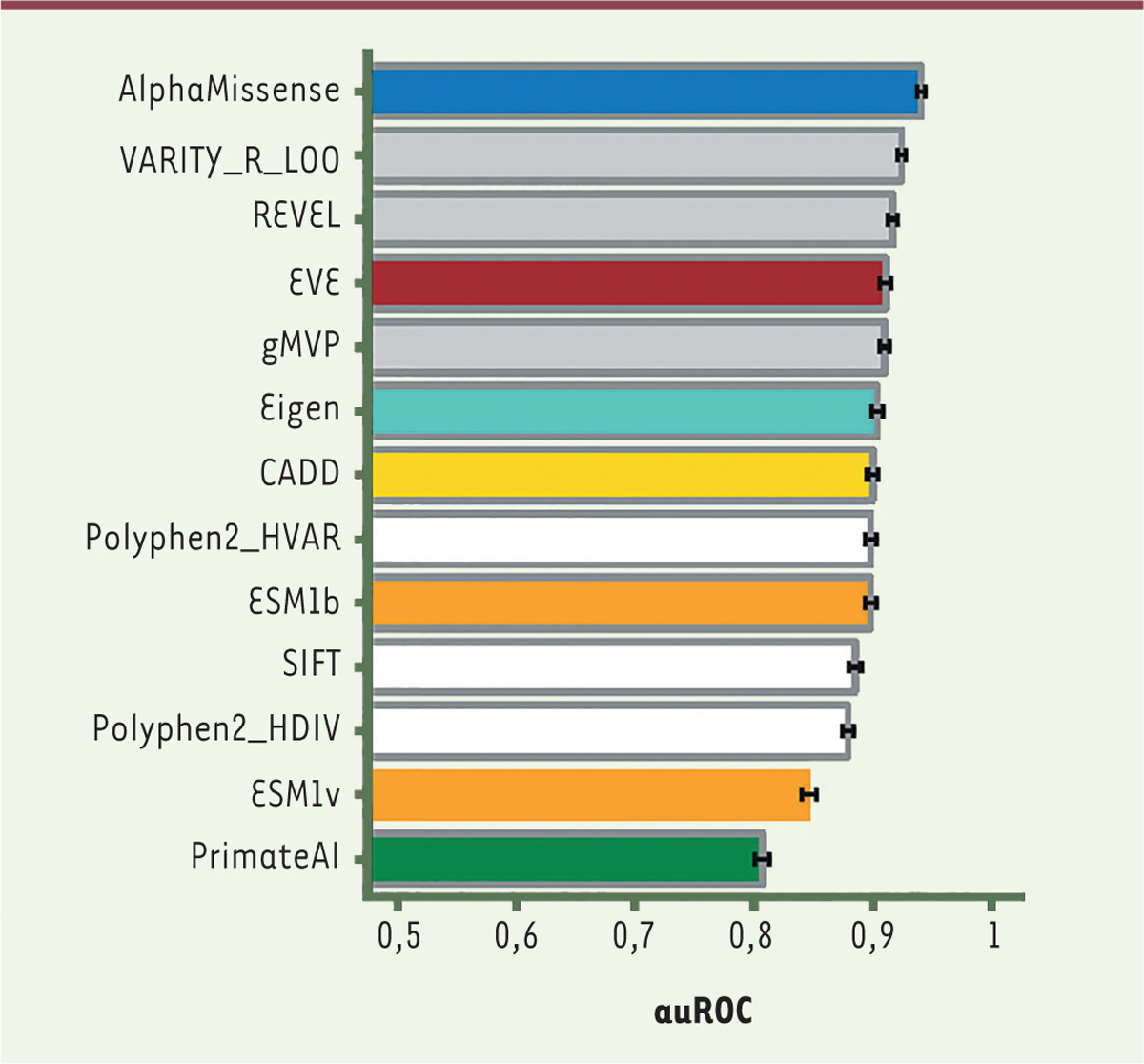
Une autre information importante concerne la comparaison entre
AlphaMissense
et d’autres algorithmes de prédiction. Il existe en effet de nombreux systèmes visant à prédire la pathogénicité d’une substitution (voir [
3
]) ; certains utilisent des données cliniques pour « entraîner » le système, d’autres tentent de s’en passer. On peut évaluer la qualité globale des prévisions fournies en analysant leur cohérence avec les données cliniques grâce à la méthode du ROC (
receiver operating curve
) [
5
] (
→
) qui tient compte simultanément de la sensibilité et de la spécificité et fournit un score (auROC, pour
area under the receiver operating curve
) d’autant plus proche de 1 que la mesure est performante
2
. Comme le montre la
Figure 2
, après analyse des 18 924 variants déjà mentionnés,
AlphaMissense
s’en sort avec les honneurs puisqu’il se classe premier – et que la plupart des systèmes qui approchent de ses performances ont été entraînés… avec les données de
ClinVar
, ce qui rend leur performance nettement moins significative puisqu’il y a recouvrement entre les données d’entraînement et les données de test.
(→) Voir la Chronique génomique de B. Jordan,
m/s
n° 3, mars 2012, page 325
 | Figure 2.
Performances de différents systèmes de classification, testés sur le jeu équilibré de 18 924 variants
ClinVar.
La qualité du résultat est donnée par le paramètre auROC (
area under the receiver operating curve
) d’autant plus proche de 1 que la mesure est performante. Les 12 algorithmes comparés à
AlphaMissense
sont répertoriés dans [
3
]. Les barres grises indiquent les systèmes ayant utilisé les données de
ClinVar
pour leur apprentissage (extrait partiel et modifié de la
Figure 2B
de [
3
]).
|
Les performances
d’AlphaMissense
font que ce système est réellement opérationnel pour évaluer la pathogénicité d’un variant. Il va être d’autant plus utile que ses concepteurs l’ont utilisé pour classifier les 71 millions de variants
missense
possibles pour l’ensemble du protéome humain, et mettent ces résultats à la disposition de la communauté scientifique
via
une base de données librement accessible [
6
]. Globalement, les 71 millions de variants s’avèrent, selon
AlphaMissense
, bénins pour 57 %, probablement pathogènes pour 32 %, et ambigus pour 11 %. Notons bien qu’il s’agit ici de l’ensemble des variants possibles, et non de ceux qui ont été observés : rappelons que sur les quatre millions de variants inclus dans
ClinVar
, seuls cent mille environ sont répertoriés comme pathogènes ou bénins, tout le reste étant de signification inconnue (VUS). Cela reflète l’étendue de notre ignorance sur les VUS, mais aussi le fait que beaucoup de variants pathogènes ne seront jamais observés car ils ont été éliminés par la sélection naturelle.
|
Les bonnes performances d’
AlphaMissense
, et la mise à disposition de l’ensemble des résultats sur le protéome humain [
6
], vont avoir d’importantes conséquences en génétique médicale et, notamment, pour l’élucidation du mécanisme génétique de maladies rares. La recherche dans ce domaine passe de plus en plus par le séquençage intégral du génome du patient, et par l’analyse des mutations qu’il présente. Celles-ci sont souvent nombreuses, plusieurs dizaines compte tenu du taux de mutations germinales observé [
7
] (
→
) ; leur interprétation en termes d’effet sur la fonction est donc essentielle. Un système d’analyse performant va permettre de gagner beaucoup de temps et de raccourcir l’« odyssée diagnostique »
3
qui, aujourd’hui, dure souvent plusieurs années. On peut ainsi s’attendre à découvrir de nombreux gènes impliqués dans des maladies rares et, plus généralement, à mieux comprendre les relations entre la structure d’une protéine et sa fonction. Certes,
AlphaMissense
n’est pas parfait, et le score de pathogénicité qu’il fournit est moins solide qu’un ensemble de données cliniques, mais la bonne corrélation montrée par la
Figure 1
indique que ce système va jouer un rôle important dans l’interprétation fonctionnelle de notre génome et va avoir un impact notable en génétique médicale.
(→) Voir la Chronique génomique de B. Jordan,
m/s
n° 8-9, août-septembre 2023, page 665
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jumper
J
,
Evans
R
,
Pritzel
A
,
et al
.
Highly accurate protein structure prediction with AlphaFold.
.
Nature
.
2021;
;
596
:
:583.
–
9
.
2.
Jordan
B
.
AlphaFold : un pas essentiel vers la fonction des protéines.
.
Med Sci (Paris)
.
2021;
;
37
:
:197.
–
200
.
3.
Cheng
J
,
Novati
G
,
Pan
J
,
et al
.
Accurate proteome-wide missense variant effect prediction with AlphaMissense.
.
Science
.
2023;
;
381
:
:eadg7492.
.
4.
Buel
GR
,
Walters
KJ
.
Can AlphaFold2 predict the impact of missense mutations on structure?
Nat Struct Mol Biol
.
2022;
;
29
:
:1.
–
2
.
5.
Jordan
B.
Les tests génétiques grand public ont-ils une utilité clinique ?
.
Med Sci (Paris)
.
2012;
;
28
:
:325.
–
328
.
7.
Jordan
B
.
Tout savoir sur les mutations germinales chez les vertébrés.
.
Med Sci (Paris)
.
2023;
;
39
:
:665.
–
7
.
|