Méthodes et outils de caractérisation de l’impact de xénobiotiques sur la reproduction
2011
| ANALYSE |
20-
Approches « omiques »
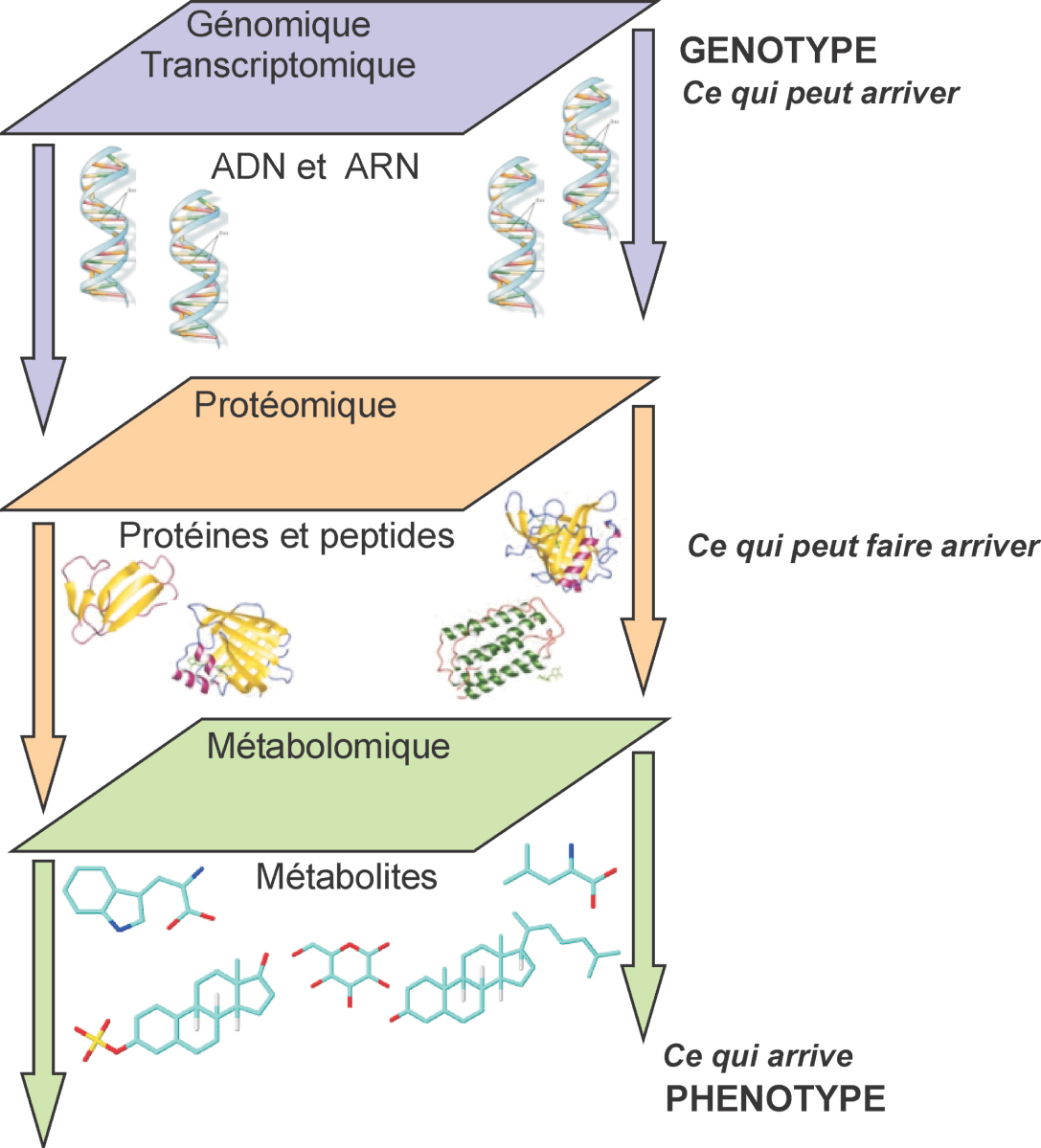
L’idée de base associée aux approches « omiques » consiste à appréhender la complexité du vivant dans son ensemble, au moyen de méthodologies les moins restrictives possibles sur le plan descriptif. Ces approches peuvent en particulier être utiles pour mettre en évidence et identifier de nouveaux biomarqueurs (d’exposition, d’effet ou de susceptibilité), générer de nouvelles connaissances sur le plan mécanistique (modes d’action), ou encore élaborer de nouveaux outils de toxicologie prédictive pour aider à l’identification des dangers. Caractérisées comme des techniques à haut débit permettant une analyse simultanée d’un grand nombre de variables, elles comprennent principalement (figure 20.1 ) : la génomique (SNPs par exemple), la transcriptomique (expression des gènes et leur régulation), la protéomique (analyse des protéines), la métabolomique (étude des métabolites produits). Ces approches permettent d’obtenir de très nombreuses informations sur la réponse cellulaire et/ou tissulaire à une exposition in vitro ou in vivo.
) : la génomique (SNPs par exemple), la transcriptomique (expression des gènes et leur régulation), la protéomique (analyse des protéines), la métabolomique (étude des métabolites produits). Ces approches permettent d’obtenir de très nombreuses informations sur la réponse cellulaire et/ou tissulaire à une exposition in vitro ou in vivo.
 ) : la génomique (SNPs par exemple), la transcriptomique (expression des gènes et leur régulation), la protéomique (analyse des protéines), la métabolomique (étude des métabolites produits). Ces approches permettent d’obtenir de très nombreuses informations sur la réponse cellulaire et/ou tissulaire à une exposition in vitro ou in vivo.
) : la génomique (SNPs par exemple), la transcriptomique (expression des gènes et leur régulation), la protéomique (analyse des protéines), la métabolomique (étude des métabolites produits). Ces approches permettent d’obtenir de très nombreuses informations sur la réponse cellulaire et/ou tissulaire à une exposition in vitro ou in vivo.Le développement des techniques à haut débit permettant ce type de profilage ou de « prise d’empreinte », ainsi que l’existence de techniques statistiques historiquement bien établies et maîtrisées laissant supposer une relative facilité de retraitement des données générées, font qu’aujourd’hui le nombre de chercheurs engagés dans cette voie est très important.
Si le passage de l’ère du tout génomique à l’ère de la protéomique a pris quelques dizaines d’années, le passage de la protéomique à la métabolomique n’a en effet pris que quelques années, puis l’apparition du concept de fluxomique1
n’a ensuite pris que pratiquement quelques mois. Aujourd’hui, se développe un concept de « Big Science », reposant sur la génération et l’analyse d’un nombre de données jamais atteint auparavant, un des objectifs étant d’exploiter simultanément les données issues de ces différentes approches, comme autant d’angles de prise de vue complémentaires décrivant les systèmes biologiques étudiés. La difficulté réside néanmoins dans l’interprétation de ces nombreuses données nécessitant de faire appel à des moyens bio-informatiques puissants. La puissance de ces outils sur le plan descriptif permet aujourd’hui une caractérisation des systèmes biologiques à une échelle et une profondeur jamais atteintes. Ces technologies se heurtent toutefois à certaines limites sur le plan explicatif, la relation entre une signature moléculaire et la toxicité n’étant pas toujours évidente, de même que la validité aussi bien que l’interprétation biologique de biomarqueurs mis en évidence par ces nouveaux outils.
 | Figure 20.1 Représentation schématique des trois grands niveaux de caractérisation des systèmes biologiques |
Transcriptomique et protéomique
Depuis 2-3 ans, des approches globales d’analyse transcriptomique ont été réalisées à partir de différents organes (ovaires, testicules, utérus, glande mammaire, placenta) suite à des expositions in utero chez les rongeurs ou de cultures cellulaires chez l’homme. Ces études, qui concernent le bisphénol A (Moral et coll., 2008 ; Lawson et coll., 2011
; Lawson et coll., 2011 ) et les phtalates (Yuan et coll., 2010
) et les phtalates (Yuan et coll., 2010 ) mettent en évidence des modifications au niveau des grandes fonctions cellulaires, telles que la différenciation, le cycle cellulaire, ou les processus de biosynthèse des hormones stéroïdes. Il est difficile de dire si ces modifications sont des causes ou des conséquences des phénotypes observés et de faire des relations directes entre exposition et ces altérations transcriptionnelles mesurées souvent longtemps après l’exposition. Cependant, ces études sont des points de départ pour définir de nouveaux biomarqueurs d’exposition et avoir une vision globale de l’ensemble des mécanismes altérés au sein d’un tissu.
) mettent en évidence des modifications au niveau des grandes fonctions cellulaires, telles que la différenciation, le cycle cellulaire, ou les processus de biosynthèse des hormones stéroïdes. Il est difficile de dire si ces modifications sont des causes ou des conséquences des phénotypes observés et de faire des relations directes entre exposition et ces altérations transcriptionnelles mesurées souvent longtemps après l’exposition. Cependant, ces études sont des points de départ pour définir de nouveaux biomarqueurs d’exposition et avoir une vision globale de l’ensemble des mécanismes altérés au sein d’un tissu.
 ; Lawson et coll., 2011
; Lawson et coll., 2011 ) et les phtalates (Yuan et coll., 2010
) et les phtalates (Yuan et coll., 2010 ) mettent en évidence des modifications au niveau des grandes fonctions cellulaires, telles que la différenciation, le cycle cellulaire, ou les processus de biosynthèse des hormones stéroïdes. Il est difficile de dire si ces modifications sont des causes ou des conséquences des phénotypes observés et de faire des relations directes entre exposition et ces altérations transcriptionnelles mesurées souvent longtemps après l’exposition. Cependant, ces études sont des points de départ pour définir de nouveaux biomarqueurs d’exposition et avoir une vision globale de l’ensemble des mécanismes altérés au sein d’un tissu.
) mettent en évidence des modifications au niveau des grandes fonctions cellulaires, telles que la différenciation, le cycle cellulaire, ou les processus de biosynthèse des hormones stéroïdes. Il est difficile de dire si ces modifications sont des causes ou des conséquences des phénotypes observés et de faire des relations directes entre exposition et ces altérations transcriptionnelles mesurées souvent longtemps après l’exposition. Cependant, ces études sont des points de départ pour définir de nouveaux biomarqueurs d’exposition et avoir une vision globale de l’ensemble des mécanismes altérés au sein d’un tissu. Des approches protéomiques ont également été récemment exploitées afin de caractériser une signature biologique caractéristique d’une exposition au bisphénol A chez le rongeur soit au niveau de la glande mammaire (Betancourt et coll., 2010 ), soit au niveau des organes impliqués dans l’immunité (Yang et coll., 2008
), soit au niveau des organes impliqués dans l’immunité (Yang et coll., 2008 ). Ces études ont ainsi mis en évidence et identifié plusieurs protéines dont l’expression semble significativement affectée par le BPA, dont certaines impliquées dans la signalisation cellulaire. Deux études protéomiques ont également été conduites chez le poisson exposé à des contaminants perfluorés, au PFOA (Wei et coll., 2008
). Ces études ont ainsi mis en évidence et identifié plusieurs protéines dont l’expression semble significativement affectée par le BPA, dont certaines impliquées dans la signalisation cellulaire. Deux études protéomiques ont également été conduites chez le poisson exposé à des contaminants perfluorés, au PFOA (Wei et coll., 2008 ) ou au PFOS (Shi et coll., 2009
) ou au PFOS (Shi et coll., 2009 ). Ces études ont montré l’existence d’une vingtaine de protéines (au niveau hépatique chez le vairon pour la première étude ou bien au niveau embryonnaire chez le zebrafish pour la seconde) dont l’abondance est significativement affectée par ces composés, ces protéines étant impliquées dans les voies physiologiques variées incluant le transport d’acides gras, le stress oxydatif, le maintien de l’homéostasie, le cycle cellulaire, ou encore les fonctions mitochondriales. Kling et coll. (2009
). Ces études ont montré l’existence d’une vingtaine de protéines (au niveau hépatique chez le vairon pour la première étude ou bien au niveau embryonnaire chez le zebrafish pour la seconde) dont l’abondance est significativement affectée par ces composés, ces protéines étant impliquées dans les voies physiologiques variées incluant le transport d’acides gras, le stress oxydatif, le maintien de l’homéostasie, le cycle cellulaire, ou encore les fonctions mitochondriales. Kling et coll. (2009 ) ont, quant à eux, réalisé une étude protéomique également chez le poisson (zebrafish) mais à partir de cultures cellulaires hépatiques exposées à des retardateurs de flamme polybromés (HBCD et/ou TBBPA), cette étude ayant là encore montré l’existence d’un profil protéique caractéristique de cette exposition, avec une partie distincte et une partie commune entre les deux substances testées. Cette étude a de plus souligné l’intérêt de cette approche pour mettre en évidence des effets de mélanges pas toujours prévisibles.
) ont, quant à eux, réalisé une étude protéomique également chez le poisson (zebrafish) mais à partir de cultures cellulaires hépatiques exposées à des retardateurs de flamme polybromés (HBCD et/ou TBBPA), cette étude ayant là encore montré l’existence d’un profil protéique caractéristique de cette exposition, avec une partie distincte et une partie commune entre les deux substances testées. Cette étude a de plus souligné l’intérêt de cette approche pour mettre en évidence des effets de mélanges pas toujours prévisibles.
 ), soit au niveau des organes impliqués dans l’immunité (Yang et coll., 2008
), soit au niveau des organes impliqués dans l’immunité (Yang et coll., 2008 ). Ces études ont ainsi mis en évidence et identifié plusieurs protéines dont l’expression semble significativement affectée par le BPA, dont certaines impliquées dans la signalisation cellulaire. Deux études protéomiques ont également été conduites chez le poisson exposé à des contaminants perfluorés, au PFOA (Wei et coll., 2008
). Ces études ont ainsi mis en évidence et identifié plusieurs protéines dont l’expression semble significativement affectée par le BPA, dont certaines impliquées dans la signalisation cellulaire. Deux études protéomiques ont également été conduites chez le poisson exposé à des contaminants perfluorés, au PFOA (Wei et coll., 2008 ) ou au PFOS (Shi et coll., 2009
) ou au PFOS (Shi et coll., 2009 ). Ces études ont montré l’existence d’une vingtaine de protéines (au niveau hépatique chez le vairon pour la première étude ou bien au niveau embryonnaire chez le zebrafish pour la seconde) dont l’abondance est significativement affectée par ces composés, ces protéines étant impliquées dans les voies physiologiques variées incluant le transport d’acides gras, le stress oxydatif, le maintien de l’homéostasie, le cycle cellulaire, ou encore les fonctions mitochondriales. Kling et coll. (2009
). Ces études ont montré l’existence d’une vingtaine de protéines (au niveau hépatique chez le vairon pour la première étude ou bien au niveau embryonnaire chez le zebrafish pour la seconde) dont l’abondance est significativement affectée par ces composés, ces protéines étant impliquées dans les voies physiologiques variées incluant le transport d’acides gras, le stress oxydatif, le maintien de l’homéostasie, le cycle cellulaire, ou encore les fonctions mitochondriales. Kling et coll. (2009 ) ont, quant à eux, réalisé une étude protéomique également chez le poisson (zebrafish) mais à partir de cultures cellulaires hépatiques exposées à des retardateurs de flamme polybromés (HBCD et/ou TBBPA), cette étude ayant là encore montré l’existence d’un profil protéique caractéristique de cette exposition, avec une partie distincte et une partie commune entre les deux substances testées. Cette étude a de plus souligné l’intérêt de cette approche pour mettre en évidence des effets de mélanges pas toujours prévisibles.
) ont, quant à eux, réalisé une étude protéomique également chez le poisson (zebrafish) mais à partir de cultures cellulaires hépatiques exposées à des retardateurs de flamme polybromés (HBCD et/ou TBBPA), cette étude ayant là encore montré l’existence d’un profil protéique caractéristique de cette exposition, avec une partie distincte et une partie commune entre les deux substances testées. Cette étude a de plus souligné l’intérêt de cette approche pour mettre en évidence des effets de mélanges pas toujours prévisibles.Métabolomique
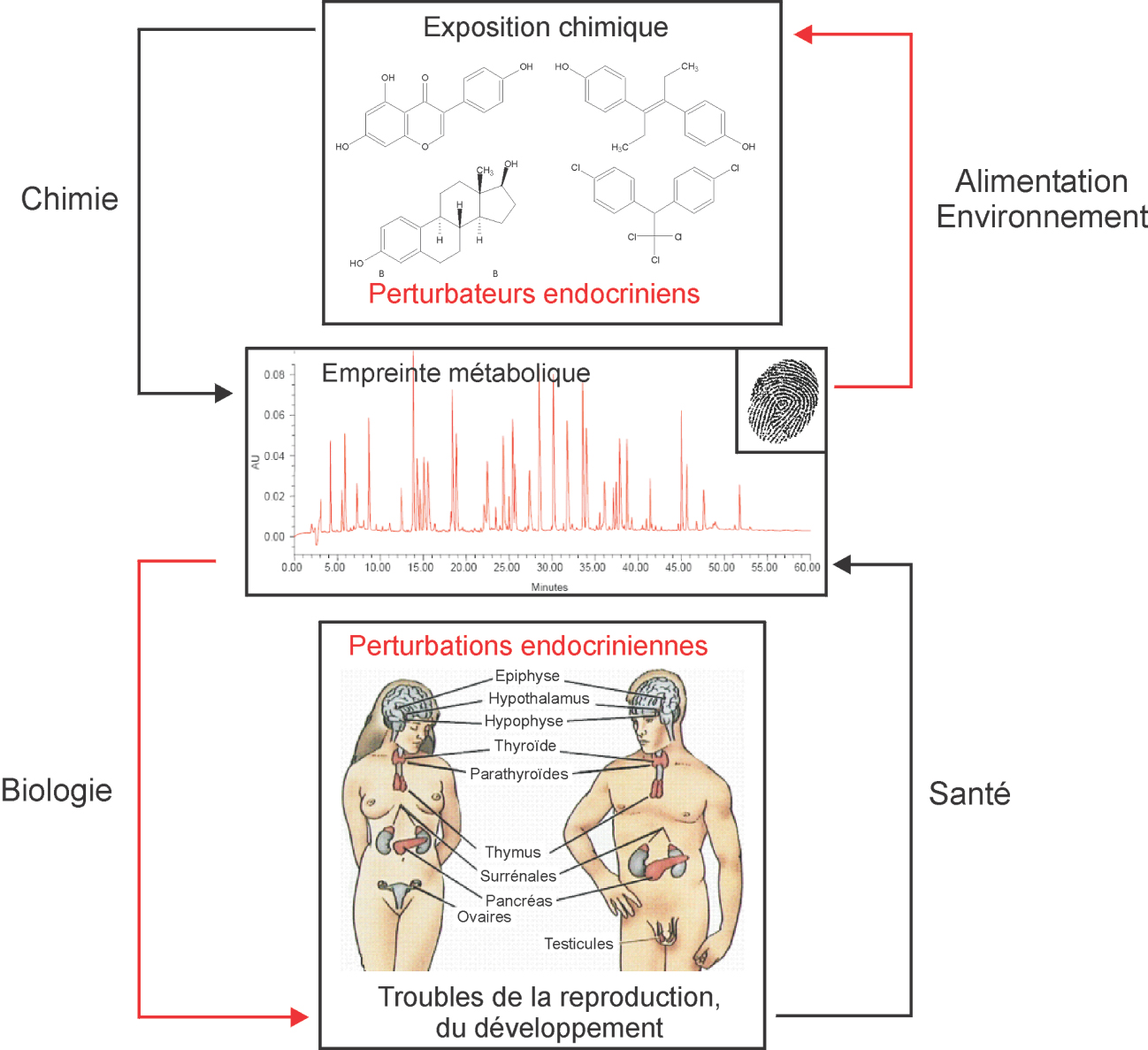
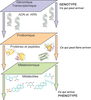
Les approches de type métabolomique s’intéressent plus particulièrement aux composés chimiques présents au sein des systèmes biologiques étudiés, après la mise en jeu des phénomènes complexes de transcription et de traduction. Il s’agit donc globalement de molécules de faible poids moléculaire, le plus souvent inférieur à 1 000 Da, qui peuvent correspondre à des substances endogènes (métabolites primaires ou secondaires) ou exogènes. De façon conceptuelle (figure 20.2 ) et dans le domaine particulier de la toxicologie, le type de signature biologique au niveau du métabolome peut donc être considérée comme reflétant de façon concomitante une exposition chimique (mesure des substances chimiques parentes ou de leurs produits directs de biotransformation/dégradation) et l’effet biologique induit par cette exposition (mesure de biomarqueurs indirects dont le niveau d’expression est sur- ou sous-exprimé consécutivement à cette exposition). Un des principaux avantages de ces approches est donc de caractériser de façon assez globale et non ciblée les perturbations biologiques liées à une exposition chimique donnée.
) et dans le domaine particulier de la toxicologie, le type de signature biologique au niveau du métabolome peut donc être considérée comme reflétant de façon concomitante une exposition chimique (mesure des substances chimiques parentes ou de leurs produits directs de biotransformation/dégradation) et l’effet biologique induit par cette exposition (mesure de biomarqueurs indirects dont le niveau d’expression est sur- ou sous-exprimé consécutivement à cette exposition). Un des principaux avantages de ces approches est donc de caractériser de façon assez globale et non ciblée les perturbations biologiques liées à une exposition chimique donnée.
 ) et dans le domaine particulier de la toxicologie, le type de signature biologique au niveau du métabolome peut donc être considérée comme reflétant de façon concomitante une exposition chimique (mesure des substances chimiques parentes ou de leurs produits directs de biotransformation/dégradation) et l’effet biologique induit par cette exposition (mesure de biomarqueurs indirects dont le niveau d’expression est sur- ou sous-exprimé consécutivement à cette exposition). Un des principaux avantages de ces approches est donc de caractériser de façon assez globale et non ciblée les perturbations biologiques liées à une exposition chimique donnée.
) et dans le domaine particulier de la toxicologie, le type de signature biologique au niveau du métabolome peut donc être considérée comme reflétant de façon concomitante une exposition chimique (mesure des substances chimiques parentes ou de leurs produits directs de biotransformation/dégradation) et l’effet biologique induit par cette exposition (mesure de biomarqueurs indirects dont le niveau d’expression est sur- ou sous-exprimé consécutivement à cette exposition). Un des principaux avantages de ces approches est donc de caractériser de façon assez globale et non ciblée les perturbations biologiques liées à une exposition chimique donnée. | Figure 20.2 Concept d’empreinte métabolique comme lien entre perturbateurs endocriniens et perturbation endocrinienne |
Sur le plan technique, ce type de profilage métabolique a été historiquement proposé via la résonance magnétique nucléaire (RMN), notamment par l’équipe de Nicholson au Royaume-Uni (1999 ). Cette technologie est toujours le support de nombreuses applications en métabolomique, y compris dans le champ de la toxicologie et de la santé. La spectrométrie de masse (MS) représente une seconde approche de choix dans ce domaine.
). Cette technologie est toujours le support de nombreuses applications en métabolomique, y compris dans le champ de la toxicologie et de la santé. La spectrométrie de masse (MS) représente une seconde approche de choix dans ce domaine.
 ). Cette technologie est toujours le support de nombreuses applications en métabolomique, y compris dans le champ de la toxicologie et de la santé. La spectrométrie de masse (MS) représente une seconde approche de choix dans ce domaine.
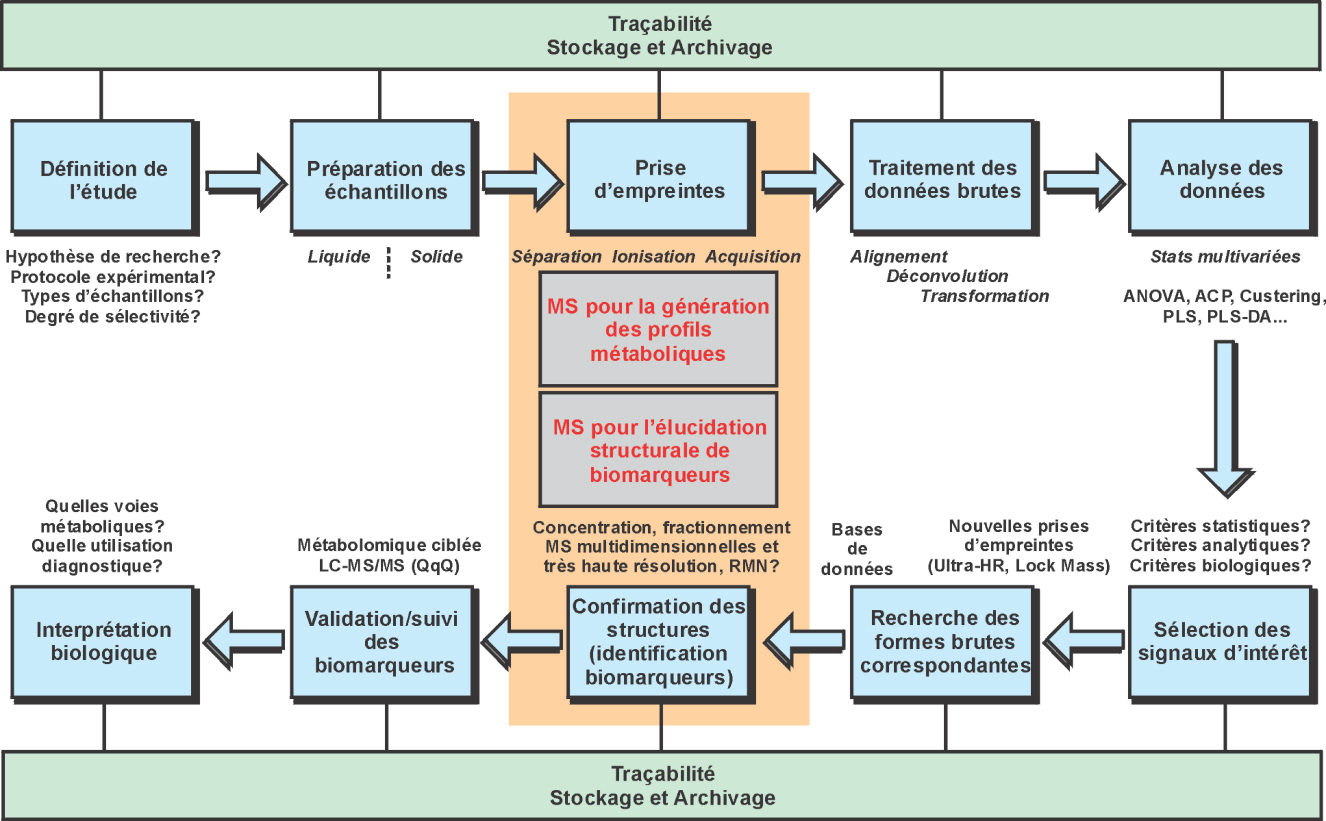
). Cette technologie est toujours le support de nombreuses applications en métabolomique, y compris dans le champ de la toxicologie et de la santé. La spectrométrie de masse (MS) représente une seconde approche de choix dans ce domaine.Au-delà de ce concept relativement simple d’empreintes ou profils métaboliques, ce domaine requiert de façon pratique la mise en œuvre d’un cadre méthodologique dépassant largement l’étape de génération de ces profils proprement dite (Antignac et coll., 2011 ). Ainsi (figure 20.3
). Ainsi (figure 20.3 ), des procédures doivent être développées et implémentées au niveau du traitement des échantillons biologique avant analyse, du retraitement des données brutes générées, de l’analyse statistique (notamment par des méthodes multivariées avancées), de l’extraction de l’information utile contenue dans ces empreintes sur la base d’un ensemble de critères complémentaires, de l’élucidation structurale des composés chimiques pouvant répondre au qualificatif de candidat biomarqueur, de la validation de ce caractère de biomarqueur, puis de l’interprétation biologique des perturbations métaboliques ainsi caractérisées.
), des procédures doivent être développées et implémentées au niveau du traitement des échantillons biologique avant analyse, du retraitement des données brutes générées, de l’analyse statistique (notamment par des méthodes multivariées avancées), de l’extraction de l’information utile contenue dans ces empreintes sur la base d’un ensemble de critères complémentaires, de l’élucidation structurale des composés chimiques pouvant répondre au qualificatif de candidat biomarqueur, de la validation de ce caractère de biomarqueur, puis de l’interprétation biologique des perturbations métaboliques ainsi caractérisées.
 ). Ainsi (figure 20.3
). Ainsi (figure 20.3 ), des procédures doivent être développées et implémentées au niveau du traitement des échantillons biologique avant analyse, du retraitement des données brutes générées, de l’analyse statistique (notamment par des méthodes multivariées avancées), de l’extraction de l’information utile contenue dans ces empreintes sur la base d’un ensemble de critères complémentaires, de l’élucidation structurale des composés chimiques pouvant répondre au qualificatif de candidat biomarqueur, de la validation de ce caractère de biomarqueur, puis de l’interprétation biologique des perturbations métaboliques ainsi caractérisées.
), des procédures doivent être développées et implémentées au niveau du traitement des échantillons biologique avant analyse, du retraitement des données brutes générées, de l’analyse statistique (notamment par des méthodes multivariées avancées), de l’extraction de l’information utile contenue dans ces empreintes sur la base d’un ensemble de critères complémentaires, de l’élucidation structurale des composés chimiques pouvant répondre au qualificatif de candidat biomarqueur, de la validation de ce caractère de biomarqueur, puis de l’interprétation biologique des perturbations métaboliques ainsi caractérisées.Si ce schéma général (figure 20.3 ) représente une base globalement commune à l’ensemble des études métabolomiques, celles-ci peuvent avoir des finalités et par conséquent des spécificités dans leur mise en œuvre. Une première finalité de ces études peut être en effet la caractérisation exhaustive de l’ensemble des « métabolites » détectés (métabolomique systématique). Toutefois, ce travail se heurte généralement à la difficulté d’identification structurale non ambiguë des substances chimiques. Une seconde finalité plus couramment visée consiste à mettre en évidence parmi les descripteurs générés, un certain nombre d’entre eux dont le niveau d’expression est significativement différent (sur- ou sous-exprimé) entre deux ou plus sous-groupes d’échantillons donnés (métabolomique différentielle).
) représente une base globalement commune à l’ensemble des études métabolomiques, celles-ci peuvent avoir des finalités et par conséquent des spécificités dans leur mise en œuvre. Une première finalité de ces études peut être en effet la caractérisation exhaustive de l’ensemble des « métabolites » détectés (métabolomique systématique). Toutefois, ce travail se heurte généralement à la difficulté d’identification structurale non ambiguë des substances chimiques. Une seconde finalité plus couramment visée consiste à mettre en évidence parmi les descripteurs générés, un certain nombre d’entre eux dont le niveau d’expression est significativement différent (sur- ou sous-exprimé) entre deux ou plus sous-groupes d’échantillons donnés (métabolomique différentielle).
 ) représente une base globalement commune à l’ensemble des études métabolomiques, celles-ci peuvent avoir des finalités et par conséquent des spécificités dans leur mise en œuvre. Une première finalité de ces études peut être en effet la caractérisation exhaustive de l’ensemble des « métabolites » détectés (métabolomique systématique). Toutefois, ce travail se heurte généralement à la difficulté d’identification structurale non ambiguë des substances chimiques. Une seconde finalité plus couramment visée consiste à mettre en évidence parmi les descripteurs générés, un certain nombre d’entre eux dont le niveau d’expression est significativement différent (sur- ou sous-exprimé) entre deux ou plus sous-groupes d’échantillons donnés (métabolomique différentielle).
) représente une base globalement commune à l’ensemble des études métabolomiques, celles-ci peuvent avoir des finalités et par conséquent des spécificités dans leur mise en œuvre. Une première finalité de ces études peut être en effet la caractérisation exhaustive de l’ensemble des « métabolites » détectés (métabolomique systématique). Toutefois, ce travail se heurte généralement à la difficulté d’identification structurale non ambiguë des substances chimiques. Une seconde finalité plus couramment visée consiste à mettre en évidence parmi les descripteurs générés, un certain nombre d’entre eux dont le niveau d’expression est significativement différent (sur- ou sous-exprimé) entre deux ou plus sous-groupes d’échantillons donnés (métabolomique différentielle).Le concept de métabolomique trouve depuis quelques années un écho particulier dans le domaine de la toxicologie (Lindon et coll., 2005 ). Une première perspective est en effet la mise en évidence globale, bien au-delà du faible nombre de « end-points » usuellement étudiés, de l’impact de certaines substances sur des systèmes biologiques donnés (Robertson et coll., 2000
). Une première perspective est en effet la mise en évidence globale, bien au-delà du faible nombre de « end-points » usuellement étudiés, de l’impact de certaines substances sur des systèmes biologiques donnés (Robertson et coll., 2000 ; Shockcor et coll., 2002
; Shockcor et coll., 2002 ; Griffin et coll., 2004
; Griffin et coll., 2004 ; Ebbels et coll., 2007
; Ebbels et coll., 2007 ). Une seconde perspective est l’identification de biomarqueurs d’exposition et/ou d’effet permettant la génération de nouvelles connaissances s’agissant de leurs modes d’actions (Clarke et coll., 2008
). Une seconde perspective est l’identification de biomarqueurs d’exposition et/ou d’effet permettant la génération de nouvelles connaissances s’agissant de leurs modes d’actions (Clarke et coll., 2008 ; Coen et coll., 2008
; Coen et coll., 2008 ; Coen, 2010
; Coen, 2010 ; Beger et coll., 2010
; Beger et coll., 2010 ). Dans ce domaine, il est toutefois souvent difficile de distinguer les modifications métaboliques résultant d’une adaptation physiologique de celles signant une toxicité effective. Par ailleurs, la question de la représentativité de l’empreinte métabolomique générée (dépendante des différents niveaux de variabilité biologique et analytique) ou encore de son exhaustivité (dépendante de la technique de mesure), se pose clairement dans le cadre de ces approches et limite parfois la significativité des résultats obtenus et leur interprétation.
). Dans ce domaine, il est toutefois souvent difficile de distinguer les modifications métaboliques résultant d’une adaptation physiologique de celles signant une toxicité effective. Par ailleurs, la question de la représentativité de l’empreinte métabolomique générée (dépendante des différents niveaux de variabilité biologique et analytique) ou encore de son exhaustivité (dépendante de la technique de mesure), se pose clairement dans le cadre de ces approches et limite parfois la significativité des résultats obtenus et leur interprétation.
 ). Une première perspective est en effet la mise en évidence globale, bien au-delà du faible nombre de « end-points » usuellement étudiés, de l’impact de certaines substances sur des systèmes biologiques donnés (Robertson et coll., 2000
). Une première perspective est en effet la mise en évidence globale, bien au-delà du faible nombre de « end-points » usuellement étudiés, de l’impact de certaines substances sur des systèmes biologiques donnés (Robertson et coll., 2000 ; Shockcor et coll., 2002
; Shockcor et coll., 2002 ; Griffin et coll., 2004
; Griffin et coll., 2004 ; Ebbels et coll., 2007
; Ebbels et coll., 2007 ). Une seconde perspective est l’identification de biomarqueurs d’exposition et/ou d’effet permettant la génération de nouvelles connaissances s’agissant de leurs modes d’actions (Clarke et coll., 2008
). Une seconde perspective est l’identification de biomarqueurs d’exposition et/ou d’effet permettant la génération de nouvelles connaissances s’agissant de leurs modes d’actions (Clarke et coll., 2008 ; Coen et coll., 2008
; Coen et coll., 2008 ; Coen, 2010
; Coen, 2010 ; Beger et coll., 2010
; Beger et coll., 2010 ). Dans ce domaine, il est toutefois souvent difficile de distinguer les modifications métaboliques résultant d’une adaptation physiologique de celles signant une toxicité effective. Par ailleurs, la question de la représentativité de l’empreinte métabolomique générée (dépendante des différents niveaux de variabilité biologique et analytique) ou encore de son exhaustivité (dépendante de la technique de mesure), se pose clairement dans le cadre de ces approches et limite parfois la significativité des résultats obtenus et leur interprétation.
). Dans ce domaine, il est toutefois souvent difficile de distinguer les modifications métaboliques résultant d’une adaptation physiologique de celles signant une toxicité effective. Par ailleurs, la question de la représentativité de l’empreinte métabolomique générée (dépendante des différents niveaux de variabilité biologique et analytique) ou encore de son exhaustivité (dépendante de la technique de mesure), se pose clairement dans le cadre de ces approches et limite parfois la significativité des résultats obtenus et leur interprétation.S’agissant des phtalates, une étude métabolomique conduite chez le rat Wistar (van Ravenzwaay et coll., 2009 ) s’est ainsi attachée à caractériser la signature plasmatique consécutive à une exposition aux phtalates (DEHP et/ou DBP). Cette étude a montré en particulier l’existence d’un niveau de concentration sans effet global au niveau du métabolome (150 mg/kg), et pour de plus fortes concentrations (1 000 à 3 000 mg/kg) une signature consécutive à une exposition en mélange significativement distincte de celle observée pour les deux composés testés considérés séparément. L’étude d’Eveillard et coll. (2009
) s’est ainsi attachée à caractériser la signature plasmatique consécutive à une exposition aux phtalates (DEHP et/ou DBP). Cette étude a montré en particulier l’existence d’un niveau de concentration sans effet global au niveau du métabolome (150 mg/kg), et pour de plus fortes concentrations (1 000 à 3 000 mg/kg) une signature consécutive à une exposition en mélange significativement distincte de celle observée pour les deux composés testés considérés séparément. L’étude d’Eveillard et coll. (2009 ), également dédiée à l’étude de l’impact du DEHP au niveau hépatique chez la souris, a quant à elle illustré l’intégration possible de données transcriptomiques et métabolomiques issues d’un même système étudié. S’agissant des substances perfluorées (PFC) une étude métabolomique a été conduite chez le rat Sprague-Dawley pour caractériser la signature plasmatique d’une exposition au N-ethyl perfluorooctanesulfonamidoethanol (N-EtFOSE) (Xie et coll., 2009
), également dédiée à l’étude de l’impact du DEHP au niveau hépatique chez la souris, a quant à elle illustré l’intégration possible de données transcriptomiques et métabolomiques issues d’un même système étudié. S’agissant des substances perfluorées (PFC) une étude métabolomique a été conduite chez le rat Sprague-Dawley pour caractériser la signature plasmatique d’une exposition au N-ethyl perfluorooctanesulfonamidoethanol (N-EtFOSE) (Xie et coll., 2009 ), révélant des marqueurs impliqués dans la prolifération de peroxysomes.
), révélant des marqueurs impliqués dans la prolifération de peroxysomes.
 ) s’est ainsi attachée à caractériser la signature plasmatique consécutive à une exposition aux phtalates (DEHP et/ou DBP). Cette étude a montré en particulier l’existence d’un niveau de concentration sans effet global au niveau du métabolome (150 mg/kg), et pour de plus fortes concentrations (1 000 à 3 000 mg/kg) une signature consécutive à une exposition en mélange significativement distincte de celle observée pour les deux composés testés considérés séparément. L’étude d’Eveillard et coll. (2009
) s’est ainsi attachée à caractériser la signature plasmatique consécutive à une exposition aux phtalates (DEHP et/ou DBP). Cette étude a montré en particulier l’existence d’un niveau de concentration sans effet global au niveau du métabolome (150 mg/kg), et pour de plus fortes concentrations (1 000 à 3 000 mg/kg) une signature consécutive à une exposition en mélange significativement distincte de celle observée pour les deux composés testés considérés séparément. L’étude d’Eveillard et coll. (2009 ), également dédiée à l’étude de l’impact du DEHP au niveau hépatique chez la souris, a quant à elle illustré l’intégration possible de données transcriptomiques et métabolomiques issues d’un même système étudié. S’agissant des substances perfluorées (PFC) une étude métabolomique a été conduite chez le rat Sprague-Dawley pour caractériser la signature plasmatique d’une exposition au N-ethyl perfluorooctanesulfonamidoethanol (N-EtFOSE) (Xie et coll., 2009
), également dédiée à l’étude de l’impact du DEHP au niveau hépatique chez la souris, a quant à elle illustré l’intégration possible de données transcriptomiques et métabolomiques issues d’un même système étudié. S’agissant des substances perfluorées (PFC) une étude métabolomique a été conduite chez le rat Sprague-Dawley pour caractériser la signature plasmatique d’une exposition au N-ethyl perfluorooctanesulfonamidoethanol (N-EtFOSE) (Xie et coll., 2009 ), révélant des marqueurs impliqués dans la prolifération de peroxysomes.
), révélant des marqueurs impliqués dans la prolifération de peroxysomes.Deux autres études, non plus de type métabolomique au sens strict (c’est-à-dire global et non ciblé) mais de profilage métabolique, limitées à un ensemble de métabolites du même type détectés de façon ciblée, ont également été conduites pour étudier l’impact d’une exposition au bisphénol A sur les nucléosides urinaires chez le rat (Cho et coll., 2009 ) ou l’impact d’une exposition au DEHP sur les principaux lipides dans le tissus cérébral d’embryons de rat (Xu et coll., 2007
) ou l’impact d’une exposition au DEHP sur les principaux lipides dans le tissus cérébral d’embryons de rat (Xu et coll., 2007 ).
).
 ) ou l’impact d’une exposition au DEHP sur les principaux lipides dans le tissus cérébral d’embryons de rat (Xu et coll., 2007
) ou l’impact d’une exposition au DEHP sur les principaux lipides dans le tissus cérébral d’embryons de rat (Xu et coll., 2007 ).
).Au-delà de ces premières études, la métabolomique reste aujourd’hui un domaine en développement, sur le plan des concepts comme sur le plan des outils et procédures, et ne permet pas encore d’appréhender la complexité de la relation entre exposition chimique (dans les conditions réelle de basses concentrations et de multi-exposition) et troubles de la reproduction (à l’échelle individuelle ou épidémiologique) telle que décrite à la figure 20.2 . Des travaux sont toutefois en cours dans ce sens (Antignac et coll., communication personnelle), s’attachant à établir chez l’Homme un lien (au mieux de corrélation) entre d’une part un profil d’imprégnation chimique le plus large possible (mesure quantitative et ciblée d’un grand nombre de substances incluant différentes classes de contaminants chimiques), d’autre part une empreinte métabolomique globale (par exemple dans le plasma ou sérum), et enfin des paramètres cliniques indicateurs de fonctions de la reproduction ou du développement (par exemple de la qualité spermatique chez l’homme).
. Des travaux sont toutefois en cours dans ce sens (Antignac et coll., communication personnelle), s’attachant à établir chez l’Homme un lien (au mieux de corrélation) entre d’une part un profil d’imprégnation chimique le plus large possible (mesure quantitative et ciblée d’un grand nombre de substances incluant différentes classes de contaminants chimiques), d’autre part une empreinte métabolomique globale (par exemple dans le plasma ou sérum), et enfin des paramètres cliniques indicateurs de fonctions de la reproduction ou du développement (par exemple de la qualité spermatique chez l’homme).
 . Des travaux sont toutefois en cours dans ce sens (Antignac et coll., communication personnelle), s’attachant à établir chez l’Homme un lien (au mieux de corrélation) entre d’une part un profil d’imprégnation chimique le plus large possible (mesure quantitative et ciblée d’un grand nombre de substances incluant différentes classes de contaminants chimiques), d’autre part une empreinte métabolomique globale (par exemple dans le plasma ou sérum), et enfin des paramètres cliniques indicateurs de fonctions de la reproduction ou du développement (par exemple de la qualité spermatique chez l’homme).
. Des travaux sont toutefois en cours dans ce sens (Antignac et coll., communication personnelle), s’attachant à établir chez l’Homme un lien (au mieux de corrélation) entre d’une part un profil d’imprégnation chimique le plus large possible (mesure quantitative et ciblée d’un grand nombre de substances incluant différentes classes de contaminants chimiques), d’autre part une empreinte métabolomique globale (par exemple dans le plasma ou sérum), et enfin des paramètres cliniques indicateurs de fonctions de la reproduction ou du développement (par exemple de la qualité spermatique chez l’homme).Bibliographie
[1] ANTIGNAC JP, COURANT F, PINEL G, MONTEAU F, LE BIZEC B. Mass spectrometric based metabolomic in the field of chemical risk: state of the art, pitfalls, and future trends.
TrAC Trends in Anal Chem. 2011, in press (doi: 10.1016/j.trac.2010.11.003);


[2] BEGER RD, SUN J, SCHNACKENBERG LK. Metabolomics approaches for discovering biomarkers of drug-induced hepatotoxicity and nephrotoxicity.
Toxicol Appl Pharmacol. 2010;
243:154- 166

[3] BETANCOURT AM, MOBLEY JA, RUSSO J, LAMARTINIERE C. A. Proteomic analysis in mammary glands of rat offspring exposed in utero to bisphenol A.
J Proteomics. 2010;
73:1241- 1253

[4] CHO SH, CHOI MH, KWON OS, LEE WY, CHUNG BC. Metabolic significance of bisphenol A-induced oxidative stress in rat urine measured by liquid chromatography-mass spectrometry.
J Appl Toxicol. 2009;
29:110- 117

[5] CLARKE CJ, HASELDEN JN. Metabolic profiling as a tool for understanding mechanisms of toxicity.
Toxicol Pathol. 2008;
36:140- 147

[6] COEN M. A metabonomic approach for mechanistic exploration of pre-clinical toxicology.
Toxicology. 2010;
278:326- 340

[7] COEN M, HOLMES E, LINDON JC, NICHOLSON JK. NMR-based metabolic profiling and metabonomic approaches to problems in molecular toxicology.
Chem Res Toxicol. 2008;
21:9- 27

[8] EBBELS TM, KEUN HC, BECKONERT OP, BOLLARD ME, LINDON JC, et coll. Prediction and classification of drug toxicity using probabilistic modeling of temporal metabolic data: the consortium on metabonomic toxicology screening approach.
J Proteome Res. 2007;
6:4407- 4422

[9] EVEILLARD A, LASSERRE F, DE TAYRAC M, POLIZZI A, CLAUS S, et coll. Identification of potential mechanisms of toxicity after di-(2-ethylhexyl)-phthalate (DEHP) adult exposure in the liver using a systems biology approach.
Toxicol Appl Pharmacol. 2009;
236:282- 292

[10] GRIFFIN JL, BOLLARD ME. Metabonomics: its potential as a tool in toxicology for safety assessment and data integration.
Curr Drug Metab. 2004;
5:389- 398

[11] KLING P, FORLIN L. Proteomic studies in zebrafish liver cells exposed to the brominated flame retardants HBCD and TBBPA.
Ecotoxicol Environ Saf. 2009;
72:1985- 1993

[12] LAWSON C, GIESKE M, MURDOCH B, YE P, LI Y, et coll. Gene expression in the fetal mouse ovary is altered by exposure to low doses of bisphenol A.
Biol Reprod. 2011;
84:79- 86

[13] LINDON JC, KEUN HC, EBBELS TM, PEARCE JM, HOLMES E, NICHOLSON JK. The Consortium for Metabonomic Toxicology (COMET): aims, activities and achievements.
Pharmacogenomics. 2005;
6:691- 699

[14] MORAL R, WANG R, RUSSO IH, LAMARTINIERE CA, PEREIRA J, RUSSO J. Effect of prenatal exposure to the endocrine disruptor bisphenol A on mammary gland morphology and gene expression signature.
J Endocrinol. 2008;
196:101- 112

[15] NICHOLSON JK, LINDON JC, HOLMES E. Metabonomics : understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data.
Xenobiotica. 1999;
29:1181- 1189

[16] ROBERTSON DG, REILY MD, SIGLER RE, WELLS DF, PATERSON DA, BRADEN TK. Metabonomics: evaluation of nuclear magnetic resonance (NMR) and pattern recognition technology for rapid in vivo screening of liver and kidney toxicants.
Toxicol Sci. 2000;
57:326- 337

[17] SHI X, YEUNG LW, LAM PK, WU RS, ZHOU B. Protein profiles in zebrafish (Danio rerio) embryos exposed to perfluorooctane sulfonate.
Toxicol Sci. 2009;
110:334- 340

[18] SHOCKCOR JP, HOLMES E. Metabonomic applications in toxicity screening and disease diagnosis.
Curr Top Med Chem. 2002;
2:35- 51

[19] VAN RAVENZWAAY B, COELHO-PALERMO CUNHA G, STRAUSS V, WIEMER J, LEIBOLD E, et coll. The individual and combined metabolite profiles (metabolomics) of dibutylphthalate and di(2-ethylhexyl)phthalate following a 28-day dietary exposure in rats.
Toxicol Lett. 2010;
198:159- 170

[20] WEI Y, CHAN LL, WANG D, ZHANG H, WANG J, DAI J. Proteomic analysis of hepatic protein profiles in rare minnow (Gobiocypris rarus) exposed to perfluorooctanoic acid.
J Proteome Res. 2008;
7:1729- 1739

[21] XIE W, WU Q, KANIA-KORWEL I, THARAPPEL JC, TELU S, et coll. Subacute exposure to N-ethyl perfluorooctanesulfonamidoethanol results in the formation of perfluorooctanesulfonate and alters superoxide dismutase activity in female rats.
Arch Toxicol. 2009;
83:909- 924

[22] XU Y, AGRAWAL S, COOK TJ, KNIPP GT. Di-(2-ethylhexyl)-phthalate affects lipid profiling in fetal rat brain upon maternal exposure.
Arch Toxicol. 2007;
81:57- 62

[23] YANG M, LEE HS, PYO MY. Proteomic biomarkers for prenatal bisphenol A-exposure in mouse immune organs.
Environ Mol Mutagen. 2008;
49:368- 373

[24] YUAN X, JONKER MJ, DE WILDE J, VERHOEF A, WITTINK FR, et coll. Finding maximal transcriptome differences between reprotoxic and non-reprotoxic phthalate responses in rat testis.
J Appl Toxicol. 2010, doi: 10.1002/jat.1601. (Epub ahead of print);


→ Aller vers SYNTHESE