| |
| Med Sci (Paris). 2003 October; 19(10): 950–954. Published online 2003 October 15. doi: 10.1051/medsci/20031910950.Génétique moléculaire humaine : des maladies monogéniques aux maladies complexes François Rousseau1 and Nathalie Laflamme2* 1Unité de recherche en génétique humaine et moléculaire, Centre de recherche de l’hôpital St-François-d’Assise du CHUQ, Université Laval, 10, rue de l’Espinay, Québec G1L 3L5, Canada 2Institut national de santé publique du Québec, Québec, Canada |
La pathogénie de la majorité des maladies implique l’interaction entre le bagage génétique d’un individu et son environnement, et ce dans des proportions variables selon la maladie. À une extrémité du spectre, on retrouve les affections « purement héréditaires », telle la dystrophie musculaire de Duchenne, dont l’apparition chez un individu repose exclusivement sur la présence d’une mutation importante du gène DMD. À l’autre extrémité du spectre se trouvent des conditions « purement environnementales », telle l’amputation accidentelle d’un doigt chez un travailleur de la construction. Entre ces deux extrêmes, se situe la vaste majorité des maladies humaines, résultant de l’effet de l’environnement sur le génome particulier et unique d’un individu. L’avènement des outils modernes de biologie moléculaire au cours des années 1970, puis leur application à la génétique humaine dans les années 1980 ont conduit à une augmentation rapide et significative des connaissances, à la fois sur l’étiologie de plusieurs maladies héréditaires et sur le fonctionnement du vivant, au sens très large. Les outils de la biologie et de la génétique moléculaires ont été appliqués successivement à l’étude de la génétique des maladies monogéniques classiques [
1–
3], à la cartographie systématique du génome humain [
4–
6], puis à son séquencage [
7,
8] et, enfin, à l’étude des maladies fréquentes à forte composante génétique (ou maladies génétiques complexes) [
9–
12]. L’identification des mutations géniques, puis de la fonction du (des) produit(s) de ces gènes, permet de révéler la biologie normale et celle de la maladie. Les maladies génétiques complexes peuvent être définies comme faisant défaut aux lois de Mendel, car elles sont plutôt déterminées par l’interaction de plusieurs facteurs génétiques et environnementaux [
13]. Les gènes impliqués dans les maladies complexes (ou polygéniques) sont pressentis comme ayant des effets individuels plus modestes que les gènes à forte pénétrance, impliqués dans les maladies héréditaires classiques mendeliennes [
14] (Figure 1). En corollaire, l’identification des gènes associés aux maladies complexes demande d’adopter des stratégies et des méthodes différentes, aussi bien en termes de recrutement des sujets de recherche qu’en termes des méthodologies utilisées [9].
 | Figure 1.
Caractéristiques comparées des maladies héréditaires classiques mendeliennes et des maladies génétiques complexes. L’abscisse représente le nombre estimé de gènes impliqués dans la maladie, tandis que l’ordonnée indique la proportion de la variance phénotypique expliquée par chacun de ces gènes. Le nombre habituel d’individus à étudier pour identifier ce ou ces gènes est aussi indiqué, montrant que les études visant à identifier les gènes impliqués dans des maladies génétiques complexes nécessitent l’analyse de plus de sujets, étant donné l’effet plus modeste de chaque gène impliqué. |
|
La séquence préliminaire du génome humain, rendue publique en février 2001, identifiait environ 30 000 gènes humains potentiels [7, 8]. Les plus récentes estimations n’ont pas changé ce nombre, qui représente environ seulement deux fois plus de gènes que D. melanogaster (la mouche à fruits), qui en possède près de 13 500. On ne connaît pas encore la fonction de tous ces gènes et beaucoup d’efforts de recherche sont portés vers cette question. Le projet génome humain a aussi permis de constater que, bien que les êtres humains diffèrent entre eux par moins de 1 % du génome (toutes ethnies confondues), on retrouve une différence de séquence toutes les 1250paires de bases entre deux copies d’un segment d’ADN donné prises au hasard [8]. Certaines de ces différences de séquence entre les « variantes » de génome humain se trouvent dans des gènes et en affectent la fonction à un degré plus ou moins important. Il est raisonnable de penser que les variantes affectant très sévèrement la fonction d’un gène seront plus rares dans la population que celles ne l’affectant que modérément ou pas du tout. La disponibilité de la séquence complète du génome humain (bien qu’encore non finalisée encore) et d’un grand nombre de variantes génétiques (identifiées en comparant la séquence de plusieurs individus) ont donné aux généticiens des outils leur permettant de s’attaquer à la génétique des maladies polygéniques, plus complexes (par définition) que les monogéniques. Mais les outils techniques (méthodes de génotypage) et méthodologiques (méthodes d’analyse statistiques) ont encore besoin d’être significativement améliorées. |
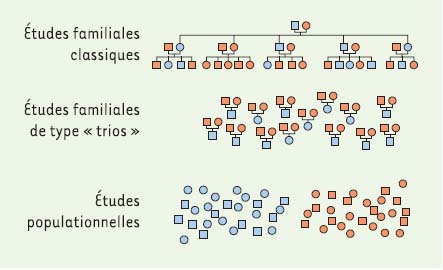
Dispositifs expérimentaux Parmi les phénotypes ou maladies polygéniques importantes aujourd’hui à l’étude, on retrouve entre autres la densité osseuse (héritabilité ≈ 0,80), les formes non familiales de cancer (cancer du sein : héritabilité ≈ 0,30, cancer colorectal : héritabilité ≈ 0,35, cancer de la prostate : héritabilité ≈ 0,40…), l’asthme (héritabilité ≈ 0,60), les maladies inflammatoires de l’intestin (héritabilité ≈ 0,50), le diabète de type I (héritabilité ≈ 0,50), l’obésité (héritabilité ≈ 0,65), l’hypertension artérielle (héritabilité ≈ 0,80) et la schizophrénie (héritabilité ≈ 0,83). Il existe trois grandes classes de dispositifs expérimentaux permettant d’identifier les gènes impliqués dans les maladies complexes (Figure 2). Le premier implique le recrutement de grandes familles sur plusieurs générations, le second étudie des « trios » d’individus provenant d’une même famille (typiquement père + mère + cas) et le dernier implique le recrutement d’échantillons de population: cas et témoins appariés pour les phénotypes qualitatifs (cancer), ou bien échantillonnage populationnel pour les phénotypes quantitatifs (densité osseuse).
 | Figure 2.
Dispositifs expérimentaux permettant d’identifier les gènes impliqués dans les maladies complexes. Les études familiales classiques demandent le recrutement du maximum de sujets possible (atteints et non atteints), ainsi que du plus grand nombre possible de familles apparentées, et ce sur le plus grand nombre de générations possibles. Les études familiales de type « trios » visent en général le recrutement de sujets non apparentés atteints de la maladie et de leurs deux parents biologiques (ou, à défaut, de frères ou sœurs): on se sert alors des allèles non transmis comme représentatifs de la population non atteinte. Les études populationnelles (ou études cas-témoins) adoptent la stratégie de recruter un grand nombre de sujets non apparentés atteints d’une même maladie et d’un nombre au moins équivalent de sujets témoins, soigneusement appariés à chaque cas pour leur origine ethnique et pour les principaux facteurs de risque connus de la maladie à l’étude. |
Ces différents dispositifs ont tous leurs avantages et leurs inconvénients. Les familles sont plus complexes à recruter, mais le degré d’apparentement entre les individus peut être utilisé pour identifier des allèlestémoins (c’est aussi le cas des études de « trios »). Les trios sont plus faciles à recruter, mais cette approche manque de puissance à cause de la perte d’information génétique chez les parents non informatifs [
15]. De plus, si les deux parents ne sont pas disponibles (comme c’est souvent le cas pour des maladies à apparition tardive), il faut alors recourir à d’autres individus apparentés (fratrie) et déduire les allèles parentaux [
16]. Enfin, les études fondées sur des échantillons de population de grande qualité autorisent un recrutement relativement rapide et moins coûteux de grands échantillons, mais sont plus sujettes à des résultats artéfactuels. Elles doivent donc être répliquées dans des échantillons indépendants. Elles permettent cependant de tenir compte des autres déterminants non génétiques impliqués dans la maladie et d’étudier les interactions gène-gène et gène-environnement. Il est aussi plus aisé d’extrapoler leurs résultats à la population générale. Étant donné ces différents avantages et inconvénients, l’utilisation d’un dispositif d’étude plutôt qu’un autre dépend du phénotype étudié et des objectifs de la recherche. Jusqu’à présent, pour les maladies complexes, les chercheurs ont beaucoup utilisé des approches de type épidémiologique, comme des études cas-témoins de haute qualité qui ont la puissance nécessaire pour détecter les effets attendus de taille plus modeste [
17,
18]. |
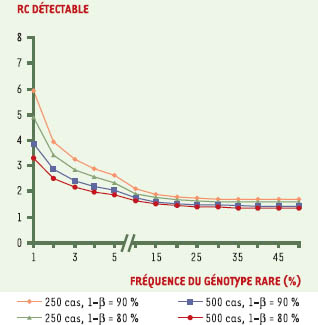
Puissance statistique et taille d’échantillon La détection d’association entre génotype et phénotype pour les traits complexes nécessite d’autant plus de puissance que le génotype étudié est rare, ou que son effet est modeste. Il existe beaucoup de littérature au sujet de la puissance nécessaire à l’étude des maladies polygéniques. À titre d’exemple, la Figure 3 représente l’effet détectable d’un génotype (rapport de cotes) d’une étude cas-témoins en fonction de la prévalence du génotype rare, selon la taille de l’échantillon et selon la puissance désirée. On peut constater l’importance d’étudier de grands échantillons. Enfin, la puissance de l’étude est affectée négativement par le problème des analyses multiples rencontré lorsque l’on teste plusieurs hypothèses ou gènes à la fois, dans des combinaisons différentes. Il est toujours préférable de travailler avec des hypothèses de départ, et être en mesure de reproduire les résultats dans un échantillon indépendant est toujours souhaitable. On peut également réaliser des analyses de permutation pour tester la stabilité de l’association observée [17].
 | Figure 3.
Effet détectable d’un génotype (rapport de cotes) d’une étude cas-témoins en fonction de la prévalence du génotype rare, selon la taille de l’échantillon et selon la puissance désirée. Calcul de puissance montrant le rapport de cotes (RC) détectable par une étude d’association génétique de type cas-témoins en fonction du nombre de cas: n = 250, losanges oranges (puissance de 90%) ou triangles verts (puissance de 80%); n = 500, carrés bleus (puissance de 90%) ou cercles rouges (puissance de 80%) et en fonction de la fréquence populationnelle du génotype rare du marqueur génétique candidat étudié. Le calcul est fondé sur un groupe témoins deux fois plus nombreux que le nombre de cas et sur une erreur α fixée à 0,05. |
|
Pour toute étude génétique, la qualité des données utilisées est cruciale. Ce phénomène n’est pas uniquement propre à ce type d’études, mais les études de liaison génétique ont vite fait ressortir l’importance de la très grande qualité du phénotypage pour assurer la qualité des résultats. Une trop grande hétérogénéité dans la qualité des données phénotypiques, ou un grand nombre d’information manquantes peuvent affecter la puissance de l’étude, en plus d’augmenter le risque d’association artéfactuelle entre le phénotype et le génotype. Un autre phénomène récemment mis en lumière est l’importance de la qualité des données génotypiques. En effet, beaucoup de méthodologies de génotypage s’accompagnent d’un taux indéterminé de faux génotypes, ou ne produisent pas de résultat pour une proportion très significative des échantillons analysés (souvent entre 5% et 10%). Cela peut affecter la puissance d’une étude, augmentant exponentiellement l’ampleur du problème à mesure que les analyses se complexifient et que plusieurs locus sont simultanément analysés. De plus, les erreurs de génotypage peuvent causer une perte artéfactuelle de l’équilibre de Hardy-Weinberg, qui est parfois utilisé comme test d’association génétique. Malheureusement, bien qu’il existe plusieurs dizaines de méthodes de génotypage performantes en termes de débit, peu d’entres elles ont été étudiées systématiquement en regard de performances analytiques (sensibilité, spécificité et taux de complétude). Par exemple, une méthode de génotypage donnant un résultat pour 95% des échantillons (ce qui peut paraître satisfaisant à première vue) signifie que 5% des échantillons n’ont pas de génotype à chaque fois qu’un marqueur est testé. De plus, notre expérience montre que, lorsque l’ADN est purifié à l’aide d’une méthode bien standardisée (telle les colonnes de Qiagen), ce ne sont généralement pas toujours les mêmes échantillons qui ne produisent pas de résultat: la proportion des échantillons n’ayant pas de génotype pour tous les marqueurs augmente donc avec le nombre de marqueurs testés: 5% au premier, 10% au deuxième, 15% aux troisième, et ainsi de suite… D’où l’importance de connaître a priori la performance de la méthode de génotypage qui sera choisie, car elle peut varier entre 1% et 10% de non-complétude à chaque marqueur. Non moins important est le taux de faux génotypes obtenu avec la méthode, qui pourra tout autant affecter les résultats [15,
19,
20]. |
Le transfert technologique vers la clinique Bien que plusieurs gènes impliqués dans les maladies monogéniques classiques aient été identifiés, la pleine exploitation en clinique de ces nouveaux outils de la génétique moléculaire a été retardée en partie à cause d’un manque de données fiables sur leurs performances diagnostiques (sensibilités et spécificités analytique et clinique) et la prévalence des mutations dans la population [
21]. Le développement et l’implantation en clinique de nouveaux outils diagnostiques est un processus complexe exigeant une approche multidisciplinaire de recherche translationnelle et évaluative. Des études réalisées sur de grands échantillons d’individus ayant un statut pathologique bien défini, selon des critères établis et rigoureusement évalués, sont nécessaires à la détermination de la performance analytique (le test peut déterminer de façon fiable - sensible et spécifique- la présence d’une substance ou d’une mutation dans un échantillon) et de la performance clinique (capacité à distinguer un individu en bonne santé d’un individu malade). Cette étape de l’évaluation a sou-vent été absente, ou seulement partiellement réalisée, ce qui a mené à une situation où certains tests valables n’ont été introduits en clinique que très laborieusement - faute de données fiables justifiant ces dépenses, alors que d’autres tests ont été introduits trop précocément en clinique sans que l’on sache vraiment la signification d’un résultat positif ou négatif. Plusieurs questions se posent quand bien même des données fiables démontrent la bonne performance analytique et clinique d’un nouveau test: doit-il être introduit ? Vient-il se substituer à une autre analyse courante ou bien s’ajoute-t-il à l’arsenal diagnostique ? Dans quelle séquence (ou algorithme) doit-il être utilisé en conjonction avec les tests existants ? Son usage doit-il être restreint à certains groupes à plus haut risque ? Son introduction affectera-t-elle les coûts de santé ? Si oui, comment ? Estce que la nouvelle technologie réduira (par l’élimination d’interventions plus coûteuses) ou augmentera (par l’induction de procédures complémentaires) les coûts globaux de santé ? Les nouveaux tests moléculaires n’échappent évidemment pas à cette nécessaire évaluation, mais ont rarement été soumis à une étude systématique de leurs performances analytiques et cliniques. Ces études nécessitent l’analyse de centaines, voire de milliers d’échantillons d’ADN provenant d’individus porteurs et normaux. Pour les tests moléculaires pour lesquels il est possiblement envisagé de procéder à une forme de dépistage systématique (tel le syndrome de l’X-fragile, l’hémochromatose, l’ostéoporose, le cancer du sein, la pré-éclampsie ou les dyslipidémies), des estimations fiables de la prévalence allélique des mutations à étudier dans la population visée doivent être obtenus [
22–
24]. |
Conclusions et perspectives La complétion du projet génome prévue pour bientôt, combinée aux multiples initiatives visant à débusquer les gènes et combinaisons de gènes déterminant la susceptibilité individuelle à développer une maladie à composante génétique importante mènera indubitablement à l’apparition d’une médecine rendue plus complexe encore par l’interprétation de multiples variables génétiques et environnementales. Les améliorations technologiques permettent maintenant de réaliser des études génétiques à très grande échelle à des coûts relativement abordables, par exemple équivalents à ceux d’un dosage de la vitamine D, et même 20 fois moins coûteux pour des méthodes de dépistage ayant des performances analytiques légèrement inférieures. Sans aucun doute devrions-nous assister à une certaine révolution de la pratique médicale en corollaire de ces découvertes, dont le rythme va s’accélérer au cours de la prochaine décennie. Et ce sont les médecins présents et futurs qui seront aux premières lignes de l’utilisation rationnelle et efficace de ces nouvelles connaissances. |
1. Koenig M, Hoffman EP, Bertelson CJ, et al. Complete cloning of the Duchenne muscular dystrophy (DMD) cDNA and preliminary genomic organization of the DMD gene in normal and affected individuals. Cell 1987; 50: 509–17. 2. Kerem B, Rommens JM, Buchanan JA, et al. Identification of the cystic fibrosis gene: genetic analysis. Science 1989; 245: 1073–80. 3. Oberle I, Rousseau F, Heitz D, et al. Instability of a 550-base pair DNA segment and abnormal methylation in fragile X syndrome. Science 1991; 252: 1097–102. 4. A comprehensive genetic linkage map of the human genome. NIH/CEPH Collaborative Mapping Group. Science 1992; 258: 67–86. 5. Donis-Keller H, Green P, Helms C, et al. A genetic linkage map of the human genome. Cell 1987; 51: 319–37. 6. Dib C, Faure S, Fizames C, et al. A comprehensive genetic map of the human genome based on 5,264 microsatellites. Nature 1996; 380: 152–4. 7. Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human genome. Nature 2001; 409: 860–921. 8. Venter JC, Adams MD, Myers EW, et al. The sequence of the human genome. Science 2001; 291: 1304–51. 9. Rao DC. Genetic dissection of complex traits: an overview. Adv Genet 2001; 42: 13–34. 10. Giguere Y, Rousseau F. The genetics of osteoporosis: complexities and difficulties. Clin Genet 2000; 57: 161–9. 11. Giguere Y, Dodin S, Blanchet C, et al. The association between heel ultrasound and hormone replacement therapy is modulated by a two-locus vitamin D and estrogen receptor genotype. J Bone Miner Res 2000; 15: 1076–84. 12. Giguere Y, Dewailly E, Brisson J, et al. Short polyglutamine tracts in the androgen receptor are protective against breast cancer in the general population. Cancer Res 2001; 61: 5869–74. 13. Thompson G. Significance levels in genome scans. Adv Genet 2001; 42: 475–86. 14. Ponder B. Cancer genetics. Nature 2001; 411: 336–95. 15. Kirk KM, Cardon LR. The impact of genotyping error on haplotype reconstruction and frequency estimation. Eur J Hum Genet 2002; 10: 616–22. 16. Morton NE, Collins A. Tests and estimates of allelic association in complex inheritance. Proc Natl Acad Sci USA 1998; 95: 11389–93. 17. Cardon L, Bell J. Association studies designs for complex diseases. Nat Rev Genet 2001; 2: 91–9. 18. Longmate J. Complexity and power in case-control association studies. Am J Hum Genet 2001; 68: 1229–37. 19. Hoh J, Wille A, Ott J. Trimming, weighting, and grouping SNPs in human case-control association studies. Genome Res 2001; 11: 2115–9. 20. Xu J, Turner A, Little J, et al. Positive results in association studies are associated with departure from Hardy-Weinberg equilibrium: hint for genotyping error ? Hum Genet 2002; 111: 573–4. 21. Committee on DNA-Diagnostics. DNA Diagnostics. Rijswijk: Health Council of the Netherlands, 1998. 22. Rousseau F, Rouillard P, Morel ML, et al. Prevalence of carriers of premutationsize alleles of the FMRI gene and implications for the population genetics of the fragile X syndrome. Am J Hum Genet 1995; 57: 1006–18. 23. Dombrowski C, Levesque S, Morel ML, et al. Premutation and intermediate-size FMR1 alleles in 10572 males from the general population: loss of an AGG interruption is a late event in the generation of fragile X syndrome alleles. Hum Mol Genet 2002; 11: 371–8. 24. Girouard J, Giguère Y, Delage R, et al. Prevalence of HFE gene C282Y and H63D mutations in a French-Canadian population of neonates and in referred patients. Hum Mol Genet 2002; 11: 185–9. |