| |
| Med Sci (Paris). 2003 August; 19(8-9): 885–891. Published online 2003 August 15. doi: 10.1051/medsci/20031989885.SRAS : 1. Le virus Isabelle Tratner* médecine/sciences, Faculté Xavier-Bichat, 16, rue Henri Huchard, 75018 Paris, France |
Qui sont les coronavirus ? Les coronavirus sont des virus sphériques enveloppés, de 60 à 220 nm de diamètre, dont la structure, en partie encore hypothétique, comporterait une nucléocapside hélicoïdale à l’intérieur d’une capside de structure icosahédrique, ellemême entourée d’une enveloppe membranaire (Figure 1) [
1–
4]. Ils ont été nommés d’après leur aspect en couronne en microscopie électronique (Figure 2).
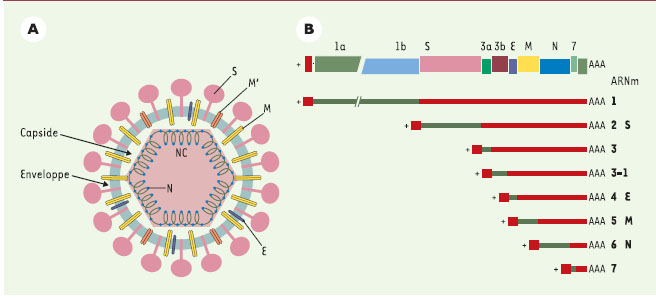
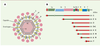 | Figure 1.
Structure des coronavirus. A. Représentation schématique d’une particule virale. L’enveloppe est formée des protéines S ( spike), M et M’ (membranaires) et E (enveloppe). La nucléocapside (NC), formée par l’ARN génomique associé à la protéine N, est contenue dans la capside, elle-même entourée de l’enveloppe. B. Structure schématique de l’ARN génomique et des ARN subgénomiques d’un coronavirus prototype. L’ARN génomique (brin +) code pour les protéines d’enveloppe et de nucléocapside ainsi que pour la réplicase, transcrite à partir de l’ORF ( open reading frame) 1a puis de l’ORF 1b par changement de phase de lecture. La polyprotéine produite par l’ORF 1a/1b est ensuite protéolysée en diverses protéines qui forment le complexe réplicatif. Les protéines structurales S, M, N et E sont traduites à partir de la première phase de lecture (en vert) des ARNm initiés en aval dans la séquence génomique du coronavirus. À l’extrémité 5’ des ARN, une séquence 5’- leader est présente, identique à l’extrémité 5’ de l’ARN génomique (boîte rouge). AAA = polyadénylation (d’après [
5]). |
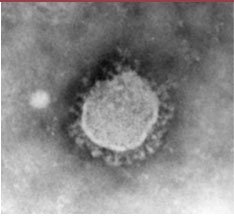
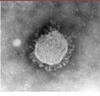 | Figure 2.
Aspect d’un coronavirus en microscopie électronique. Le nom coronavirus provient de l’aspect en couronne des spicules formées par la protéine S à la surface de l’enveloppe virale (photo, Pierre Lebon). |
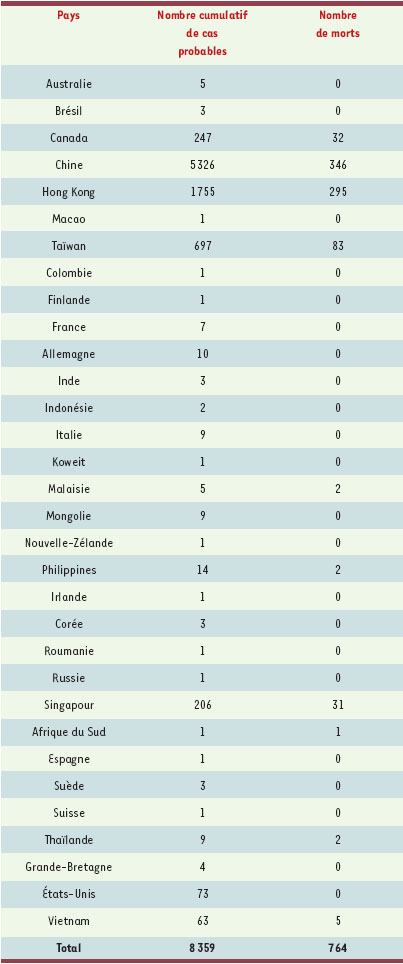
 | Tableau I.
Situation mondiale le 17 juin 2003 (données OMS: http://www.
who.int/csr/sars/country/en/). |
Biologie moléculaire Coronavirus connus Leur génome est constitué d’un ARN linéaire simple brin, non segmenté, de polarité positive (qui peut donc être directement traduit en protéine), d’environ 30 kb (le plus grand des virus à ARN) [ 4] et qui code pour 7 à 10 protéines. Certaines de ces protéines sont bien caractérisées comme la réplicase et les protéines structurales N (nucléocapside), S ( spike), E (enveloppe, aussi appelée sM) et M et M’ (membrane), ainsi que la protéine de surface HE ( haemagglutinin esterase) qui n’est présente que chez certains coronavirus. La protéine N est une nucléoprotéine qui s’associe à l’ARN pour former la nucléocapside. La protéine S, comme la protéine E, est une protéine de l’enveloppe. Glycoprotéine de grande taille (de 1 100 à 1 450 acides aminés), elle forme des extensions (spicules) à la surface de la particule virale et est responsable de l’attachement à la cellule hôte et de la fusion membranaire lors de l’infection, ainsi que de l’induction d’anticorps neutralisants. La protéine M est la protéine majoritaire de la capside, mais elle est également insérée dans l’enveloppe où elle interagit avec la protéine S, et présente au niveau de la nucléocapside où elle interagit avec la protéine N. La protéine M’ serait une protéine M modifiée. Le gène de la réplicase code pour une protéine présomptive de 740 à 800 kDa qui présente des homologies de séquence avec diverses protéines (protéases, ARN polymérase dépendante de l’ARN, facteur de croissance et protéine à doigt de zinc) qui sont produites par coupure protéolytique du produit de traduction primaire. SARS-CoV Deux équipes, l’une américaine [
2] et l’autre canadienne [
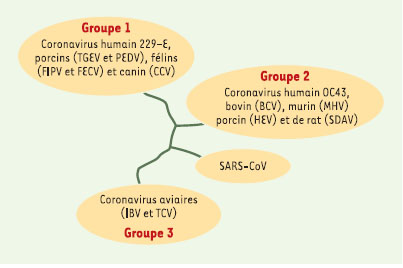
3], ont réalisé le séquençage complet du génome des coronavirus isolés à partir des prélèvements réalisés sur des patients atteints de SRAS. Les deux séquences ne varient que par une dizaine de bases sur 29 000. La séquence confirme l’appartenance du virus au groupe des coronavirus, mais diffère de celle des deux coronavirus humains connus. On y retrouve les gènes codant pour les protéines de structure, pour la réplicase et une dizaine de cadres de lecture codant pour des protéines qui ne présentent aucune homologie de séquences avec des protéines déjà caractérisées. Le gène de la protéine de surface HE est absent du génome du SARS-CoV. La comparaison de séquence avec les réplicases et les protéines N, M, S et E des coronavirus déjà connus produit des pourcentages d’identité qui varient de 20 à 50 % et place le SARS-CoV dans un nouveau groupe qui ne semble pas avoir évolué à partir d’un coronavirus déjà connu (Figure 3). 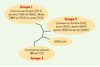 | Figure 3.
Arbre phylogénétique simplifié des différents coronavirus connus. Les coronavirus sont subdivisés en trois groupes sur la base des propriétés antigéniques et des homologies de séquences. Le SARS-CoV n’appartient à aucun des groupes déjà identifiés (d’après [ 2, 3, 5]). TGEV, transmissible gastroentritis virus ; PEDV, porcine epidemic diarrhea virus ; FIPV, feline infectious peritonitis virus ; FECV, feline enteric coronavirus ; CCV, canine coronavirus ; BCV, bovine coronavirus, MHV, murine hepatitis virus ; HEV, hemagglutinating encephalomyelitis virus ; SDAV, sialodacryoadenitis virus ; SARS-CoV, severe acute respiratory syndrome coronavirus ; IBV, infectious bronchitis virus ; TCV, turkey coronavirus
|
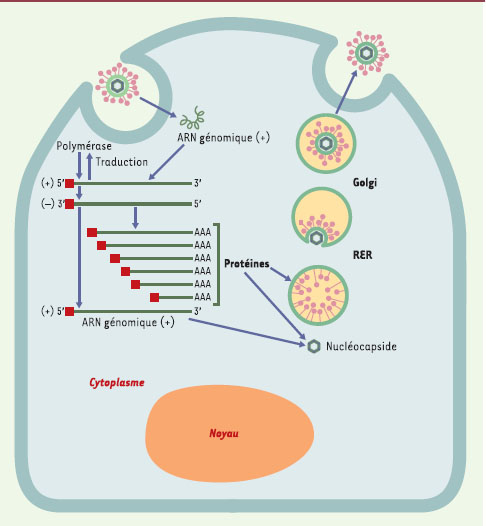
Cycle infectieux Coronavirus connus Le virus se réplique dans le cytoplasme des cellules infectées (Figure 4). Le génome viral pénètre dans le cytoplasme par endocytose et/ou fusion membranaire. À partir de l’ARN génomique, une polymérase est traduite qui synthétise, par un mécanisme encore mal connu, un brin d’ARN de polarité négative. Celui-ci servira de matrice pour la production d’ARN messagers codant pour les différentes protéines de capside, et d’ARN génomiques qui seront ensuite encapsidés. Les particules virales sont transportées et relarguées à la surface des cellules via l’appareil de Golgi. 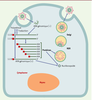 | Figure 4.
Cycle infectieux d’un coronavirus. L’ensemble du processus de réplication virale a lieu dans le cytoplasme. Après interaction des protéines S avec des récepteurs cellulaires et fusion membranaire, l’ARN génomique viral introduit dans le cytoplasme donne naissance, par traduction, à la réplicase qui porte l’activité polymérase. La polymérase permet la production d’ARN génomique et d’ARNm codant pour les différentes protéines structurales du coronavirus. L’ARN génomique s’associe à la protéine N pour former la nucléocapside. Les particules virales sont assemblées et excrétées de la cellule via le réticulum endoplasmique rugueux (RER) et dans l’appareil de Golgi. La séquence 5’-leader à l’extrémité 5’ des ARN est représentée en rouge. AAA: polyadénylation |
SARS-CoV La protéine de nucléocapside N du SARS-CoV contient une séquence riche en acides aminés basiques qui est absente de tous les autres coronavirus connus et qui pourrait être un signal de translocation nucléaire. Il est possible que cette protéine ait acquis une nouvelle fonction nucléaire qui pourrait expliquer le pouvoir pathogène de ce virus [ 3]. Hôtes et maladies Coronavirus connus Virus à tropisme multiple (respiratoire, entérique, neurologique et hépatique), les coronavirus infectent les vertébrés, oiseaux et mammifères. Chez l’homme, ils seraient responsables de 10 à 30 % des rhumes (en deuxième position après les rhinovirus). Ils provoquent des infections touchant tout l’arbre respiratoire [
6] et ont été incriminés, chez l’enfant, dans des entérocolites nécrosantes. Des coronavirus induisent des pathologies chez le chat (péritonite infectieuse féline), chez le chien et le porc (gastro-entérite) et chez les oiseaux et volailles (affections respiratoires). L’ensemble des coronavirus connus est classé en trois groupes sur la base des propriétés antigéniques (Figure3). Les deux coronavirus humains prototypes, 229 E et OC43, appartiennent à deux groupes différents (1 pour 229 E et 2 pour OC43). SARS-CoV Pour identifier le virus responsable du SRAS, les tissus et sécrétions biologiques de personnes atteintes de la maladie ont été soumis à un dépistage immunohistochimique pour la présence de micro-organismes pathogènes bactériens, viraux et fongiques connus [
7–
9]. Des résultats négatifs ont été obtenus dans tous les cas, y compris avec les anticorps dirigés contre les coronavirus des groupes 1, 2 et 3. L’ensemble des analyses immuno-histochimiques et génomiques converge donc pour placer le SARS-CoV dans un nouveau groupe de coronavirus indépendant des trois groupes déjà caractérisés (Figure 3). Récepteurs cellulaires Coronavirus connus Des récepteurs cellulaires responsables de l’interaction avec la protéine S des coronavirus ont été identifiés pour plusieurs souches de coronavirus. Les virus du groupe 1 (dont l’HCoV 229 E) utilisent l’aminopeptidase N comme récepteur, une métalloprotéase présente à la surface des cellules épithéliales de l’intestin, du poumon et du rein [
10]. D’autres coronavirus, dont l’HCoV OC43, interagissent avec les résidus de type acide sialique sur les glycoprotéines cellulaires via la protéine de surface HE, présente chez les virus du groupe 2 [
11]. Des données récentes obtenues in vitro sur un coronavirus à tropisme gastro-intestinal suggèrent que ces deux types d’interactions pourraient être nécessaires à l’infection des cellules de l’épithélium intestinal in vivo [
12]. Le complexe majeur d’histocompatibilité de classe I a également été décrit comme récepteur de l’HCoV OC43 [
13]. SARS-CoV La faible homologie entre les protéines S du SARS-CoV et des autres coronavirus connus (20 à 27%) ne permet pas d’émettre d’hypothèses quant au type de récepteur cellulaire impliqué dans l’infection par ce nouveau coronavirus. On note cependant l’absence du gène codant pour la protéine HE chez le SARS-CoV. |
Une exceptionnelle capacité de mutation et de recombinaison Coronavirus connus Les coronavirus, comme de nombreux autres virus à ARN, présentent une variabilité génétique importante. Elle est à la fois la conséquence de l’absence d’activité de correction d’erreur de l’ARN polymérase responsable de la réplication de leur matériel génétique et d’une fréquence élevée de recombinaison. Dans le cas du coronavirus murin MHV ( mouse hepatitis virus), la recombinaison peut survenir avec une fréquence de 1,3 % pour 1300 nucléotides, et atteindre ainsi 25% pour l’ensemble du génome [
14]. Des événements de recombinaison ont également été observés lors de la co-infection d’oeufs fécondés avec deux souches de coronavirus aviaires [
15]. Cette variabilité génétique a été dans le passé la cause d’un changement de tropisme d’une souche de coronavirus porcin [
16]. In vitro, la seule modification d’un acide aminé dans la région aminoterminale de la protéine S a permis de modifier le tropisme d’une souche de coronavirus porcin qui, de gastroentérique et respiratoire, est devenu exclusivement respiratoire [
17]. SARS-CoV La séquence des protéines prédite à partir du séquençage complet du génome du SARS-CoV n’est pas en faveur de son émergence par mutation ou recombinaison de coronavirus connus. Les pourcentages d’homologies caractérisés sont faibles et s’appliquent à des domaines dispersés au sein des protéines. Les données actuellement disponibles suggèrent que le SARS-CoV pourrait être issu soit d’un coronavirus humain non pathogène, et de ce fait jamais identifié, soit du franchissement de barrière d’espèce par un virus non caractérisé. |
Diagnostic et traitements Diagnostic Dès avril 2003, l’hypothèse qu’un coronavirus serait l’agent infectieux responsable du SRAS était proposé par trois équipes différentes ayant utilisé des approches expérimentales similaires [ 7– 9,
18]. Après infection de cellules en culture par des prélèvements issus de cas avérés de SRAS, l’observation en microscopie électronique des cellules infectées suggérait la présence d’un coronavirus. L’amplification par RT-PCR aléatoire, puis le séquençage d’un fragment d’ADN spécifique des cellules infectées a confirmé l’appartenance du virus isolé au groupe des coronavirus. L’ARN de ce virus est présent dans les différents types de prélèvements analysés (sécrétions respiratoires, urine, selles et biopsies) pour la plupart des patients étudiés. On observe également la présence d’anticorps contre ce nouveau virus chez les patients infectés. La disponibilité de la séquence complète du virus va permettre d’affiner les analyses par RT-PCR qui sont pour l’instant les seuls outils diagnostiques fiables et rapides, dans l’attente du développement éventuel d’anticorps sensibles et spécifiques. Mode de transmission Chez le chat, le chien et le porc, la transmission de la maladie est principalement de type oro-fécal. La maladie à tropisme pulmonaire est transmise par inhalation d’aérosol, par contact direct avec une personne infectée ou indirectement par des objets contaminés. Une étude réalisée au sein du personnel soignant du Queen Mary Hospital de Hong Kong indique que le port du masque est l’élément le plus déterminant dans la protection contre une infection par le SRAS et suggère donc que l’inhalation d’aérosol serait le mode de transmission majeur de la maladie [
19]. Traitements utilisés Les traitements établis empiriquement [
20] sont les traitements généralement utilisés pour les pneumopathies: antiviraux et antibiotiques à large spectre accompagnés de corticostéroïdes pour réduire la réaction inflammatoire qui peut être dévastatrice lors de charges virales importantes. L’efficacité de ces traitements n’est pour l’instant pas démontrée. En particulier, l’utilisation comme antiviral de la ribavirine, un nucléoside analogue de la guanosine, est très controversée au vu de ses effets secondaires potentiels (anémie hémolytique, effet tératogène), alors que les premiers essais in vitro n’indiquent pas d’efficacité de cette molécule ni sur la réplication du virus, ni sur sa propagation d’une cellule à l’autre. Perspectives thérapeutiques Il existe des vaccins contre les coronavirus pour les animaux de compagnie ou d’élevage, mais aucun chez l’homme, la pathologie ayant été jusqu’à ce jour bénigne. Ces vaccins sont de type virus inactivé. Des essais thérapeutiques en cours - rapportés par la presse - utiliseraient le sérum de patients guéris de la pneumopathie atypique pour traiter des malades résistants à l’association antiviraux/ corticostéroïdes [
21]. Les premiers résultats seraient encourageants (70% des 70 patients traités auraient quitté l’hôpital), mais des cohortes plus importantes devront être traitées avant de substituer ce traitement au protocole en cours. Par ailleurs, il existe des inhibiteurs puissants et spécifiques de l’interaction avec l’aminopepidase N, récepteur cellulaire de certains coronavirus, dont l’action sur les cellules infectées par le SARS-CoV pourra être testée. D’autres inhibiteurs de l’infection par les coronavirus ont pour cible la principale protéase virale (3CLpro) qui contrôle le clivage du produit de traduction du gène de la réplicase. Les premières modélisations structurales réalisées sur la protéase du SARS-CoV indiquent une importante conservation de la structure du site de fixation du substrat et permettent d’espérer que les inhibiteurs existants pourront être utilisés dans la lutte contre le SRAS [
22]. |
Moins d’un an s’est écoulé depuis l’apparition de cette nouvelle maladie dans la province du Guangdong au sud de la Chine. Durant cette courte période, au moins 8000 personnes ont été atteintes par la maladie dans 32pays différents et plus de 700 en sont mortes. Dans le même temps, l’agent infectieux principal a été identifié et son génome séquencé. L’épidémie semble aujourd’hui circonscrite dans la plupart des foyers majeurs. La situation reste cependant préoccupante, en particulier concernant une possible expansion aux pays en voie de développement, incapables d’assurer les mesures sanitaires propres à enrayer l’épidémie, comme c’est peut-être déjà le cas dans certaines provinces chinoises. De nombreuses questions restent posées: le coronavirus est-il le seul agent pathogène impliqué ou la gravité des symptômes dépend-elle de l’association avec d’autres agents infectieux (paramyxovirus ou bactérie de type Chlamydiae) qui ont été identifiés chez une proportion significative de malades atteints du SRAS ? Quels sont les modes précis de transmission ? Et surtout quelle est l’origine de ce nouveau coronavirus pathogène pour l’homme ? Au-delà de cette dernière interrogation, force est de constater qu’il faut s’attendre à l’avenir à l’émergence de nouvelles maladies infectieuses que les moyens modernes de communication et les mouvements des populations pourront propager rapidement à l’ensemble de la planète. La «mondialisation » devra aussi être sanitaire et médicale.

|
Je remercie vivement le Dr Astrid Vabret pour ses commentaires et suggestions.
|
1. Vabret A, Brouard J, Petitjean J, Eugene-Ruellan G, Freymuth F. Infections à coronavirus humains. Importance et diagnostic. Presse Med 1998; 27: 1813–7. 2. Rota PA, Oberste MS, Monroe SS, et al. Characterization of a novel coronavirus associated with severe acute respiratory syndrome. Science 2003; 300: 1394–9. 3. Marra MA, Jones SJ, Astell CR, et al. The genome sequence of the SRASassociated coronavirus. Science 2003; 300: 1399–404. 4. Risco C, Anton IM, Enjuanes L, Carrascosa JL. The transmissible gastroenteritis coronavirus contains a spherical core shell consisting of M and N proteins. J Virol 1996; 70: 4773–7. 5. Enjuanes L, Sola I, Almazan F, et al. Coronavirus derived expression systems. J Biotechnol 2001; 88: 183–204. 6. Vabret A, Mourez T, Gouarin S, Petitjean J, Freymuth F. An outbreak of coronavirus OC43 respiratory infection in Normandy, France. Clin Infect Dis 2003; 36: 985–9. 7. Poutanen SM, Low DE, Henry B, et al. Identification of severe acute respiratory syndrome in Canada. N Engl J Med 2003; 348: 1995–2005. 8. Drosten C, Gunther S, Preiser W, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med 2003; 348: 1967–76. 9. Ksiazek TG, Erdman D, Goldsmith CS, et al. A novel coronavirus associated with severe acute respiratory syndrome. N Engl J Med 2003; 348: 1953–66. 10. Yeager CL, Ashmun RA, Williams RK, et al. Human aminopeptidase N is a receptor for human coronavirus 229E. Nature 1992; 357: 420–2. 11. Vlasak R, Luytjes W, Spaan W, Palese P. Human and bovine coronaviruses recognize sialic acidcontaining receptors similar to those of influenza C viruses. Proc Natl Acad Sci USA 1988; 85: 4526–9. 12. Schwegmann-Wessels C, Zimmer G, Laude H, Enjuanes L, Herrler G. Binding of transmissible gastroenteritis coronavirus to cell surface sialoglycoproteins. J Virol 2002; 76: 6037–43. 13. Collins AR. Human coronavirus OC43 interacts with major histocompatibility complex class I molecules at the cell surface to establish infection. Immunol Invest 1994; 4-5: 313–21. 14. Baric RS, Fu K, Schaad MC, Stohlman SA. Establishing a genetic recombination map for murine coronavirus strain A59 complementation groups. Virology 1990; 177: 646–56. 15. Kottier SA, Cavanagh D, Britton P. Experimental evidence of recombination in coronavirus infectious bronchitis virus. Virology 1995; 213: 569–80. 16. Enjuanes L, Sanchez C, Gebauer F, Mendez A, Dopazo J, Ballesteros ML. Evolution and tropism of transmissible gastroenteritis coronavirus. Adv Exp Med Biol 1993; 342: 35–42. 17. Ballesteros ML, Sanchez CM, Enjuanes L. Two amino acid changes at the Nterminus of transmissible gastroenteritis coronavirus spike protein result in the loss of enteric tropism. Virology 1997; 227: 378–88. 18. Peiris JS, Lai ST, Poon LL, et al. Coronavirus as a possible cause of severe acute respiratory syndrome. Lancet 2003; 361: 1319–25. 19. Seto WH, Tsang D, Yung R, et al. Effectiveness of precautions against droplets and contact in prevention of nosocomial transmission of severe acute respiratory syndrome (SRAS). Lancet 2003; 361: 1519–20. 20. So LK, Lou AC, Yam LY, et al. Development of a standard treatment protocol for severe acute respiratory syndrome Lancet 2003; 361: 1615–7. 21. Crainte d’un désastre sanitaire dans les campagnes chinoises. Le Monde, 7 mai 2003. 22. Anand K, Ziebuhr J, Wadhwani P, Mesters JR, Hilgenfeld R. Coronavirus main proteinase (3CLpro) structure: Basis for design of anti-SRAS drugs. Science 2003 publié online le 13 mai 2003; 300: 1763–7. |