| |
| Med Sci (Paris). 2002 January; 18(1): 70–78. Published online 2002 January 15. doi: 10.1051/medsci/200218170.HIF-1 : régulateur central de l’hypoxie Emmanuel Gothié and Jacques Pouysségur* E. Gothié, J. Pouysségur : Institut de Recherches Signalisation, Biologie du développement et Cancer , Cnrs UMR 6543, IFR 72, Centre A. Lacassagne, 33, avenue de Valombrose, 06189 Nice, France |
L’homme, comme la plupart des espèces animales, présente une dépendance totale vis-à-vis de l’oxygène (dioxygène) pour sa survie. Sans un apport adéquat en oxygène, l’organisme est donc condamné à mourir extrêmement rapidement. Durant l’embryogenèse, les organismes supérieurs développent des systèmes respiratoire et circulatoire complexes pour assurer la disponibilité de l’oxygène à toutes les cellules de l’organisme. Une diminution en oxygène (hypoxie) va donc entraîner une réponse physiologique générale de l’organisme pour compenser ce manque. Ainsi, chez l’animal, une réponse rapide a lieu à la suite de la détection du taux d’oxygène sanguin au niveau de structures spécialisées situées dans la crosse aortique (les barorécepteurs). Un signal dopaminergique est envoyé au cerveau qui émet à son tour des signaux augmentant la respiration et le rythme cardiaque. Il en résulte une augmentation de la pression sanguine et de la saturation en oxygène nécessaires au métabolisme tissulaire. Dans le cas d’une période prolongée en condition d’hypoxie (exemple de personnes effectuant un séjour en altitude), des modifications telles qu’une augmentation du métabolisme anaérobie - ou glycolyse (avec pour conséquence la sécrétion d’acide lactique dans les muscles) - et du nombre de globules rouges sanguins, permettent de compenser le manque d’oxygène et de fournir une énergie suffisante aux processus cellulaires [1–4]. Ces réponses de l’organisme sont bien connues sur le plan physiologique, mais aussi sur le plan moléculaire grâce aux efforts de nombreuses équipes de recherche, principalement au cours des deux dernières décennies. L’objectif de cette revue est de faire le point sur le régulateur clé de cette réponse cellulaire à l’hypoxie, le facteur induit par l’hypoxie-1 (HIF-1 pour hypoxia inducible factor-1), et de présenter les perspectives de recherches futures pour décrypter cette voie complexe. |
Mise en évidence de HIF-1 L’hypoxie a été montrée comme étant capable de stimuler l’expression de l’érythropoïétine (EPO), une hormone glycoprotéique synthétisée principalement au niveau du rein. Cette hormone est acheminée par voie sanguine jusqu’à la moelle osseuse pour y stimuler les précurseurs des globules rouges qui vont proliférer et se différencier en érythrocytes, augmentant le nombre de globules rouges sanguins et donc le potentiel de captage en oxygène du sang. L’étude du promoteur de l’EPO a permis de mettre en évidence une séquence enhancer en position 3’ non codante du gène (5’-TACGTGCT-3’) qui est sensible à l’hypoxie et qui a été nommée HRE (hypoxia response element). Cette séquence fixe en condition d’hypoxie un complexe protéique nommé HIF-1 [5]. La découverte de cette activité HIF-1 a permis le clonage des deux ADN complémentaires impliqués dans cette activité [6, 7] qui codent pour les deux protéines du complexe HIF-1, HIF-1α et HIF-1ß. HIF-1ß a été identifié comme étant le facteur ARNT1 (aryl hydrocarbon receptor nuclear translocator), une protéine déjà décrite et impliquée dans les phénomènes de détoxification des cellules. En revanche, HIF-1α est spécifique de la réponse hypoxique. Les gènes codant pour HIF-1α et ARNT1 ont été clonés chez de nombreuses espèces (souris, rat, xénope, drosophile…) et leur séquence protéique est très conservée (90 % d’homologie entre l’homme, le rat et la souris). Le gène hif-1α, localisé sur le chromosome 14 (14q21-q24), est constitué de 15 exons chez l’homme. Des isoformes issues d’épissages alternatifs ont été mises en évidence pour HIF-1α d’abord chez la souris et le rat [8, 9] puis récemment chez l’homme [10]. Ces phénomènes d’épissage ne sont pas tous conservés entre les espèces et leurs rôles physiologiques restent à déterminer. Le gène hif-1β/arnt1 est localisé sur le chromosome 1 humain (1q21). Des phénomènes d’épissage alternatif sont aussi décrits pour arnt1, ainsi le gène humain présente un exon alternatif retrouvé aussi chez le rat. Un autre épissage est également décrit chez la truite. Chez l’homme, les transcrits de hif-1α sont fortement exprimés dans tous les organes de façon constitutive [11]. Cette expression ubiquiste a été confirmée chez la souris [9,12]. Les transcrits du gène arnt1 sont exprimés de façon ubiquiste et constitutive chez la souris [13], une distribution semblable étant globalement retrouvée chez le rat [14]. |
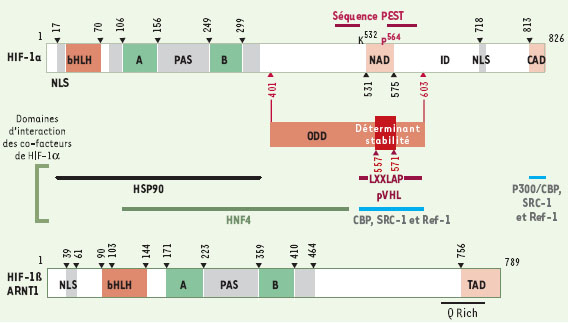
HIF-1 est un hétérodimère constitué des deux sous-unités HIF-1α et ARNT1/HIF-1ß (Figure 1). HIF-1α et ARNT1 humains possèdent respectivement 826 et 789 acides aminés. Ils contiennent des domaines bHLH (basic-helix loop helix) et PAS (PER-ARNT-SIM) à leur extrémité amino-terminale et font donc partie d’une superfamille de protéines contenant ces domaines (AHR, aryl hydrocarbon receptor, SIM, single-minded, PER, period, CYC, cycle…). Le motif HLH intervient dans la dimérisation des protéines tandis que la région basique qui le précède intervient dans la fixation et la spécificité de la liaison de la protéine à l’ADN. PAS est le sigle provenant des noms des protéines dans lesquelles des séquences répétées imparfaites ont été découvertes initialement (protéines de drosophile, PER et SIM, et la protéine des vertébrés ARNT). HIF-1α contient d’autres domaines fonctionnels importants pour sa fonction de régulateur de la transcription de gènes cibles par l’hypoxie. Il s’agit tout d’abord de ses deux domaines de transactivation situés dans la partie carboxy-terminale. Étudiés par plusieurs équipes, leur structure et leur régulation fine sont bien connues [15–17]. Le premier domaine de transactivation (TAD-N ou NAD) correspond aux acides aminés 531-575. Le second TAD (TAD-C ou CAD), en position carboxy-terminale dans la protéine, correspond aux acides aminés 813-826. Ils sont séparés par un domaine inhibiteur de la transcription [15]. La région précédant le domaine TAD-N, pourrait aussi présenter une activité d’inhibition [16]. ARNT1 contient aussi un domaine TAD dans sa partie carboxy-terminale, mais ce dernier n’est pas impliqué dans la réponse à l’hypoxie [16], ce qui souligne encore que HIF-1α est l’élément clé de la réponse à l’hypoxie. Un domaine responsable de la dégradation (oxygen-dependent degradation domain ou ODD) de HIF-1α en normoxie par le protéasome [18] et situé entre les acides aminés 401-603 a été mis en évidence [19] (voir Régulation de l’activité de HIF-1). Une étude détaillée de ce domaine a montré que les différentes sections (acides aminés 401-496, 497-529 et 530-603) étaient toutes capables de conférer des induc-tions par l’hypoxie à des degrés différents. Néanmoins, une analyse comparée des séquences des protéines de la famille de HIF-1α (notée HIF-α) et des expériences complémentaires ont souligné l’importance d’une séquence de 15 acides aminés (557-571) responsable de la stabilisation par l’hypoxie des protéines HIF-α [20] et retrouvée aussi dans la protéine homologue de HIF-1α chez la drosophile: Similar (Sima). Deux séquences PEST de 20 acides aminés (riches en proline, acide gluta-mique, sérine ou thréonine) ont été décrites pour HIF-1α, mais ne semblent pas être impliquées de façon claire dans l’instabilité de la protéine [19]. Elles sont localisées au niveau des acides aminés 499-518 et 581-600 [6] à la fin du domaine ODD. Enfin, deux séquences de localisation nucléaire ont été décrites. La première séquence possède une structure bipartite 17RRKEKSRDAARSRRSKE33 (similaire au NLS, nuclear localisation sequence, de la nucléoplasmine) et est localisée dans le domaine bHLH. Elle est réprimée par le domaine PAS-B, ce qui entraîne une rétention cytoplasmique de la protéine. La seconde séquence 718RKRK721 est apparentée au NLS retrouvé dans l’antigène grand-T de SV40. Cette séquence jouerait un rôle clé dans l’import nucléaire dépendant de l’hypoxie de HIF-1α [21]. Les domaines de fixation connus des co-fac-teurs de HIF-1, p300/CBP, SRC-1, Ref-1, HSP90 et pVHL, sont aussi indiqués sur la figure 1 (voir Régulation de l’activité de HIF-1).  | Figure 1. Structure des sous-unités humaines de HIF-1. Domaines fonctionnels des deux sous-unités de HIF-1 et, lorsqu’ils sont connus, domaines d’interaction des co-facteurs de HIF-1α. ODD : oxygen dépendent dégradation motif ; LXXLAP : motif conservé de C. elegans à l’homme, et reconnu par la HIF-proline hydroxylase ; pVHL : von Hippel Lindau protein : PAS : Per, Arnt, Sim motif ; NAD et CAD : domaines de transactivation N- et C-terminal ; NLS : séquences de localisation nucléaire. |
|
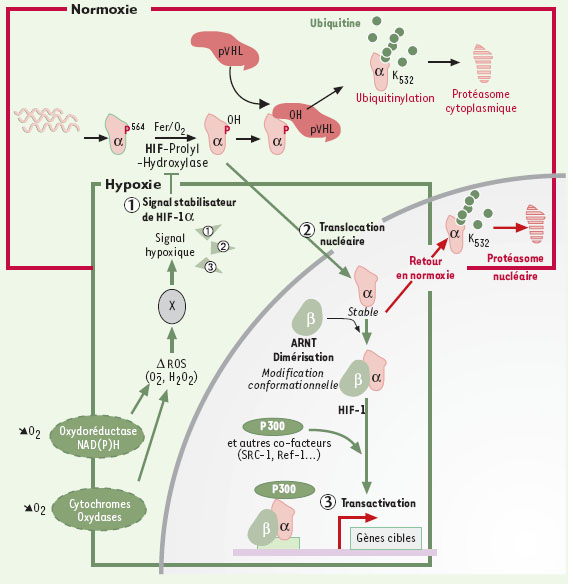
Régulation de l’activité de HIF-1 Induction hypoxique de HIF-1 Si les voies de signalisation en aval du facteur HIF-1 sont bien documentées, les mécanismes en amont par lesquels les variations d’oxygène sont détectées au niveau cellulaire sont demeurés très longtemps inconnus (Figure 2). Ainsi, plusieurs hypothèses sur la nature du senseur de l’oxygène étaient avancées sans pour autant que les données disponibles soient suffisantes pour valider un modèle plutôt qu’un autre. Néanmoins, les équipes de P.J. Ratcliffe et de W.G. Kaelin Jr viennent de publier des données remarquables sur le mécanisme d’activation de HIF-1 impliquant une activité hydroxylase dépendante du fer et de l’oxygène. L’enzyme correspondante - dénommée HIF-PH pour HIF-α prolylhydroxylase - serait le senseur de l’oxygène cellulaire [ 22, 23]. Tout récemment, trois HIF-PH conservées de C. elegans à l’homme viennent d’être identifiées [ 56– 57] *. D’autres hypothèses fondées sur l’intervention d’enzymes impliquées dans la formation de molécules oxygénées réactives (ROS pour reactive oxygen species) sont aussi avancées (voir Perspectives dans la détermination de la signalisation en amont de HIF-1). En ce qui concerne HIF-1, les éléments clés de sa régulation et de son activité sont désormais bien connus même si certaines zones d’ombre demandent encore à être éclaircies. Ainsi, les ARNm des deux sous-unités de HIF-1 sont exprimés de façon constitutive et stable dans la plupart des cellules [ 24]. Au niveau protéique, ARNT1/HIF-1ß est lui aussi stable. En revanche, HIF-1α est dégradé extrêmement rapidement en condition de normoxie (demi-vie de l’ordre de 5 min) [ 6] par le système ubi-quitine/protéasome [ 18, 19, 25, 26].  | Figure 2.Régulation de l’activité de HIF-1. En condition de normoxie, la protéine HIF-1α est très instable. Elle fixe pVHL, après une hydroxylation sur sa proline 564 dépendante de l’oxygène et du fer. La protéine pVHL fait partie d’un complexe (contenant les élongines B et C et la protéine CUL-2) qui possède une activité E3 ubiquitine ligase. HIF-1α est donc ubiquitinylé en normoxie puis dégradé par le protéasome. En condition d’hypoxie, HIF-1α n’est plus hydroxylé et ne fixe plus pVHL. Le signal hypoxique va aussi déclencher la translocation nucléaire de HIF-1a. Il peut dès lors se fixer à son partenaire HIF-1ß/ARNT1 ainsi qu’à d’autres co-facteurs tels que p300/CBP pour activer des gènes sous la dépendance d’éléments de réponse à l’hypoxie (HRE). Enfin, l’hypoxie permet d’augmenter directement la capacité transcriptionnelle de HIF-1 via ses domaines transactivateurs. Il est possible que d’autres senseurs (oxydoréductases, cytochromes) participent aussi à l’élaboration du signal hypoxique via les ROS. De retour en hypoxie, HIF-1α est rapidement dégradé, une étape pouvant être assurée par le protéasome nucléaire [ 55]. |
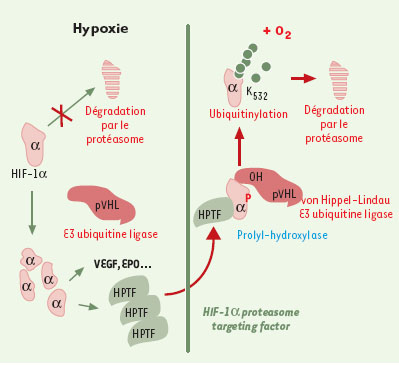
Le facteur suppresseur de tumeur pVHL (von Hippel-Lindau) - cible de mutations dans les cellules germinales donnant le syndrome de cancer héréditaire caractéristique de la maladie du même nom - est clairement impliqué dans ce mécanisme. Il forme avec les élongines B et C et la culline-2 (Cul-2), un complexe qui présente une activité E3 ubiquitine ligase dans des extraits cellulaires. Ce complexe présente aussi des similitudes de structure et de fonction avec les composants du complexe multiprotéique SCF (Skp1 - Cul-1 - F-box protein), qui cible les protéines régulatrices du cycle cellulaire au système de dégradation par l’ubiquitine (→) [27, 28]. pVHL se fixe via son domaine ß au domaine de transactivation N-TAD de HIF-1α contenant le déterminant de 15 acides aminés, conservé entres les éléments HIF-α [20, 27–29]. Dans cette région, le motif LXXLAP est extrêmement important pour l’interaction entre pVHL et le domaine N-TAD d’HIF-1α , une mutation de ce domaine abolissant totalement celle-ci. En fait, pVHL reconnaît une forme hydroxylée au niveau de la proline de ce domaine (Pro564), une modification apportée par HIF-PH. D’autres acides aminés comme les Leu562 et Tyr565, et les Ser551 et Thr552 sont aussi importants, mais sont sans doute impliqués pour faciliter l’hydroxylation de P564 (respectivement [22, 23] et [26]). Enfin, la lysine K532 du domaine N-TAD est clairement essentielle pour la dégradation d’HIF-1α [27]. pVHL dirige directement l’ubiquitinylation d’HIF-1α (et d’HIF-2oc) et la dégradation par le protéasome en normoxie qui s’ensuit [27]. L’hypoxie entraîne la stabilisation de cette sous-unité avec une diminution de l’ubiquitinylation de HIF-1α [25] - un phénomène spécifique de HIF-1α puisque l’hypoxie ne modifie pas l’ubiquitinylation de p53 [29] -permettant ainsi son accumulation et l’activation de la voie du HIF-1 consécutive. En fait, l’interaction pVHL/HIF-1α n’a lieu qu’en normoxie, tandis que l’hypoxie, le cobalt ou la DFO, empêchant la HIF-PH d’hydroxyler HIF-1α , inhibent cette interaction et stabilisent le facteur de transcription [22, 23]. (→) m/s 1999, n°8-9, p. 1008 et n° 11, p. 1259 HPTF (HIF-1α proteasome targeting factor) est aussi un facteur impliqué dans la dégradation de HIF-1α . Il fonctionne selon un mécanisme de régulation par rétro-contrôle de la dégradation d’HIF-1α ([30] et figure 3) analogue à celui rencontré pour le couple p53/MDM2. Dans ce dernier, p53 règle son propre niveau d’expression en induisant l’expression de son régulateur négatif MDM2. HPTF pourrait s’avérer être en réalité le senseur HIF-PH, une hypothèse que nous avons récemment proposée [58]*. De façon intéressante, il a été aussi récemment décrit que p53 peut lui aussi intervenir sur la dégradation de HIF-1α par le protéasome à la suite d’une ubiquitinylation dirigée par MDM2 [31]. Toutefois, cette notion demande à être confirmée.
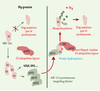 | Figure 3.Boucle de régulation de HIF- lα par HPTF (HIF-lα proteasome targe-ting factor). En condition d’hypoxie, le HIF-1a s’accumule et le complexe HIF-1 entraîne l’expression des gènes cibles spécifiques comme le gène codant pour le VEGF, l’EPO, mais aussi HPTF. Ce facteur s’accumule en hypoxie et participe avec pVHL à la dégradation rapide du HIF-1α lors du retour en normoxie [ 30]. HPTF accumulé sous forme “non-activé” en situation d’hypoxie serait une forme nductible de HIF-proline hydroxylase [ 56– 58]. |
Une fois stabilisé, HIF-1α est transporté dans le noyau. Cette étape est dépendante de son signal de localisation nucléaire sensible à l’hypoxie, situé en position carboxy-terminale. Il peut se dimériser dans ce compartiment avec HIF-1ß qui est pour sa part exclusivement nucléaire [32, 33]. L’hétérodimère se fixe alors sur les éléments HRE des gènes sensibles à l’hypoxie et induit la transactivation de ces derniers. L’hypoxie joue donc à plusieurs niveaux sur la sous-unité HIF-1α : en la stabilisant, en favorisant sa translocation nucléaire et, enfin, en augmentant l’activité transactivatrice de ses deux domaines de transactivation. Plusieurs co-facteurs peuvent intervenir en se fixant au complexe et en augmentant la transactivation. Il a ainsi été montré que p300/CBP est capable de se fixer au domaine C-TAD de HIF-1α et d’activer la transactivation [21]. TIF2 (pour transcription intermediary factor 2) et SRC-1 (pour steroid-receptor co-activators) sont aussi capables d’interagir avec HIF-1α d’une manière dépendante de l’hypoxie et d’augmenter le potentiel de transactivation inductible sous hypoxie. SRC-1 peut produire cet effet en synergie avec p300/CBP et la protéine de régulation redox Ref-1 est capable d’augmenter fortement cet effet, ce qui indique que ces trois protéines sont des composants importants de la voie de signalisation de l’hypoxie [34]. HIF-1α interagit encore avec d’autres co-facteurs tels que la protéine chaperon HSP90 [24]. HSP90 est nécessaire à l’activation de HIF-1 en hypoxie. Il permet notamment, en se liant à HIF-1α en normoxie, que celui-ci adopte une conformation activable par l’hypoxie, un rôle cohérent avec son rôle de protéine chaperon [35, 36]. L’interaction HSP90-HIF-1α semble liée aux conditions d’oxygénation, HSP90 se dissociant de HIF-1α en situation d’hypoxie [35]. Par ailleurs, la modification de conformation de HIF-1α par HIF-1ß dans le noyau n’a pas lieu en l’absence d’une conformation de HIF-1α dirigée par HSP90 [36]. La présence de partenaires cellulaires spécifiques éventuels peut donner lieu à une réponse à l’hypoxie spécifique de tissu. Ainsi, il est proposé que le récepteur nucléaire hépatique HNF4 participe avec HIF-1 au contrôle de l’expression de l’érythropoïétine [3, 37]. Autres régulations de HIF-1 : induction non hypoxique de HIF-1 / phosphorylation / isoformes Bien que l’hypoxie soit le facteur majoritaire d’induction de HIF-1α au niveau des cellules, d’autres stimulus, tels que l’insuline, l’IGF-1 et 2 (insulin-like growth factor), l’EGF (epidermal growth factor) ou le FGF (fibroblast growth factor), sont capables d’augmenter le niveau de ce facteur de transcription dans certains types cellulaires. Il a en outre été montré une activation consécutive de gènes contrôlés par des HRE tels que vegf, glut 1 et 3, alda, pgk. Dans des cellules musculaires lisses vasculaires (VSMC), HIF-1α est fortement augmenté en situation de normoxie par des agonistes de récepteurs membranaires (angiotensine II, thrombine et PDGF, platelet derived growth factor) et capable d’induire aussi la formation du VEGF [ 38]. Une voie indépendante de l’hypoxie est donc aussi capable d’induire la formation du complexe HIF-1 fonctionnel et ce par un mécanisme dépendant également de la stabilisation de HIF-1α . La corrélation entre l’induction de gènes impliqués dans le transport du glucose ou de la glycolyse par l’hypoxie mais aussi par l’insuline via les mêmes éléments (HIF-1α et HIF-1ß), laisse penser que ces derniers sont aussi essentiels pour l’activation de gènes nécessaires pour fournir à la cellule l’énergie requise dans des conditions de normoxie [39]. De façon intéressante, l’augmentation de HIF-1α dans es VSMC est dépendante de la production de ROS. Cette voie, pour l’instant spécifique de ces cellules, pourrait jouer un rôle clé dans la production du VEGF en normoxie et donc induire la perméabilité des vaisseaux en phase d’inflammation [38]. Des modifications par phosphorylation / déphosphory-ation ont été proposées comme essentielles dans la voie menant à l’activation de HIF-1 [4]. Notre laboratoire a récemment montré que HIF-1α était fortement phosphorylé in vivo. Cette phosphorylation entraîne des modifications importantes dans le profil de migration de la protéine confirmant les hypothèses de modifications post-traductionnelles déjà émises [6]. Les p42/p44 MAPK sont capables de phosphoryler HIF-1α in vivo, de reproduire ce profil électrophorétique et d’augmenter son activité transactivatrice [40]. L’existence d’autres membres de la famille bHLH/PAS pouvant fixer HIF-1ß (AHR, SIM…), d’isoformes de HIF-1α récemment découvertes (HIF-2α1,, HIF-3α2,) ou de formes résultant d’un épissage alternatif laisse supposer des phénomènes de compétition entre les différents éléments [24]. Par ailleurs, la présence d’autres partenaires potentiels pour HIF-1α (ARNT2 et ARNT33) laisse envisager d’autres hétérodimères fonctionnels pouvant également interférer avec la voie centrale de la réponse cellulaire dépendante de HIF-1. |
HIF-1, régulateur central de l’expression de gènes en hypoxie Décrit au départ comme le régulateur de l’expression du gène epo en hypoxie, HIF-1 s’est révélé être en réalité le régulateur central de l’expression de nombreux gènes sous hypoxie. Comme vu précédemment, HIF-1 est exprimé dans tous les types cellulaires examinés dont la plupart n’expriment pas d’érythropoïétine. Ainsi, HIF-1 ne semble pas restreint à cette seule activité. Cela a été confirmé par l’analyse de promoteurs de gènes connus pour être sensibles à l’hypoxie, notamment les enzymes de la voie glycolytique qui, pour la plupart, présentent des HRE dont l’activité a pu être vérifiée [41]. Ces enzymes permettent un basculement sur le métabolisme anaérobie et la production d’énergie (via la voie glycolytique) nécessaire aux cellules lorsque les apports en oxygène sont réduits. Des sites fonctionnels HRE ont aussi été trouvés dans de nombreux autres gènes parmi lesquels le facteur de croissance de l’endothélium vasculaire (VEGF pour vascular endothelial growth factor), qui entraîne une augmentation de l’angiogenèse rencontrée au cours du développement, de processus physiologiques ou physiopathologiques, la NO synthase inductible (i-NOS) et l’hème oxygénase-1 (HO-1) qui donnent lieu à la synthèse des vasodilatateurs monoxyde d’azote (NO) et monoxyde de carbone (CO) respectivement, la tyrosine hydroxylase (TH) qui est une enzyme clé dans la régulation de la respiration, et la transferrine qui comme l’EPO intervient dans l’érythropoïèse [3, 42]. L’ensemble de ces gènes permettent au niveau cellulaire, mais aussi à l’échelle de l’organisme, une réponse adaptative à l’hypoxie et HIF-1 est la clé de voûte de l’ensemble de ces réponses physiologiques [3]. Le tableau I présente l’intégralité des gènes actuellement connus réglés par le facteur HIF-1 (d’après [41]).  | Tableau 1 : Gènes réglés au niveau transcriptionnel par HIF-1. |
|
Implication de HIF-1 dans l’embryogenèse et la physiopathologie La démonstration de l’importance de HIF-1 dans l’homéostasie de l’oxygène a été clairement établie par l’invalidation des gènes (KO pour knock-out) codant pour les deux sousunités du complexe. En premier lieu, le KO de HIF-1ß [43] a permis de montrer l’importance du complexe HIF-1. En effet, l’invalidation du gène entraîne un retard du développement, des malformations au niveau du tube neural, de la vésicule vitelline, du placenta, qui conduisent à la mort embryonnaire au jour E 10,5 de la gestation. Dans un second temps, plusieurs équipes indépendantes [44–46] ont réalisé le KO de HIF-1α et ont ainsi pu démontrer un rôle global de HIF-1α au niveau du développement et de la physiologie vasculaire. En effet, des souris HIF-1(α−/− ne sont pas viables (mort embryonnaire au jour E 10,5 de la gestation) présentant une déficience au niveau de la vascularisation et des malformations cardiaques et neuronales [44]. Des similitudes avec le phénotype d’embryon VEGF+/- [47,48] montrent un lien clair entre l’expression du VEGF et celle de HIF-1α , nécessaire pour un développement harmonieux de la structure vasculaire. Les différentes isoformes d’HIF-1α ou d’HIF-1ß ne peuvent pas remplacer les sous-unités manquantes et compenser totalement leur fonction dans la réponse à l’hypoxie. HIF-1α et HIF-1ß ne sont donc pas des gènes redondants et l’hétérodimère HIF-1 exerce indiscutablement un rôle essentiel dans le développement embryonnaire. Les études réalisées sur des cellules ES HIF-1α−/− ont aussi pu démontrer le rôle central de ce facteur de transcription dans la régulation des gènes impliqués dans le métabolisme glycolytique [44, 45]. L’adaptation des cellules cancéreuses à l’hypoxie est une étape critique pour la progression tumorale. Des tumeurs induites chez la souris nude, par injection de cellules d’hépatome c4 déficientes en ARNT1, sont significativement moins abondantes que celles induites par les cellules sauvages Hepa1 et présentent une densité capillaire moindre corrélée à une absence de transcrits du VEGF [49]. En ce qui concerne HIF-1α , les tumeurs induites chez la souris nude par injection de cellules ES HIF-1α−/− sont faiblement vascularisées et moyennement hémorragiques contrairement à celles issues de cellules ES HIF-1α+/+. Le facteur de transcription HIF-1 joue donc un rôle clé dans le développement tumoral par son action angiogénique via le contrôle de l’expression du VEGF. |
Perspectives dans la détermination de la signalisation en amont de HIF-1 La découverte de l’implication de plusieurs HIF-proline hydroxylases est une avancée majeure dans les mécanismes “senseur” d’oxygène de la signalisation hypoxique. Les futures investigations devront révéler les étapes qui contrôlent l’activité de ces enzymes, leur inductibilité, notamment par l’hypoxie (voir HPTF et auto-contrôle), leur distribution et localisation subcellulaire. Par ailleurs, les phénomènes de phosphorylation sont essentiels dans la signalisation du HIF-1 : à quels niveaux interviennent-ils et quelles sont les kinases/phosphatases responsables de la régulation de l’activité de HIF-1. Existe-t-il aussi d’autres facteurs intermédiaires ? Enfin, si les effets du cobalt et de la DFO sont expliqués par leur action au niveau de la proline hydroxylase, plusieurs observations expérimentales concernant notamment les ROS, restent à éclaircir. Ces métabolites de l’oxygène pourraient effectivement constituer des seconds messagers impliqués dans la signalisation de l’hypoxie, et les enzymes les engendrant être d’autres senseurs d’oxygène cellulaires. Par exemple, une oxydase NAD(P)H et la superoxyde-dismutase peuvent respectivement produire des anions superoxyde (O-2) et de l’eau oxygénée (H2O2). Dans ce sens, une oxydoréductase NAD(P)H de type cytochrome b a été identifiée chez les mammifères et proposée comme senseur d’oxygène [50]. La diminution de la formation de ces espèces oxygénées réactives en hypoxie pourrait être le déclencheur de la signalisation d’HIF-1 [51]. Par ailleurs, les cytochromes oxydases - éléments de la chaîne mitochondriale de transport des électrons -pourraient elles aussi avoir un rôle dans cette signalisation. En effet, en condition d’hypoxie, la réduction de l’oxygène en eau est diminuée au profit de la formation d’O2-, un résultat inverse du modèle précédent. Néanmoins, diverses expériences ont donné des résultats en faveur de l’un ou l’autre des modèles, et restent ainsi insuffisantes pour trancher le débat [51]. Les limitations inhérentes aux outils pharmacologiques sont donc ici indiscutables et il s’avère nécessaire de développer des approches génétiques pour décortiquer entièrement cette voie de signalisation complexe. Une première approche consistant à exprimer dans des cellules des marqueurs de surface exprimés en hypoxie à l’aide de constructions sous contrôle d’éléments de réponses à l’hypoxie, a permis d’isoler plusieurs mutants exprimant faiblement ou pas le marqueur de surface. Un seul s’est révélé positif et correspondait à une mutation pour HIF-1α [52]. Dans l’étude menée, aucun autre élément de la voie de l’hypoxie n’a pu être découvert. Notre laboratoire développe actuellement une approche similaire, mais fondée cette fois sur une sélection négative. L’objectif est de faire exprimer dans des lignées cellulaires des constructions cytostatiques ou cytotoxiques sous la dépendance de HRE. Des clones cellulaires résistants devront présenter des mutations au niveau de la voie de signalisation d’HIF-1. Une étude de ces mutants devrait nous permettre de caractériser des éléments de régulation de la proline hydroxylase spécifique de HIF-α et éventuellement d’autres intermédiaires de la voie de l’hypoxie. Par ailleurs, les cellules tumorales situées au sein des tumeurs solides étant très souvent extrêmement résistantes à la radiothérapie, l’utilisation de tels vecteurs activables par l’hypoxie reste un outil thérapeutique potentiel. Une expérience similaire réalisée par Ruan et al. a notamment déjà permis de montrer que l’expression réglée par l’hypoxie d’une protéine toxique (protéine apoptotique Bax sous la dépendance de HRE) est possible dans des cellules tumorales de cerveau (U-87 MG et U-251 MG-NCI) et entraîne la mort par apoptose des cellules transfectées sous anoxie [53]. Le rôle essentiel du facteur HIF-1 au cours du développement, dans la physiologie ou au niveau pathologique, fait de la découverte des mécanismes clés de son activation un véritable challenge. Les découvertes à venir sur cette voie de signalisation permettront sans aucun doute de nouvelles approches thérapeutiques de maladies sévères comme le cancer, ou les ischémies cérébrales ou cardiaques. Allant dans ce sens, il a été récemment montré que le peptide apparenté à la cathéline PR39 (prolin and arginine-rich peptide) est capable d’induire fortement l’angiogenèse en inhibant la dégradation d’HIF-1α par le système ubiquitine/protéasome. Ce peptide et des analogues de celui-ci pourraient donc s’avérer être des agents efficaces pour l’induction de l’angiogenèse thérapeutique [54]. |
Nous remercions vivement Edurne Berra, Christiane Brahimi-Horn et David Busti pour la relecture du manuscrit et leurs suggestions. Les travaux provenant de notre laboratoire ont été soutenus par le Cnrs, l’Inserm, l’Association pour la Recherche contre le Cancer (ARC) et la Ligue Nationale pour la Recherche contre le Cancer (Équipe labellisée). |
Footnotes |
1. Goldberg MA, Schneider TJ. Similarities between the oxygen -sensing mechanisms regulating the expression of vascular endothelial growth factor and erythropoietin. J Biol Chem 1994; 269:4355–9. 2. Jiang BH, Rue E, Wang GL, Roe R, Semenza GL. Dimerization, DNA binding, and transactivation properties of Hypoxiainducible factor 1. J Biol Chem 1996; 271 : 17771–8. 3. Guillemin K, Krasnow MA. The hypoxic response: huffing and HIFing. Cell 1997; 89 : 9–12. 4. Semenza GL. Hypoxiainducible factor 1: master regulator of O2 homeostasis. Curr Opin Genet Dev 1998; 8: 588–94. 5. Semenza GL, Nejfelt MK, Chi SM, Antonarakis SE. Hypoxiainducible nuclear factors bind to an enhancer element located 3’ to the human erythro-poietin gene. Proc Natl Acad Sci USA 1991; 88 : 5680–4. 6. Wang GL, Jiang BH, Rue EA, Semenza G. L. Hypoxiainducible factor 1 is a basic-helixloop-helix-PAS heterodimer regulated by cellular O2 tension. Proc Natl Acad Sci USA 1995; 92 : 5510–4. 7. Wang GL, Semenza GL. Purification and characterization of Hypoxiainducible factor 1. J Biol Chem 1995; 270 : 1230–7. 8. Wenger RH, Rolfs A, Kvietikova I, Spielmann P, Zimmermann, DR, Gassmann M. The mouse gene for Hypoxiainducible factor-1alpha-genomic organization, expression and characterization of an alternative first exon and 5’ flanking sequence. Eur J Biochem 1997; 246 : 155–65. 9. Luo G, Gu YZ, Jain S, Chan WK, Carr KM, Hogenesch JB, Bradfield CA. Molecular characterization of the murine HIf-1 alpha locus. Gene Expr 1997; 6 : 287–99. 10. Gothie E, Richard DE, Berra E, Pages G, Pouyssegur J. Identification of alternative spliced variants of human hypoxia-inducible factor-1alpha. J Biol Chem 2000; 275 : 6922–7. 11. Wiener CM, Booth G, Semenza GL. In vivo expression of mRNAs encoding hypoxiainducible factor 1. Biochem Biophys Res Commun 1996; 225 : 485–8. 12. Wenger RH, Rolfs A, Marti HH, Guenet JL, Gassmann M. Nucleotide sequence, chromosomal assignment and mRNA expression of mouse hypoxia-inducible factor-1 alpha. Biochem Biophys Res Commun 1996 223 : 54–9. 13. Hirose K, Morita M, Ema M, et al. cDNA cloning and tissuespecific expression of a novel basic helixloop-helix/PAS factor (Arnt2) with close sequence similarity to the aryl hydrocarbon receptor nuclear translocator (Arnt). Mol Cell Biol 1996; 16 : 1706–13. 14. Drutel G, Kathmann M, Heron A, Schwartz JC, Arrang JM. Cloning and selective expression in brain and kidney of ARNT2 homologous to the Ah receptor nuclear translocator (ARNT). Biochem Biophys Res Commun 1996; 225 : 333–9. 15. Jiang BH, Zheng JZ, Leung SW, Roe R, Semenza G.L. Transactivation and inhibitory domains of hypoxiainducible factor 1alpha. Modulation of transcriptional activity by oxygen tension. J Biol Chem 1997; 272 : 19253–60. 16. Pugh CW, O’Rourke JF, Nagao M, Gleadle JM, Ratcliffe PJ. Activation of hypoxia-inducible factor-1; definition of regulatory domains within the alpha subunit. J Biol Chem 1997; 272 : 11205–14. 17. O’Rourke JF, Tian YM, Ratcliffe PJ, Pugh CW. Oxygen-regulated and transactivating domains in endothelial PAS protein 1: comparison with hypoxiainducible factor-1alpha. J Biol Chem 1999; 274 : 2060–71. 18. Salceda S, Caro J. hypoxia-inducible factor 1alpha (HIF-1alpha) protein is rapidly degraded by the ubiquitin-proteasome system under normoxic conditions. Its stabilization by hypoxia depends on redox-induced changes. J Biol Chem 1997; 272 : 22642–7. 19. Huang LE, Gu J, Schau M, Bunn HF. Regulation of hypoxia-inducible factor 1alpha is mediated by an O2-dependent degradation domain via the ubiquitinproteasome pathway. Proc Natl Acad Sci USA 1998; 95 : 7987–92. 20. Srinivas V, Zhang LP, Zhu XH, Caro J. Characterization of an oxygen/redox-dependent degradation domain of hypoxia-inducible factor alpha (HIF-alpha) proteins. Biochem Biophys Res Commun 1999; 260 : 557–61. 21. Kallio PJ, Okamoto K, O’Brien S, et al. Signal transduction in hypoxic cells: inducible nuclear translocation and recruitment of the CBP/p300 coactivator by the hypoxiainducible factor-1alpha. EMBO J 1998; 17 : 6573–86. 22. Jaakkola P, Mole DR, Tian Y, et al. Targeting of HIF-alpha to the von Hippel-Lindau ubiquitylation complex by O2-regulated Prolyl hydroxylation. Science 2001; 292 : 468–72. 23. Ivan M, Kondo K, Yang H, et al. HIF alpha targeted for VHL- mediated destruction by proline hydroxylation: Implications for O2 sensing. Science 2001; 292 : 464–8. 24. Gradin K, McGuire J, Wenger RH, et al. Functional interference between hypoxia and dioxin signal transduction pathways: competition for recruitment of the Arnt transcription factor. Mol Cell Biol 1996; 16 : 5221–31. 25. Kallio PJ, Wilson WJ, O’Brien S, Makino Y, Poellinger L. Regulation of the hypoxiainducible transcription factor 1aipha by the ubiquitin-pro-teasome pathway. J Biol Chem 1999; 274 : 6519–25. 26. Sutter CH, Laughner E, Semenza GL. Hypoxiainducible factor 1alpha protein expression is controlled by oxygen-regulated ubiquitination that is disrupted by deletions and missense mutations. Proc Natl Acad Sci USA 2000; 97 : 4748–53. 27. Tanimoto K, Makino Y, Pereira T, Poellinger L. Mechanism of regulation of the hypoxiainducible factor-1 alpha by the von Hippel-Lindau tumor suppressor protein. EMBO J 2000; 19 : 4298–309. 28. Cockman ME, Masson N, Mole DR, et al. Hypoxia inducible factor-alpha binding and ubiquitylation by the von Hippel-Lindau tumor suppressor protein. J Biol Chem 2000; 275 : 25733–41. 29. Ohh M, Park CW, Ivan M, et al, Ubiquitination of hypoxiainducible factor requires direct binding to the betadomain of the von Hippel-Lindau protein. Nat Cell Biol 2000; 2 : 423–7. 30. Berra E, Richard DE, Gothie E, Pouyssegur J. HIF-1-dependent transcriptional activity is required for oxygenmediated HIF-1alpha degradation. FEBS Lett 2001; 491 : 85–90. 31. Ravi R, Mookerjee B, Bhujwalla ZM, et al. Regulation of tumor angiogenesis by p53-induced degradation of hypoxia-inducible factor 1alpha. Genes Dev 2000; 14 : 34–44. 32. Pollenz RS, Sattler CA, Poland A. The aryl hydrocarbon receptor and aryl hydrocarbon receptor nuclear translocator protein show distinct subcellular localizations in Hepa 1e1c7 cells by immunofluorescence microscopy. Mol Pharmacol 1994; 45 : 428–38. 33. Eguchi H, Ikuta T, Tachibana T, Yoneda Y, Kawajiri K. A nuclear localization signal of human aryl hydrocarbon receptor nuclear translocator/hypoxia-inducible factor 1beta is a novel bipartite type recognized by the two components of nuclear poretargeting complex. J Biol Chem 1997; 272 : 17640–7. 34. Carrero P, Okamoto K, Coumailleau P, O’Brien S, Tanaka H, Poellinger L. Redox-regulated recruitment of the transcriptional coactivators CREB-binding protein and SRC-1 to hypoxia-inducible factor 1alpha. Mol Cell Biol 2000; 20 : 402–15. 35. Minet E, Mottet D, Michel G, et al. Hypoxia-induced activation of HIF-1: role of HIF-1aipha-Hsp90 interaction. FEBS Lett 1999; 460 : 251–6. 36. Kallio PJ, Pongratz I, Gradin K, McGuire J, Poellinger L. Activation of hypoxia-inducible factor 1alpha: posttranscriptional regulation and conformational change by recruitment of the Arnt transcription factor. Proc Hatl Acad Sci USA 1997; 94 : 5667–72. 37. Galson DL, Tsuchiya T, Tendler DS, et al. The orphan receptor hepatic nuclear factor 4 functions as a transcriptional activator for tissue-specific and hypoxia-specific erythropoietin gene expression and is antagonized by EAR3/COUP-TF1. Mol Cell Biol 1995; 15 : 2135–44. 38. Richard DE, Berra E, Pouyssegur J. Nonhypoxic pathway mediates the induction of hypoxia-inducible factor 1alpha in vascular smooth muscle cells. J Biol Chem 2000; 275 : 26765–71. 39. Zelzer E, Levy Y, Kahana C, Shilo BZ, Rubinstein M, Cohen B. Insulin induces transcription of target genes through the hypoxia-inducible factor HIF-1alpha/ARNT. EMBO J 1998; 17 : 5085–94. 40. Richard DE, Berra E, Gothie E, Roux D, Pouyssegur J. p42/p44 mitogenactivated protein kinases phosphorylate hypoxia-inducible factor 1alpha (HIF-1aipha) and enhance the transcriptional activity of HIF-l. J Biot Chem 1999; 274 : 32631–7. 41. Semenza GL. hypoxia-inducible factor 1: oxygen homeostasis and disease pathophysiology. Trends Mol Med 2002 (sous presse). 42. Bianchi L, Tacchini L, Cairo G. HIF-1-mediated activation of transferrin receptor gene transcription by iron chelation. Nucleic Acids Res 1999; 27 : 4223–7. 43. Maltepe E, Schmidt JV, Baunoch D, Bradfield CA, Simon MC. Abnormal angio-genesis and responses to glucose and oxygen deprivation in mice lacking the protein ARNT. Nature 1997; 386 : 403–7. 44. lyer NV, Kotch LE, Agani F, et al. Cellular and developmental control of O2 homeostasis by hypoxia-inducible factor 1 alpha. Genes Dev 1998; 12:149–62. 45. Carmeliet P, Dor Y, Herbert JM, et al. Role of HIF-1alpha in hypoxia-mediated apoptosis, cell proliferation and tumour angiogenesis. Nature 1998; 394 : 485–90. 46. Ryan HE, Lo J, Johnson RS. HIF-1 alpha is required for solid tumor formation and embryonic vascularization. EMBO J 1998; 17 : 3005–15. 47. Carmeliet P, Ferreira V, Breier G, et al. Abnormal blood vessel development and lethality in embryos lacking a single VEGF allele. Nature 1996; 380: 435–9. 48. Ferrara N, Carver-Moore K, Chen H, et al. Heterozygous embryonic lethality induced by targeted inactivation of the VEGF gene. Nature 1996; 380 : 439–42. 49. Maxwell PH, Dachs GU, Gleadle JM, et al. hypoxia-inducible factor-1 modulâtes gerne expression in solid tumors and influences both angiogenesis and tumor growth. Proc Natl Acad Sci USA 1997; 94 : 8104–9. 50. Zhu H, Qiu H, Yoon HW, Huang S, Bunn HF. Identification of a cytochrome b-type NAD(P)H oxidoreductase ubiquitously expressed in human cells. Proc Natl Acad Sci USA 1999 96; 14742–7. 51. Semenza GL. Perspectives on oxygen sensing. Cell 1999; 98 : 281–4. 52. Wood SM, Wiesener MS, Yeates KM, et al. Selection and analysis of a mutant cell line defective in the hypoxiainducible factor-1 alphasubunit (HIF-1alpha). Characterization of hif-1alpha-dependent and -independent hypoxiainducible gene expression. J Biol Chem 1998; 273 : 8360–8. 53. Ruan H, Wang J, Hu L, Lin CS, Lamborn KR, Deen DF. Killing of brain tumor cells by hypoxia-responsive element mediated expression of BAX. Neoplasia 1999; 1 : 431–7. 54. Li J, Post M, Volk R, et al. PR39, a peptide regulator of angiogenesis. Nat Med 2000; 6 : 49–55. 55. Berra E, Roux D, Richard DE, Pouysségur J. hypoxia-inducible factor -1alpha (HIF-1alpha) escapes O2-driven proteosomal degradation irrespectively of its subcellular localization. EMBO Rep 2001; 2 : 615–20. 56. Epstein AC, Gleadle JM, McNeill LA, et al. C. elegans EGL-9 and mammalian homologs define a family of dioxygenases that regulate HIF by prolyl hydroxylation. Cell 2001; 107 : 43–54. 57. Bruick RK, McKnight SL. A conserved family of prolyl-4-hydroxylases that modify HIF. Science 2001; 294 : 1337–40. 58. Brahimi-Horn C, Berra E, Pouysségur J. Hypoxia: the tumor’s gateway to progression along the angiogenic pathway. Trends Cell Biol 2001; 11 : S32–6. |