Les ARN ribosomiques (ARNr) constituent le cœur de l’échafaudage enzymatique du ribosome. Les gènes qui les codent sont donc investis d’une des fonctions les plus fondamentales qui soit associée à l’expression des gènes de ménage (housekeeping genes). Cependant, des observations récentes ont fait naître l’idée que ces gènes, longtemps négligés, pourraient également contrôler directement des aspects importants du comportement cellulaire.
Les ARNr 18S, 5.8S et 28S sont les produits de la transcription des gènes ribosomiques, qui représente entre 35 et 60 % de la transcription nucléaire totale dans une cellule eucaryote en phase de prolifération [1]. La transcription résiduelle correspond à la transcription des ARNm des protéines ribosomiques, de l’ARNr 5S et des petits ARN nucléaires requis pour la biogenèse des ribosomes. Une part importante de la transcription nucléaire, près de 80 % chez la levure et jusqu’à 50 % dans une cellule de mammifère, est donc dévolue à l’assemblage des composants de la machinerie de la traduction protéique. La transcription des gènes ribosomiques et l’assemblage des ribosomes ont lieu dans le nucléole. Si ce compartiment nucléaire est également le site de plusieurs autres fonctions - le cycle cellulaire, la sénescence et des aspects du transport sont sous le contrôle de facteurs qui y sont localisés [2] - la transcription des gènes ribosomiques semble néanmoins être au cœur des événements survenant dans le nucléole.
Il existe une relation entre la régulation de la transcription dans le ribosome et la taille du nucléole [3], et l’existence même de ce dernier dépend d’une machinerie transcriptionnelle fonctionnelle [4]. La régulation de la transcription des ARNr a donc vraisemblablement une influence sur un large éventail de fonctions cellulaires, qu’elles soient ou non associées à la biogenèse des ribosomes.
Jusqu’à tout récemment, on admettait que la régulation de la transcription des gènes ribosomiques n’était qu’un effet indirect de modifications du métabolisme cellulaire. Or, nos travaux ont montré que tel n’est pas le cas. En effet, nous avons observé que la stimulation de cellules de mammifères par l’EGF (epidermal growth factor) provoque une augmentation immédiate de la transcription des gènes ribosomiques. De plus, nos résultats confirment que cet effet résulte de la phosphorylation directe du facteur de transcription UBF (upstream binding factor) par la MAP-kinase (mitogen-activated protein kinase) ERK1/2 (extracellular signal-regulated kinase). Les facteurs de croissance activent rapidement ERK1 et 2 par l’intermédiaire d’une cascade de molécules de signalisation dont l’oncogène Raf et la kinase MEK1/2 (MAP-ERK kinase). Notre observation d’un effet direct et extrêmement rapide de l’activation de ERK sur l’expression des gènes ribosomiques a lieu de surprendre. On peut en déduire que ces gènes jouent probablement un rôle actif dans la détermination de la capacité de croissance et de prolifération des cellules et que la déréglement de leur expression pourrait constituer une étape nécessaire de la carcinogenèse.
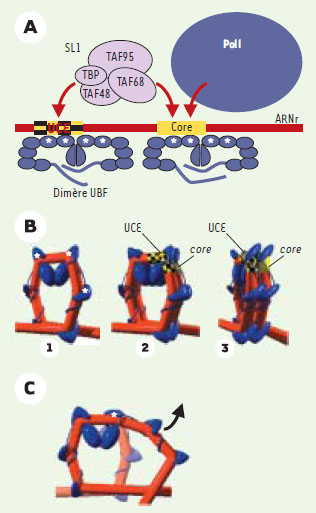
La plupart des eucaryotes possèdent plus d’une centaine de gènes chromosomiques codant pour l’ARNr, disposés sous forme d’une ou de plusieurs répétitions en tandem. Chez les mammifères, les promoteurs des gènes ribosomiques sont composés d’environ 150 paires de bases d’ADN comprenant deux motifs importants : l’élément de contrôle UCE (upstream control element) en amont, et l’élément promoteur basal (core) [1, 5]. Ces derniers peuvent être discernés de façon génétique mais ne montrent pas d’homologie de séquence entre des espèces d’ordres différents. Trois facteurs sont essentiels à la transcription des gènes ribosomiques : le facteur de remodelage de l’ADN UBF, à boîtes HMG1 (high mobility group), le complexe de sélectivité SL1 (selectivity factor 1) et l’ARN polymérase I (Figure 1A). Le complexe SL1 est lui-même composé de quatre polypeptides : TBP (TATA box-binding protein) et trois facteurs associés à TBP et spécifiques de l’ARN polymérase I (les TAF) [1]. Selon le modèle qui est actuellement proposé, UBF s’associerait d’abord au promoteur pour permettre le recrutement de SL1, soit par l’induction d’un changement de conformation de l’ADN, soit par le biais d’une interaction directe protéine-protéine, voire par une combinaison des deux événements. UBF, probablement sous forme de deux dimères, et SL1 interagissent à la fois avec les éléments UCE et core du promoteur (Figure 1A).
| Figure 1. Rôle d’UBF dans l’assemblage du complexe d’initiation de la polymérase I sur les gènes ribosomiques. A. La liaison de deux dimères d’UBF aux éléments UCE et core du promoteur favorise le recrutement de SL1 et de l’ARN polymérase I (PolI). Le complexe SL1 est lui-même formé de TBP (TATA box-binding protein) et de trois TAF (TBP-associated factors). Les sites de phosphorylation d’UBF sont représentés par des astérisques. B. La structure de l’enhancesome permet la juxtaposition des éléments UCE et core du promoteur des gènes ribosomiques. (1) L’enhancesome est formé par la liaison d’un dimère d’UBF à une séquence de 140 paires de base d’ADN. (2) et (3). L’interaction entre deux dimères adjacents d’UBF pourrait entraîner la formation de deux enhancesomes dans la région promotrice, représentée ici sous deux angles différents. C. La phosphorylation par la MAP-kinase d’une boîte HMG1 d’UBF est susceptible de provoquer une ouverture de la structure. Un enhancesome unique est schématisé à la fois lorsqu’une boîte HMG1 d’UBF est soit phosphorylée (rouge/bleu foncé), soit non phosphorylée (rouge/bleu pâle). |
Nos travaux antérieurs ont montré qu’un dimère d’UBF possède la capacité d’induire, au moyen de trois de ses boîtes HMG1, la formation d’une structure semblable au nucléosome dans laquelle 140 paires de bases d’ADN forment une boucle [6–9], et que nous avons appelé l’enhancesome (Figure 1B). Il est probable que deux de ces structures se forment au niveau du promoteur des gènes ribosomiques, de façon à juxtaposer ses deux éléments principaux, UCE et core, un modèle qui explique le recrutement coopératif de SL1 au niveau de ces séquences (Figure 1B). Il apparaît clairement que la formation de l’enhancesome doit être précédée du désassemblage de la structure nucléosomique associée aux gènes inactifs, les deux structures étant mutuellement exclusives. Par conséquent, UBF jouerait un rôle lors de l’activation génique, non seulement en favorisant la dissociation d’une structure chromatinienne répressive, mais aussi lors de la formation du complexe de pré-initiation en permettant le recrutement de SL1 et de l’ARN polymérase I. Dans ce contexte, il est toutefois surprenant de constater qu’in vivo, UBF se lie à l’ensemble des séquences des gènes ribosomiques, sans spécificité ou préférence apparentes [10]. L’enhancesome pourrait donc posséder une seconde fonction correspondant à l’assemblage d’une structure chromatnienne alternative, spécifique des gènes ribosomiques actifs.
L’interaction d’UBF avec l’ADN, et plus précisément avec le promoteur des gènes ribosomiques, est au cœur de sa fonction. Nous avons été d’autant plus surpris d’observer que la phosphorylation, par la MAP-kinase, des deux premières boîtes HMG1 d’UBF nécessaires à l’activation de la transcription, empêche leur liaison à l’ADN. De même, nous avons montré que si l’inhibition de la phosphorylation de UBF, induite par la substitution des sites de phosphorylation par des alanines, n’affecte pas sa capacité de liaison à l’ADN, elle abolit néanmoins l’effet activateur d’UBF sur la transcription. Ces mutations abolissent également la stimulation de la transcription des gènes ribosomiques par la cascade MAP-kinase. Par ailleurs, il nous a été impossible de créer une forme suractivée d’UBF en mutant en glutamates ou en aspartates les acides aminés ciblés par ERK, moyen souvent utilisé pour simuler leur phosphorylation. Ainsi, nos résultats suggèrent que la cohabitation des formes phosphorylée et non phosphorylée d’UBF est nécessaire à la stimulation de l’expression des gènes ribosomiques par les facteurs de croissance. Le mécanisme de cette stimulation est probablement lié au rôle de remodelage d’UBF. La phosphorylation par la MAP-kinase des boîtes HMG1 d’UBF empêcherait leur interaction avec leurs cibles et, par conséquent, la courbure de l’ADN induite par la liaison de chacune des boîtes HMG1. Des cycles de phosphorylation-déphosphorylation d’UBF provoqueraient alors des ouver tures et des fermetures partielles dans l’ADN de l’enhancesome (Figure 1C). De tels changements dynamiques seraient requis pour permettre à la fois l’assemblage du complexe d’initiation, le dégagement de la polymérase du promoteur ou son déplacement à travers la chromatine des gènes ribosomiques, voire la transition d’une chromatine nucléosomiques inactive à une chromatine « enhancesomique » active.
Quel que soit le mécanisme précis expliquant la régulation de l’activité des gènes codant pour les ARNr par les facteurs de croissance, la modulation rapide par ces derniers de l’expression de ces gènes est sans doute un processusclé dans la prolifération et le destin cellulaires. À cet égard, nos résultats récents doivent conduire à une ré-évaluation du rôle des gènes ribosomiques dans le développement, la différenciation cellulaire et le processus tumoral.