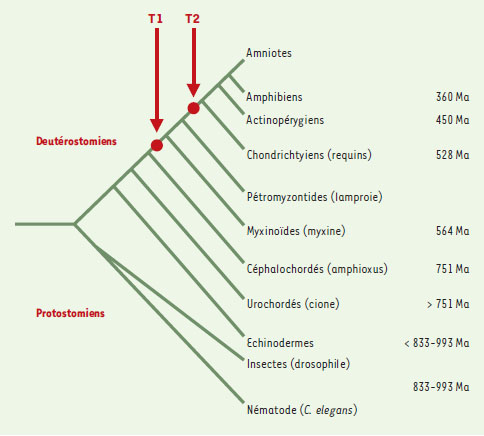
L’hypothèse selon laquelle le génome des vertébrés aurait évolué via plusieurs tours de polyploïdisations a déjà plus de trente ans [1]. Le génome des actuels vertébrés à mâchoires aurait dû conserver la trace de ces phénomènes sous la forme d’un réseau de régions paralogues (voir glossaire) au sein desquelles toutes les paires de gènes dupliqués auraient subi cet événement simultanément. L’analyse de la carte du génome humain [2] a permis d’identifier différentes régions de paralogie de ce type parmi lesquelles celle qui contient les gènes codant pour les éléments du complexe majeur d’histocompatibilité (CMH) [3] pour lequel le processus de duplication s’est vraisemblablement produit il y a au moins 420 millions d’années [4]. Des gènes ancres à évolution plus lente ont été mis en évidence à proximité des gènes humains du CMH [4]. Afin de savoir si le génome de notre ancêtre a subi des duplications en bloc, nous avons choisi [5]: (1) d’analyser en détail les gènes dupliqués présents dans la série de paralogie du CMH (analyses phylogénétiques et cartes), (2) de vérifier l’hypothèse selon laquelle la duplication en bloc serait survenue après la radiation des vertébrés et (3) de cloner les gènes équivalents chez l’amphioxus (ou lancelet), choisi comme animal modèle en raison de sa position au sein de l’arbre évolutif des métazoaires (Figure 1) .
| Figure 1. Cladogramme montrant les relations phylogénétiques des principaux groupes actuels (les longueurs de branches ne sont pas proportionnelles au temps de divergence séparant deux lignées terminales de leur ancêtre commun). Les temps de divergence sont calculés à partir des données moléculaires (sous l’hypothèse de l’horloge moléculaire) et calibrés à partir de données fossiles) et doivent être interprétés comme une échelle relative. T1 et T2 représentent le premier et le deuxième tour de duplication à grande échelle. Ma: millions d’années. |
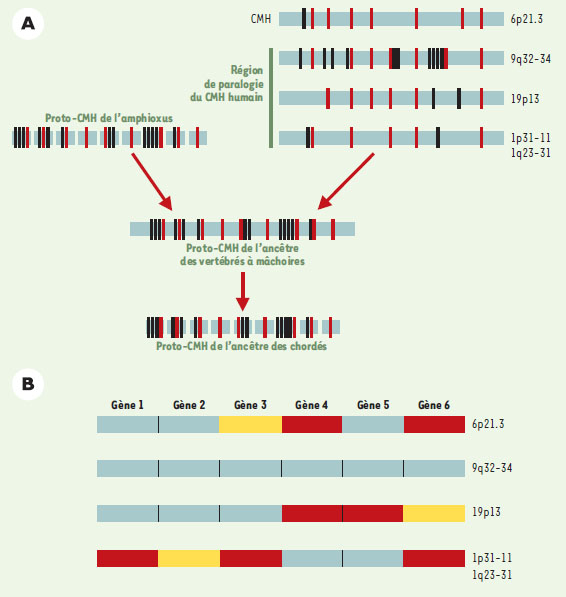
Parmi les 18 gènes ancres définis comme tels par une recherche dans les bases de données de nouveaux gènes paralogues et de leurs relations phylogénétiques, onze ont été sélectionnés pour cette étude. Un cosmide (voir glossaire) cloné a ensuite été entièrement séquencé et analysé pour chaque gène ancre afin d’étudier le contenu et les relations phylogénétiques des gènes de l’amphioxus et de leurs correspondants humains (Figure 2). Les gènes avoisinant chaque gène ancre sur les cosmides correspondants ont alors été prédits par analyse bio-informatique. Un total de 41 gènes sont présents sur l’ensemble des cosmides séquencés (incluant les gènes ancres et les gènes avoisinants) (400 kilobases). Ces 41 gènes sont alors utilisés pour rechercher des séquences semblables dans les bases de données. Des similitudes significatives sont observées pour 34 gènes (7 gènes ne montrent pas de similitudes significatives avec les séquences présentes dans les bases de données), parmi lesquels 33 incluent des séquences humaines. L’analyse des jeux de données permet l’attribution du critère d’orthologie à 31 gènes de l’amphioxus parmi lesquels 9 sont des gènes ancres et 22 des gènes qui leur sont adjacents. L’analyse des relations phylogénétiques des gènes ancres de l’amphioxus permet de conclure que les duplications ayant engendré les différentes familles de gènes ancres chez les vertébrés se sont produites entre 766-420 millions d’années (pour la diversification des vertébrés osseux) et probablement entre 766-528 millions d’années (pour la séparation des vertébrés à mâchoires).
| Figure 2. Évolution de la région du proto-CMH des chordés et de ses gènes. A. Hypothèse de reconstitution de l’évolution des régions génomiques paralogues du CMH. En comparant les régions de l’amphioxus et les régions de paralogie humaines, il est possible de reconstituer le proto-CMH ancestral des vertébrés à mâchoires. L’état de la région génomique du chordé ancestral reste inconnu en raison du caractère non lié des gènes de l’amphioxus. Les gènes ancres sont indiqués en rouge, les gènes adjacents aux gènes ancres de l’amphioxus et de leurs orthologues humains sont indiqués en noir. B. Profils de substitution au sein des gènes paralogues du CMH humain. Nous avons utilisé uniquement les gènes qui ont au moins trois copies dans les régions de paralogie du CMH. Pour chacun de ces gènes, nous avons comparé le taux de substitution des différents gènes paralogues. Les gènes indiqués en bleu ont un profil de substitution significativement plus lent que les gènes indiqués en rouge. Les boîtes jaunes correspondent aux paralogues perdus. La position relative des gènes dans les régions de paralogie du CMH humain est arbitraire. |
L’analyse de la signification statistique de la distribution des gènes humains orthologues des gènes de l’amphioxus a été réalisée dans quatre régions de paralogie du CMH humain, sur les chromosomes 1, 6, 9 et 19 (Figure 2) . Vingt-deux gènes de l’amphioxus situés au voisinage des gènes ancres clonés ont au moins un orthologue humain pour un total de 42 orthologues humains parmi lesquels seize sont localisés dans une région de paralogie du CMH. La distribution des gènes humains orthologues des gènes voisins des gènes ancres de l’amphioxus est statistiquement différente d’une distribution au hasard pour ces quatre régions ce qui indique l’existence d’un lien évolutif entre les régions génomiques de l’amphioxus et les quatre régions génomiques humaines. Le modèle le plus probable permettant d’expliquer le lien entre les régions génomiques de l’amphioxus et les régions de paralogie dans le CMH humain est l’existence d’un ancêtre génomique commun aux quatre régions génomiques humaines ayant subi une duplication en bloc après la séparation céphalochordés-vertébrés. Les régions de paralogie du CMH sont donc des régions parasynténiques (voir glossaire) qui ont été dupliquées en bloc au cours d’une période comprise entre 766 et 528 millions d’années.
L’aspect le plus frappant de l’évolution des régions dupliquées est le nombre de gènes fixés. La région génomique du chromosome 9 contient en effet deux fois le nombre de la plupart des gènes dérivés de la région génomique ancestrale contrairement aux trois autres régions. Ainsi, en termes d’organisation génomique, la région du chromosome 9 est beaucoup plus semblable à la région génomique ancestrale supposée que ne le sont les trois autres régions de paralogie du CMH. Le caractère ancestral de la région du chromosome 9 s’observe également au niveau des gènes. L’analyse du modèle de substitution (calcul du taux de changement des acides aminés le long de la séquence permettant d’estimer la vitesse d’évolution d’une protéine) des familles géniques ayant plus de deux paralogues dans les régions de paralogie du CMH (six familles paralogues) montre que les gènes de la région du chromosome 9 ont toujours un taux de substitution faible comparé aux trois autres régions paralogues (6 gènes sur 6, ce qui est statistiquement significatif) induisant une évolution moins rapide des gènes de cette région. L’essence d’un tel phénomène reste obscure, mais divers processus peuvent être impliqués comme les contraintes fonctionnelles, les contraintes d’expression et de régulation (expression ubiquitaire versus spécifique d’un tissu).
Ce travail suggère que les régions dupliquées ont des destins différents: une région peut être « préservée » (contrainte fonctionnelle) et entretenir une fonction ancestrale alors que d’autres peuvent être plus « libres », conduisant potentiellement à l’émergence de nouveaux gènes et/ou familles géniques ce qui est supposé pour la « véritable » région génomique du CMH sur le chromosome 6 [4]. L’échantillonnage d’autres régions génomiques est nécessaire afin de déterminer à quel point ce phénomène peut être généralisé.
Cette étude valide donc l’hypothèse de la duplication en bloc des régions de paralogie du CMH et constitue la première étape démontrant que des processus de polyploïdisation se sont produits dans notre génome ancestral après la séparation céphalochordés/vertébrés et avant la radiation des gnathostomes, c’est-à-dire entre 766 et 528 millions d’années. En reconstruisant la région ancestrale des quatre régions de paralogie du CMH, nous avons découvert un aspect inattendu de l’évolution des régions dupliquées: une région a conservé un état ancestral à la fois en terme d’organisation et de profil de substitution des gènes. Notre génome comporte donc une grande région apparemment sous sélection négative (processus qui freine l’évolution des séquences; par exemple, une contrainte fonctionnelle) un concept appliqué peu fréquemment aux gènes pris individuellement ou aux segments de gènes. Il est important de déterminer dans quelle mesure cette découverte peut être généralisée à d’autres régions du génome. Ces analyses soulignent l’importance de la phylogénie et de la phylogénomie, non seulement pour révéler l’histoire évolutive de notre génome mais également pour une meilleure compréhension des organisations génomiques actuelles.