| |
| Med Sci (Paris). 2002 November; 18(11): 1157–1159. Published online 2002 November 15. doi: 10.1051/medsci/200218111157.« Mourir ou ne pas mourir » telle est la question et comment un minuscule petit ver y répondit Prix Nobel de Médecine 2002 Michel Labouesse* IGBMC, 1, rue Laurent Fries, BP10142, 67404 Illkirch Cedex, France |
L’aventure commence en Angleterre au milieu des années 1960. Sydney Brenner venait de participer en compagnie de François Jacob et Francis Crick à la découverte de l’ARN messager et du code génétique, et à ce titre aurait déjà mérité de recevoir le prix Nobel. À l’époque le sentiment de ces pionniers était que l’un des plus grands problèmes de la biologie, celui de la transmission du patrimoine génétique et sa transformation en protéine venait d’être résolu. Aux yeux de S. Brenner, le défi suivant était de comprendre quelles règles président à l’élaboration d’un organisme multicellulaire et au fonctionnement du système nerveux. Il choisit pour répondre à ce défi d’utiliser la recette même qui s’était avérée précédemment payante: l’approche génétique appliquée à des organismes aussi simples que possible. Restait à trouver l’organisme sur lequel appliquer cette recette. Ce fut C. elegans. L’avènement de la génétique moléculaire démontrera l’extraordinaire conservation des gènes au cours de l’évolution et lui donnera raison: ce qui est vrai pour C. elegans l’est pour l’éléphant (et l’homme). C’est ici qu’interviennent deux défricheurs français, deux lyonnais, V. Nigon et J. Brun, qui dès la fin des années 1940 s’étaient intéressés à ce nématode vivant à l’état libre dans la terre, avaient décrit les premières étapes du développement, et montré qu’il est possible d’identifier des mutants morphologiques. Parmi d’autres organismes « concurrents », C. elegans fut retenu en raison de sa durée de vie extrêmement courte (3 jours), de sa transparence, de sa nombreuse progéniture et de la facilité de propagation en laboratoire. Pendant dix années, S. Brenner s’est attaché à créer les outils génétiques qui feront le succès de C. elegans. Dans un article fondateur de 1974, il décrit les conditions de culture, l’isolement de mutations qui affectent la motilité de l’animal (mutants unc) ou sa forme (mutants dpy), et surtout la première carte génétique des six chromosomes [1]. Travaillant au Laboratoire de Biologie Moléculaire du MRC à Cambridge (Royaume-Uni), il eut besoin de toute son énergie pour convaincre ses collègues de l’intérêt de ses recherches. Heureusement, Cambridge ne manque pas d’esprits indépendants. John Sulston en était un qui fut l’un des premiers à le rejoindre, et il pouvait compter sur ses relations d’antan. C’est ainsi que Bob Horvitz, étudiant de Jim Watson et Walter Gilbert - l’un et l’autre prix Nobel - vint le rejoindre pour un postdoc. D’autres esprits aventureux les rallièrent, dont John White qui établit ultérieurement la carte complète des synapses de l’animal. |
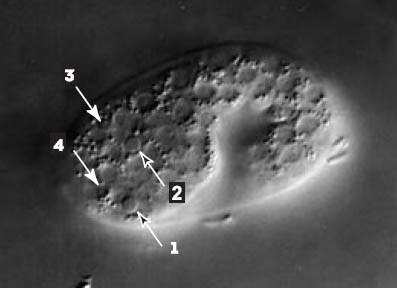
John Sulston (Sir John) fut celui qui bâtit l’édifice permettant l’éclosion de C. elegans en tant qu’organisme modèle et réalisa les premières observations sur l’apoptose. Sa première contribution fondamentale fut de retracer la succession des divisions qui conduisent un œuf fraîchement fécondé à un adulte de 959 cellules - on parle du lignage cellulaire. La quasi-invariance d’un individu à l’autre du cycle des divisions, tant dans l’espace que dans le temps ou dans la nature des cellules filles produites, a rendu possible ce tour de force, auquel Bob Horvitz participa pour les divisions survenant au cours du développement larvaire [2, 3]. Il faut se représenter ce que l’établissement de ce lignage a signifié, pour les expérimentateurs, de travail patient à regarder les cellules se diviser puis à transcrire les observations sous la forme de dessins, le tout à une époque où l’informatique n’existait pas et où la qualité des microscopes était bien moindre. Chemin faisant, John Sulston et ses collaborateurs notèrent que les mêmes 131 cellules meurent invariablement au cours du développement normal (Figure 1). Cette observation leur suggéra qu’il existe un programme spécifique conduisant certaines cellules à mourir tandis que d’autres deviennent, qui des neurones, qui des cellules épithéliales ou encore des cellules musculaires. La mort d’une cellule perdait ses oripeaux de phénomène secondaire et ennuyeux pour accéder au statut de destin prometteur. Ultérieurement, J. Sulston identifia après traitement avec un mutagène chimique trois mutants appelés ced-1, ced-2 et nuc-1 qui affectent l’élimination des cellules mortes et de leur ADN, montrant ainsi qu’il devait être possible d’analyser certaines étapes de l’apoptose par une approche génétique [4].  | Figure 1. Quatre corps apoptotiques dans un embryon de C. elegans, du plus récemment formé (1) au plus résorbé (4). C’est en recherchant des mutants chez lesquels ce type de corps apoptotique n’apparaissait plus ou bien ne disparaissait plus que la plupart des gènes impliqués dans le déclenchement de l’apoptose puis la phagocytose des cellules mortes ont été identifiés. |
La deuxième contribution majeure de Sir John fut, une fois le lignage connu, de s’attaquer à la constitution d’une carte ordonnée du génome de C. elegans sous la forme de cosmides, à l’époque la première entreprise de son genre pour un organisme multicellulaire. Cette carte fut ultérieurement complétée sous l’impulsion de Bob Waterston (Washington University, États-Unis) par des chromosomes artificiels de levure. Puis il s’attaqua, avec Bob Waterston et son collaborateur de longue date Alan Coulson, au séquençage du génome, opération qui vit son achèvement en 1998 avec la publication de la première séquence du génome d’un organisme multicellulaire [5]. Pendant toute l’opération qui s’étala sur 15 ans, John Sulston mit un point d’honneur à diffuser les informations en temps réel ou presque. Il s’est aussi fait l’un des plus ardents pourfendeurs de toutes les tentatives visant à breveter les gènes. |
Robert Horvitz (Bob) fut celui qui utilisa au mieux le matériau créé par S. Brenner et J. Sulston pour ouvrir des pans entiers de la connaissance grâce à une intuition, une persévérance et une énergie impressionnantes. Il décida d’utiliser l’analyse génétique pour trouver de nouvelles mutations affectant le déclenchement de la mort cellulaire programmée [6]. Un grand nombre de ces mutants furent le fruit du travail d’un étudiant, Ron Ellis, qui rechercha patiemment au microscope, parmi près de 50000 animaux ayant été exposés à un mutagène, ceux chez lesquels trop peu de cellules mouraient ou bien trop peu de celllules mortes n’étaient pas phagocytées [7]. Puis, Bob Horvitz et ses collaborateurs surent ensuite donner à ces mutants un visage moléculaire, les ordonner et reconnaître qu’il existe des homologues fonctionnels chez les vertébrés. Ainsi, il montra que l’apoptose résulte d’une cascade d’événements, commençant dans certains cas par l’activation transcriptionnelle d’un gène qui peut déclencher la mort en contrôlant l’activité de la protéine CED-9, un acteur central du processus qui, chez les vertébrés, correspond à Bcl-2 [8, 9]. CED-9 règle lui-même l’activité de deux protéines, appelées CED-4 et CED-3, dont les homologues furent ultérieurement découverts chez les vertébrés, CED-3 étant une protéase – la caspase -qui va provoquer la mort de la cellule [6, 10]. Puis il s’attaqua à la caractérisation moléculaire des gènes impliqués dans la phagocytose des cellules mortes, entreprise qui s’avéra encore une fois féconde [11]. En effet, tous les gènes qu’il identifia sont conservés chez les vertébrés chez lesquels ils participent également au processus phagocytique en contrôlant en particulier le réarrangement du cytosquelette, prélude nécessaire à la phagocytose. Son travail eut bien sûr un retentissement considérable puisqu’il est bien établi maintenant qu’une dérégulation de l’apoptose peut conduire soit à l’apparition de cancers (en cas d’absence), ou bien à un grand nombre de maladies neurodégénératives, de certaines formes d’infarctus, de maladies inflammatoires par exemple (en cas d’excès). Mais la contribution de Bob Horvitz dépasse de beaucoup le cadre de l’apoptose puisqu’il a joué un rôle majeur dans la compréhension des étapes importantes de l’organogenèse et qu’il a formé une génération de neurobiologistes qui dominent actuellement différents domaines des neurosciences. Un très grand nombre de découvertes majeures liées à la recherche sur C. elegans ont été apportées par ses anciens étudiants ou postdocs: on songe par exemple à la mise en évidence d’un lien entre réponse à l’insuline et vieillissement, à la compréhension du rôle des présénilines dans la maladie d’Alzheimer ou encore à la mise en évidence de l’existence des petits ARN non codants. |
« La communauté C. elegans » En dehors de l’importance de la contribution scientifique de ce trio, l’aspect le plus singulier de cette aventure tient en deux mots, solidarité et communauté. Il n’est pas exagéré de dire qu’en 1995, date de la découverte des ARN interférents par des chercheurs utilisant C. elegans et qui a rendu le modèle plus accessible que jamais, 95 % des responsables de laboratoire utilisant C. elegans avaient travaillé avec Sydney Brenner ou Bob Horvitz, ou bien avaient été formés par quelqu’un ayant directement travaillé avec eux. Ce lien étroit et direct, ainsi que la nécessité déjà évoquée de rester soudés pour convaincre un environnement au départ très sceptique, ont forgé un esprit unique de solidarité et de corps dans la communauté, qui a été caractérisée par des échanges très libres d’informations avant publication. Le journal The Worm Breeder’s Gazette en fut longtemps le vecteur. Cet esprit de corps, très certainement insufflé à l’origine par Sydney Brenner, fut la clé du succès de l’entreprise et concrètement se traduisit par l’effort de quelques-uns (J. Sulston en particulier) au service de la communauté. J’espère avoir pu transmettre un peu de l’enthousiasme qui a animé ces trois chercheurs, et est toujours présent chez les générations suivantes. Vive C. elegans ! |
L’auteur remercie J.L. Bessereau pour la relecture de ce texte. |
1. Brenner S. The genetics of Caenorhabditis elegans. Genetics 1974; 77: 71–94. 2. Sulston JE, Horvitz HR. Post-embryonic cell lineages of the nematode Caenorhabditis elegans. Dev Biol 1977; 56: 110–56. 3. Sulston JE, Schierenberg E, White JG, Thomson JN.The embryonic cell lineage of the nematode Caenorhabditis elegans. Dev Biol 1983; 100: 64–119. 4. Hedgecock EM, Sulston JE, Thomson JN. Mutations affecting programmed cell deaths in the nematode Caenorhabditis elegans. Science 1983; 220: 1277–9. 5. The C. elegans Sequencing Consortium. Genome sequence of the nematode C. elegans: a platform for investigating biology. Science 1998; 282: 2012–8. 6. Ellis HM, Horvitz HR. Genetic control of programmed cell death in the nematode C. elegans. Cell 1986; 44: 817–29. 7. Ellis RE, Horvitz HR. Two C. elegans genes control the programmed deaths of specific cells in the pharynx. Development 1991; 112: 591–603. 8. Hengartner MO, Horvitz HR. C. elegans cell survival gene ced-9 encodes a functional homolog of the mammalian proto-oncogene bcl-2. Cell 1994; 76: 665–76. 9. Conradt B, Horvitz HR. The TRA-1A sex determination protein of C. elegans regulates sexually dimorphic cell deaths by repressing the egl-1 cell death activator gene. Cell 1999; 98: 317–27. 10. Yuan J, Shaham S, Ledoux S, Ellis HM, Horvitz HR. The C. elegans cell death gene ced-3 encodes a protein similar to mammalian interleukin-1 betaconverting enzyme. Cell 1993; 75: 641–52. 11. Wu YC, Horvitz HR. C. elegans phagocytosis and cell-migration protein CED-5 is similar to human DOCK180. Nature 1998; 392: 501–4. |