| |
| Med Sci (Paris). 2002 December; 18(12): 1245–1256. Published online 2002 December 15. doi: 10.1051/medsci/200218121245.Architecture du noyau et régulation transcriptionnelle Frédéric Pâques and Thierry Grange Institut Jacques Monod du Cnrs, Universités Paris 6-7, Tour 43, 2, place Jussieu, 75251 Paris Cedex 05, France |
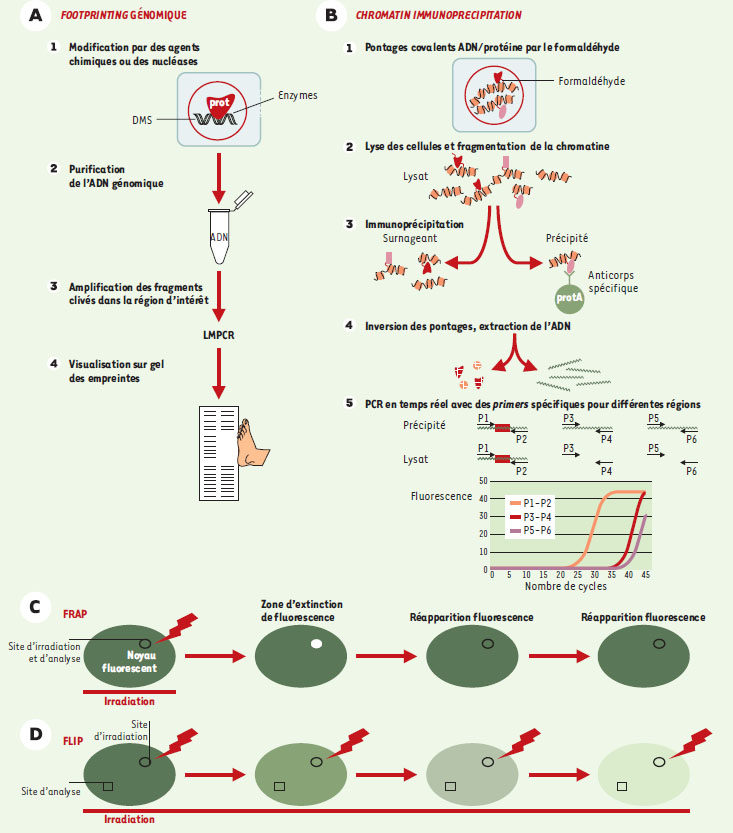
Comportement dynamique des protéines réglant la transcription Notre compréhension des mécanismes de régulation transcriptionnelle chez les eucaryotes supérieurs a reposé, pendant de nombreuses années, sur les concepts issus des approches biochimiques développées à partir de quelques systèmes modèles (levure, embryons de drosophile ou de xénope, cellules HeLa, foie de rat) et complétées par des approches génétiques (essentiellement menées chez la levure et la drosophile) et de transgenèse (une grande partie de cette dernière approche repose sur l’analyse du comportement biologique de cellules transfectées in vitro de façon transitoire). Récemment, notre connaissance de ces mécanismes s’est singulièrement enrichie : d’une part, les approches de biochimie et de biologie moléculaire s’efforcent d’être moins réductionnistes et intègrent de plus en plus les événements individuels dans leur contexte chromati-nien et chromosomique; d’autre part, les approches de biologie cellulaire entrent en scène et l’innovation technologique dans ce domaine permet d’analyser de plus en plus finement l’organisation nucléaire et de suivre les mouvements nucléaires des complexes identifiés par la biochimie. Cette convergence des approches réductionnistes et globales introduit beaucoup de dynamique dans une vision auparavant bien trop statique. Deux voies d’études ont largement contribué à cette convergence: (1) la généralisation des méthodes d’analyse biochimiques ex vivo (Figure 1 AB) a permis de déterminer la séquence des événements moléculaires menant à l’activation d’une poignée de gènes in chromosomo1; (2) l’analyse du comportement après photobleaching de protéines de fusion avec les protéines fluorescentes (GFP, Figure 1 CD) a permis de suivre la dynamique moléculaire de ces protéines au sein du noyau. La confrontation des données issues de ces approches permet de commencer à établir des modèles intégrés qui nous rappellent ce que les physiciens savent depuis longtemps mais qu’un certain nombre de biologistes n’ont pas toujours gardé à l’esprit: les molécules bougent sans arrêt.  | Figure 1.Méthodes moléculaires et cellulaires d’étude des interactions ADN/protéine et de la dynamique de ces interactions. A. Footprinting in vivo. L’amplification du signal par LMPCR ( ligation mediated polymerase chain reaction) et le marquage sont détaillés dans [ 60]. B. Immunoprécipitation de chromatine (ChIP). C. FRAP. La protéine de fusion fluorescente, exprimée in vivo, occupe en général l’essentiel du noyau (vert) avec une densité variable suivant les régions. Une petite partie du noyau (cercle noir) est soumise à une irradiation par un laser, jusqu’à extinction de la fluorescence. Après arrêt de l’irradiation, on mesure le temps de recouvrement de la fluorescence dans la zone irradiée. Ce recouvrement de la fluorescence témoigne de l’arrivée de molécules situées auparavant hors de la région irradiée. D. FLIP. Une petite région est soumise à une irradiation continue. On mesure alors la perte de fluorescence en un autre point (rectangle) du noyau. Cette perte de fluorescence indique la fraction mobile des protéines présentes dans le noyau (et qui finit donc par passer sous le rayon laser). |
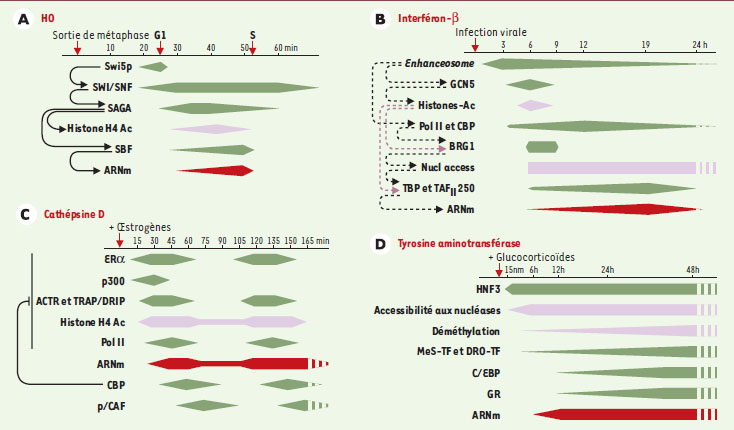
Cinétique de l’activation de la transcription au niveau moléculaire Les approches biochimiques et génétiques ont permis d’identifier une pléthore de co-régulateurs et d’activités enzymatiques susceptibles de jouer un rôle dans la régulation transcriptionnelle [ 1, 2]. Cependant, l’implication réelle de ces protéines dans la régulation de l’expression des gènes, leur rôle exact et les modalités de leur action restaient ambigus. Les analyses biochimiques telles qu’elles sont réalisées, soit purement in vitro, dans un contexte acellulaire, soit ex vivo dans des systèmes de transfection transitoire de molécules d’ADN, ne reconstituent que partiellement ce qui se passe sur le chromosome, et les concentrations absolues et relatives des différents composants analysés sont la plupart du temps très éloignées de celles qui existent dans les conditions physiologiques. Les analyses génétiques, particulièrement lourdes à mettre en œuvre chez les vertébrés, sont souvent d’interprétation difficile, du fait de la complexité des réseaux génétiques au sein desquels les régulateurs transcriptionnels interviennent, et de la redondance des fonctions au sein des différents membres des familles multigéniques. Les approches moléculaires d’analyse ex vivo des interactions entre protéines et gènes ont permis de lever un certain nombre d’ambiguïtés et de compléter ou de réévaluer l’idée que l’on se faisait de la fonction de ces régulateurs. En effet, ces approches ont mis en évidence les modalités et les cinétiques de recrutement d’un certain nombre de facteurs au niveau de quelques gènes cibles, et permis l’établissement de parallèles entre ces recrutements et les événements aboutissant aux modifications de ces gènes. Deux méthodes d’analyse ont été particulièrement utilisées: le footprinting génomique (Figure 1A) [3], et l’immunoprécipitation de la chromatine (ChIP, Figure 1B) [4]. Les méthodes de footprinting génomique consistent à suivre indirectement les changements de conformation chromatinienne et les recrutements de protéines contactant l’ADN en analysant la réactivité de l’ADN vis-à-vis d’agents chimiques ou de nucléases. La qualité des informations que l’on peut obtenir de cette analyse repose sur la capacité d’établir un parallèle entre la réactivité intracellulaire de l’ADN et les changements de réactivité induits par des événements que l’on a pu reconstituer in vitro. L’immunoprécipitation de la chromatine permet, grâce à l’utilisation d’anticorps dirigés contre des épitopes spécifiques, de suivre directement le recrutement ou les modifications covalentes de protéines faisant partie d’un complexe contactant l’ADN. Cette approche directe peut paraître plus séduisante, mais elle est toutefois limitée par la qualité des anticorps utilisés (spécificité, affinité, sensibilité aux modifications induites par l’agent pontant utilisé) et ne dépend pas uniquement de la présence des épitopes reconnus mais aussi de leur accessibilité, ce qui peut fausser les interprétations. Il est donc sage de se garder de penser que l’ensemble des résultats obtenus avec cette approche est solidement établi. Au cours des deux dernières années, l’utilisation systématique de ces approches a permis d’établir un scénario relativement précis de l’activation transcriptionnelle d’une poignée de gènes. La Figure 2 présente quatre exemples représentatifs illustrant la diversité de mécanismes d’activations mettant en jeu des combinaisons différentes d’acteurs.  | Figure 2. Quatre exemples d’activation de gènes modèles. Les recrutements de facteurs de transcription sont figurés en vert, les modifications de chromatine en violet clair et l’activation transcriptionnelle en rouge. Les pointillés indiquent que les processus semblent se poursuivre au-delà des périodes analysées. A. HO. Les flèches traduisent des relations de cause à effet observées in vivo. B. Interféron-β. Les flèches traduisent des relations de cause à effet observées in vitro. C. Cathepsine D. D. Tyrosine aminotransférase. Nucl access : accessibilité aux nucléases. |
La première cascade d’événements à avoir été caractérisée in chromosomo a été l’activation du gène HO de levure (Figure 2A) [5]. La fixation transitoire du facteur Swi5 en fin de mitose déclenche le recrutement séquentiel d’une série de protéines, qui aboutira à l’expression de l’endonucléase HO en fin de phase G1. Parmi les corégulateurs recrutés, on trouve deux complexes de protéines agissant sur la chromatine: l’un, le complexe SWI/SNF, utilise l’ATP pour permettre les mouvements des nucléosomes, l’autre, le complexe SAGA, déclenche l’acétylation des queues N-terminales des histones. Lors de l’activation du gène HO, l’action du complexe SWI/SNF précède celle du complexe SAGA et la conditionne. Il semble que ceci soit une situation particulière à la sortie de mitose, moment où le remodelage des nucléosomes par le complexe SWI/SNF serait nécessaire afin de rendre la chromatine des chromosomes condensés accessible aux complexes acétyltransférases [6]. Un autre cas bien décrit est celui de l’activation du gène de l’interféron-β en réponse à une infection virale (Figure 2B). L’analyse de son mécanisme a été effectuée à la fois ex vivo (ChIP) à partir de cellules HeLa, et in vitro (système acellulaire reconstitué), et les résultats de ces deux approches sont concordants [7]. Comme dans l’exemple précédent, certains facteurs (Gcn5 et Brg1) sont recrutés de façon transitoire, tandis que d’autres perdurent jusqu’à la dissociation des protéines liant l’enhancer, une vingtaine d’heures après l’infection. Mais, contrairement au système précédent, le recrutement d’un membre d’un complexe histone acétyltransférase (GCN5) précède, et semble permettre, celui d’un membre d’une machinerie de remodelage utilisant l’ATP (BRG1). In vitro, l’acétylation des histones pourrait faciliter l’action des machineries de remodelage utilisant l’ATP en créant des sites d’interaction pour les protéines de ces complexes (BRG1 et hBrm) qui contiennent des motifs de reconnaissance des queues acétylées: les domaines bromo [8]. La cinétique de recrutement des facteurs diffère selon l’architecture des séquences régulatrices comme le montre l’analyse in vitro de l’enhancer du gène de l’interféron-β [7]. L’ enhancer sauvage comprend des sites de fixation pour les facteurs ATF-2, NF-κB et IRF1 qui recrutent la protéine CBP qui recrute elle-même l’ARN polymérase II d’une manière qui précède le recrutement de la protéine TBP (Figure 2B). Lorsque cet enhancer est remplacé par un multimère de sites NF-κB, le recrutement de l’ARN polymérase II devient dépendant de celui de TBP, et indépendant de CBP [7]. L’événement final (l’activation de la transcription) peut donc s’effectuer en suivant deux chemins différents, suivant la combinatoire des facteurs interagissant avec les séquences régulatrices et la généralisation de ce concept expliquerait fort bien la divergence des résultats obtenus sur les différents systèmes expérimentaux étudiés. L’ activation du gène de la cathepsine D par le récepteur des œstrogènes (ERα) représente un cas intéressant par le caractère cyclique du recrutement de différents complexes de co-activateurs (Figure 2C). Une première étude avait montré que la transcription de ce gène était induite en une heure, parallèlement au recrutement d’un ensemble de co-activateurs, mais chutait quelques heures plus tard, après que ces co-activateurs se soient dissociés, probablement à la suite de l’acétylation de l’un d’entre eux (ACTR) par un autre (CBP) [9]. Une deuxième étude, analysant plus précisément la cinétique précoce de cette réaction, a permis de mettre en évidence le caractère cyclique de l’activation transcriptionnelle, ainsi que de l’assemblage des complexes de coactivateurs [10]. L’activation du gène codant pour la tyrosine aminotransférase (Tat) de rat par le récepteur des glucocorticoïdes (GR) représente encore un autre cas de figure, où des interactions transitoires du GR déclenchent la construction progressive d’un complexe impliquant un nombre croissant de facteurs interagissant de façon coopérative, permettant ainsi l’augmentation de la durée de vie de l’interaction du GR avec cet enhancer (Figure 2D) [11]. Dans ce cas, le recrutement des complexes co-activateurs n’a pas été caractérisé. Cependant, la fixation des différents facteurs de transcription et le remodelage de la chromatine ont été décrits de façon précise. Le GR induit tout d’abord un remodelage de la chromatine au niveau d’un enhancer éloigné de 2,5 kb du promoteur et, simultanément, le recrutement du facteur HNF3 dans cette région. À ce stade, aucune interaction stable entre le GR et l’enhancer ne peut être détectée par footprinting car le GR n’interagit avec ses séquences cibles que de façon transitoire bien que récurrente [12]. Au cours d’une activation prolongée (48 heures), le GR déclenche en outre la déméthylation de quatre cytosines situées dans l’enhancer, permettant le recrutement de deux nouvelles protéines (MeS-TF et DR0-TF) [13], ainsi que la stimulation de l’expression du facteur C/EBP et son recrutement stable sur l’enhancer [11]. Lorsque l’ensemble des autres activateurs transcriptionnels a été recruté, l’interaction du GR avec l’ADN est stabilisée, probablement grâce aux multiples interactions entre protéines liées à l’enhancer. Ces événements locaux déclenchent d’une part une hyperacétylation des histones H3 et H4 qui se propage sur 6 kb et, d’autre part, l’activation de l’ARN polymérase II au niveau du promoteur proximal. Dans ces quatre exemples, les événements s’enchaînent dans un ordre déterminé qui conditionne l’entrée en scène des différents facteurs mais le scénario diffère dans chaque cas. L’ensemble des données obtenues actuellement suggère que ces différences de scénario seraient dues, d’une part, à la nature de l’assemblage des facteurs régulateurs se liant à l’ADN et, d’autre part, à la structure de la chromatine établie préalablement à l’action de ces facteurs régulateurs. Dynamique des complexes au niveau cellulaire Cependant, les méthodes moléculaires que nous avons décrites, ChIP et footprinting in vivo ne mesurent que l’occupation moyenne d’un site dans les cellules d’une population donnée et, de ce fait, présentent un certain nombre de limites. Tout d’abord, elles ne rendent pas compte de l’hétérogénéité de la dynamique chromatinienne au sein d’une même population. Par exemple, elles laissent penser que l’activation du gène Tat est un phénomène progressif, alors que les méthodes d’ARN FISH ( fluorescent in situ hybridization), qui permettent de suivre la transcription de chaque gène individuellement dans chaque noyau, suggèrent un modèle stochastique. Nous avons ainsi observé que la proportion de cellules où le gène Tat est transcrit passe de 2 % à 80 % au cours de deux jours d’activation, et que les gènes sont transcrits de façon intermittente au cours de cette période. Par ailleurs, les méthodes de biologie moléculaire ex vivo ne permettent pas de détecter des interactions dont la durée de vie est courte et qui ne se produisent pas de manière synchrone. Dans l’exemple du contrôle du gène de la cathepsine développé ci-dessus, l’alternance des phases d’association et de dissociation des complexes activateurs avec ce gène a été identifiée car la durée de chaque phase était suffisamment longue (1h30), et que leur survenue était synchrone dans la population [ 10]. Les approches strictement moléculaires ne sont donc pas les plus appropriées à l’étude de la dynamique des complexes. Deux méthodes, le FRAP ( fluorescence recovery after photobleaching) et le FLIP ( fluorescence loss in photobleaching), ont permis récemment de faire évoluer notre vision de la dynamique des structures nucléaires (Figure 1 CD) [ 14]. Ces méthodes sont toutes deux fondées sur l’utilisation de protéines fusionnées avec la GFP, et elles ont l’avantage d’être applicables à l’observation de cellules individuelles. En revanche, la microscopie n’est pas actuellement assez résolutive pour permettre l’analyse de gènes uniques. Ces approches ont été particulièrement utiles pour montrer que des structures bien définies et apparemment assez stables, comme le nucléole et les compartiments où sont concentrés les facteurs d’épissage, sont en fait le siège d’un flux perpétuel de protéines s’y associant et s’en dissociant, et que les protéines les constituant sont renouvelées en moins d’une minute [ 14, 15]. La plupart de ces mouvements ne nécessitent pas d’énergie chimique et semblent dus à une diffusion aléatoire des protéines dans le nucléoplasme, diffusion limitée par les interactions que ces protéines établissent entre elles et avec la chromatine. En fait, la chromatine, et particulièrement les histones du cœur du nucléosome semblent être les éléments les moins mobiles du noyau. Plus de 80 % des histones H3 et H4 assemblées lors de la réplication ne bougent plus jusqu’à la réplication suivante [ 16]. Les histones H2A et H2B, qui semblent pouvoir être dissociées du nucléosome lors de la transcription, sont plus mobiles : 3 % peuvent s’échanger en quelques minutes, et 40 % en quelques heures [ 16]. L’histone H1 serait encore plus mobile, 75 à 90 % de ces molécules pouvant s’échanger en 4 minutes, bien que la fraction d’histone H1 associée à l’hétérochromatine semble beaucoup plus statique [ 17]. Finalement, certaines protéines chromatiniennes comme HMG17 ne resteraient pas associées à la chromatine plus de 30 secondes [ 15]. Afin de faciliter la visualisation de la dynamique des interactions de facteurs régulateurs de la transcription au niveau des gènes cibles en utilisant ces approches microscopiques, les expériences ont été réalisées sur des locus amplifiés en tandem et répétés de quelques dizaines à quelques milliers de fois [ 18– 20]. Même si ces locus amplifiés en tandem sont très artificiels, les résultats obtenus confirment le caractère dynamique de l’interaction des facteurs régulateurs avec les gènes cibles. En revanche, les facteurs sont encore plus mobiles que les observations biochimiques ex vivo ne le laissaient supposer. L’interaction d’une fusion entre le GR et la GFP avec une répétition de 200 copies en tandem d’un gène cible du GR (le LTR du virus murin de tumeur mammaire) se matérialise par l’apparition d’un site intense de fluorescence en réponse à la stimulation par le ligand hormonal [ 20]. L’analyse par FRAP, ou par FLIP, a permis de montrer que le GR restait associé à cette cible moins de 10 secondes. En présence d’hormone, ce site contient aussi les activateurs AP-2 et NF1 et les coactivateurs CBP, SRC1 et BRG1 [ 21]. Le recrutement des co-activa-teurs SRC1 et CBP par une protéine chimérique dérivée du récepteur des œstrogènes (ER), une fusion CFP-répresseur lac-ER, au niveau de multimères d’opérateur lac montre aussi que les interactions entre récepteurs des stéroïdes et co-activateurs sont très dynamiques et que les co-activateurs recrutés sur le gène cible s’échangent en moins de 30 secondes [ 22]. En conclusion, les approches de biologie moléculaire ex vivo et de biologie cellulaire convergent pour montrer que les interactions de facteurs régulateurs de la transcription avec leurs gènes cibles sont très dynamiques. Cette dynamique doit permettre une adaptation très rapide de l’expression génétique aux variations d’intensité des signaux qui la règlent. |
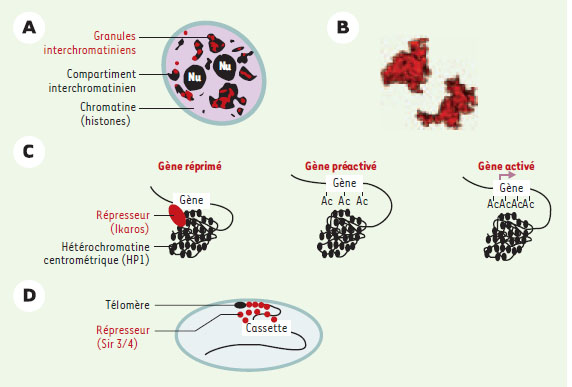
Aspects dynamiques de la compartimentation du noyau interphasique L’intérieur du noyau interphasique n’est pas compartimenté par des membranes, mais on y distingue des zones où certains composants nucléaires sont concentrés, créant des structures identifiées depuis longtemps par les approches microscopiques: nucléole, corps de Cajal, corps PML (promyelocytic) ou granules interchromatiniens [23]. Comme nous l’avons discuté auparavant, ces structures résultent d’interactions extrêmement dynamiques entre protéines s’assemblant entre elles et/ou avec certaines structures chromatiniennes. La chromatine peut être divisée en deux compartiments clairement distincts en microscopie électronique: l’hétérochromatine et l’euchromatine. L’hétérochroma-tine apparaît plus condensée: elle n’est pas transcrite et une partie est concentrée à la périphérie du noyau, probablement parce que la protéine d’hétérochromatine HP1 interagit avec le récepteur de la lamine B qui est fiché dans la membrane nucléaire interne. Par ailleurs, les chromosomes n’occupent pas l’intérieur du noyau comme des spaghettis, mais chaque chromosome occupe un territoire chromosomique distinct [24]. Cette apparente organisation du noyau est certainement liée à son activité fonctionnelle, comme en témoignent les changements majeurs de l’architecture nucléaire en fonction de l’état de différenciation de la cellule [25]. En revanche, la nature des liens entre organisation nucléaire et expression génétique n’est toujours pas claire: est-ce que la structure pré-existe à la fonction et la conditionne, ou est-elle simplement la résultante de l’activité fonctionnelle? Certains liens entre expression génétique et organisation nucléaire commencent à se préciser et il semble que ces deux aspects soient étroitement interdépendants, les modifications de la structure chromatinienne et de l’activité transcriptionnelle induiraient des réorganisations architecturales tandis que la topologie affecterait la probabilité de modification de la structure chromatinienne et de l’expression des gènes. Territoires chromosomiques et architecture nucléaire Chez les mammifères, la répartition de la chromatine dans le noyau a été étudiée dans des cellules vivantes grâce à des protéines résultant de la fusion entre histones et GFP (Figure 3A) [ 24]. La chromatine n’occupe pas tout le volume du noyau et laisse apparaître des lacunes, qui définissent le compartiment interchromatinien, dans lequel sont concentrées les « machineries » de maturation des transcrits, comme les granules interchromatiniens (Figure 3A). Les approches d’hybridation in situ avec des sondes détectant spécifiquement un chromosome, ou les marquages de l’ADN des chromosomes ex vivo au cours de la réplication, ont permis d’assigner à chaque chromosome un volume spécifique: le territoire chromosomique (Figure 3B) [ 24]. L’existence d’un territoire délimité implique qu’au sein de chaque chromosome la fibre chromatinienne se replie sur elle-même, ou sur des protéines ancres, mais la nature des interactions permettant un tel repliement n’est pas connue [ 24]. Si la position relative de chaque territoire chromosomique ne semble pas déterminée, en revanche, les chromosomes riches en chromatine inactive sont plutôt en périphérie du noyau [ 26]. Cela résulte probablement de l’interaction de l’hétérochromatine avec la membrane nucléaire interne.  | Figure 3. Compartimentation nucléaire et impact sur l’expression génétique. A. La chromatine (fond violet clair) et les machineries d’épissage n’occupent pas le même espace nucléaire. Type d’image obtenue avec un double marquage des histones et d’un composant du splicéosome [24]. B. Territoires chromosomiques: reconstruction de l’espace occupé par deux chromosomes homologues d’une même cellule [24]. Notez les inhomogénéités de forme des deux chromosomes homologues, la compaction des territoires et les nombreuses invaginations. C. Modélisation des corrélations observées entre la position relative d’un gène vis-à-vis de l’hétérochromatine centromérique et son expression dans le cas des gènes β-globine ou des gènes cibles de la protéine Ikaros. D. Modèle de répression des cassettes silencieuses du type sexuel chez la levure grâce à la proximité d’une zone de concentration des facteurs répresseurs Sir3 et Sir4 recrutés par les télomères. |
Cette organisation en territoires chromosomiques séparés les uns des autres par un espace interchromatinien interconnecté et riche en composants intervenant lors des processus de transcription et d’épissage suggère que la production et la maturation des ARN pourraient avoir lieu à la surface des territoires. En effet, bien que les territoires aient des formes irrégulières et possèdent de nombreuses invaginations (Figure 3B), il semble que les molécules de grande taille et donc les complexes préformés qui assurent la production des ARN ne puissent pas diffuser librement à l’intérieur [24]. Les ARNm nouvellement synthétisés se trouvent en périphérie des territoires, suggérant que leur transcription ait bien lieu à la surface des chromosomes [27]. Selon ce modèle, la position d’un gène dans un territoire chromosomique devrait donc influencer les modalités de son expression. Bien que la généralité du phénomène soit discutée [61], on observe de telles corrélations dans un certain nombre de cas. Par exemple, un gène porté par le chromosome X inactif et, de ce fait, inactivé, est plutôt localisé au centre du territoire spécifiquement assigné au chromosome X inactif [28]. En revanche, un gène porté par le X inactif, mais échappant à l’inactivation, semble plutôt en périphérie du territoire correspondant. Le complexe majeur d’histocompatibilité se trouve préférentiellement sur une boucle chromatinienne qui s’étend en dehors du territoire du chromosome qui le porte, et la fréquence avec laquelle cette extrusion est observée dépend de l’activité transcriptionnelle du locus [29]. Si les gènes actifs se trouvent bien en périphérie des territoires et les gènes inactifs au centre, comment ces derniers peuventils être réactivés? On peut imaginer au moins deux scénarios: (1) l’activation pourrait être déclenchée par des protéines pionnières qui ne seraient pas assemblées dans des complexes, et pourraient donc diffuser au cœur du territoire; (2) l’activation serait contemporaine d’une phase du cycle cellulaire où la topologie du territoire est remaniée, par exemple lors de la réplication de l’ADN ou en sortie de mitose. En effet, ces deux phases du cycle s’accompagnent d’importants mouvements de portions de chromosome [30]. Dans les cas où l’activation transcriptionnelle d’un gène inactif peut s’effectuer en l’absence de réplication de l’ADN, quel type de mécanisme permettrait de repositionner les gènes actifs en périphérie? On peut imaginer que les reconfigurations de la chromatine induites par des activités de remodelage ou par la machinerie transcriptionnelle puissent permettre une telle relocalisation. En effet, la fixation sur l’ADN d’un activateur fort induit une décondensation majeure de la fibre chromatinienne visible sur les multimères de séquences cibles [18]. De même, la stimulation de la transcription par l’ARN polymérase II (une machinerie efficace de remodelage de la chromatine [31]) induit le même type de réarrangement [21]. Un tel modèle topologique de contrôle de l’expression génétique permet de concilier deux notions antagonistes: l’existence d’un état stable d’activité de la transcription et l’instabilité des interactions des protéines qui contrôlent cette expression génétique. En effet, un gène inactif se trouvant dans un espace appauvri en complexes macromoléculaires permettant la transcription sera difficilement activable, ce qui stabilisera son état inactif. En revanche, une fois le gène réactivé et relocalisé dans un espace enrichi en machineries de transcription, il sera beaucoup plus facilement activable, ce qui stabilisera son état actif. Compartimentation des gènes cibles et des facteurs régulateurs Même si la compartimentation du contrôle de l’expression génétique au sein du territoire chromosomique n’est pas encore solidement établie, plusieurs exemples liant topologie nucléaire et expression génétique montrent de plus en plus clairement l’importance de la dimension topologique. Tous font intervenir l’association avec le compartiment hétérochromatinien et/ou la périphérie nucléaire où l’hétérochromatine est concentrée chez les eucaryotes supérieurs. On sait depuis longtemps que l’hétérochromatine peut inactiver la transcription en cis des gènes qui sont insérés à proximité de cette hétérochromatine, et l’hétérochromatine se propagerait ainsi le long des chromosomes à partir de « points d’entrée ». Mais, plus récemment, on a observé quelques cas d’association entre hétérochromatine et gènes inactifs dans l’espace nucléaire en l’absence de proximité le long du chromosome. Le premier exemple clair vient de la drosophile où, contrairement aux mammifères, les chromosomes homologues sont appariés dans le noyau interphasique. L’insertion d’un bloc d’ADN satellite d’hétérochromatine à proximité d’un gène (brown) entraîne l’inactivation des deux allèles du gène, qu’ils portent ou non l’insertion (phénotype brown dominant), ainsi que leur relocalisation à proximité de l’hétérochromatine périnucléaire [32, 33]. Il est probable que ce soit cette relocalisation de l’allèle sauvage dans un territoire nucléaire inactif qui soit responsable de son inactivation. Chez les mammifères, on a observé plusieurs cas d’association différentielle de gènes avec l’hétérochromatine centromérique en fonction de leur état d’expression (Figure 3C). Au cours du développement des cellules érythroïdes, ou à la suite de diverses mutations de séquences régulatrices, le locus portant le gène codant pour la chaîne β de la globine humaine peut exister sous divers états d’activité, soit complètement inactivé et fermé (chromatine résistante aux nucléases et hypo-acétylée), soit ouvert mais inactif (chromatine sensible aux nucléases et acétylée sur plusieurs dizaines de kb), soit actif (Figure 3C) [34]. Dans l’état fermé et inactif, le locus est associé à l’hétérochromatine centromérique dans le noyau interphasique, tandis qu’il en est dissocié dans l’état pré-activé comme dans l’état actif. Ce n’est donc pas la transcription elle-même qui est responsable de cette relocalisation. En revanche, on ne sait pas si c’est la relocalisation qui permet l’ouverture de la chromatine, ou si c’est cette ouverture qui induit la relocalisation. Au cours de la différenciation des lymphocytes B ou T, on observe aussi une relocalisation de gènes au voisinage de l’hétérochromatine lorsqu’ils sont inactivés [35, 36]. Ce processus semble sous le contrôle de la protéine Ikaros, une protéine qui se lierait à la fois au promoteur des gènes inactivés et à l’ADN satellite centromérique et entraînerait de ce fait l’association de ces gènes avec l’hétérochromatine centromérique [37]. Le recrutement par Ikaros des allèles inactifs des chaînes lourdes et légères des immunoglobulines à proximité de l’hétérochromatine centromérique pourrait aussi être responsable de l’exclusion allélique [38]. Cette dynamique de localisation différentielle à proximité de l’hé-térochromatine peut aussi être observée avec des transgènes. Un transgène inséré dans l’hétérochromatine centromérique est localisé en périphérie de cette hété-rochromatine sous l’action de facteurs régulateurs, et au centre de cette hétérochromatine en l’absence d’action de ces facteurs régulateurs [39]. Ce mouvement hors du centre de l’hétérochromatine semble nécessaire, mais n’est pas suffisant pour que le transgène soit exprimé. Finalement, dans le cas d’une insertion en tandem d’un grand nombre de copies d’un transgène portant un opérateur lac dans un site qui provoquait une localisation préférentielle de ce transgène en périphérie du noyau, on a observé que son activation par une chimère VP16-répresseur lac-GFP induisait une relocalisation du transgène vers l’intérieur du noyau [19]. Ces résultats suggèrent qu’un activateur transcriptionnel peut dissocier l’interaction d’un gène inactif avec l’hé-térochromatine lorsqu’il agit sur ce gène. Est-ce parce que les gènes inactifs sont assemblés en hétérochroma-tine qu’ils tendent à se localiser à proximité de l’hétéro-chromatine (HP1 pouvant interagir avec elle-même), ou est-ce parce qu’ils sont à proximité de l’hétérochroma-tine qu’ils sont inactifs (HP1 étant recruté plus facilement du fait que sa concentration locale est plus élevée dans ce voisinage)? Il est probable que les deux phénomènes agissent ensemble et se renforcent mutuellement. L’analyse chez la levure de la formation de structure chromatinienne répressive au niveau des télomères et des cassettes silencieuses du type sexuel (HMR et HML) apporte des arguments en faveur d’un modèle où la localisation à proximité d’une zone de concentration élevée en facteurs répresseurs induirait une hypersensibilité à l’action de ces facteurs. La chromatine autour des télomères est répressive du fait du recrutement des protéines Sir3 et Sir4 qui sont chargées à l’extrémité du télomère par la protéine Rap1, et qui se propagent ensuite le long du chromosome du fait de l’interaction des protéines Sir3 et Sir4 avec les nucléosomes [40, 41]. Les télomères et les protéines Sir3 et Sir4 sont regroupés en quelques focus localisés en périphérie du noyau (à raison de plusieurs télomères par focus) et l’intégrité de ces focus semble nécessaire à la formation ou au maintien de la structure chromatinienne répressive [42]. Les mêmes protéines Sir3 et Sir4 maintiennent une structure chromatinienne répressive aux cassettes silencieuses du type sexuel où elles sont recrutées par un complexe de protéines interagissant avec des séquences silencers. Chacune de ces cassettes silencieuses est localisée à proximité d’un télomère, sans qu’il y ait une zone ininterrompue de chromatine réprimée par Sir3 et Sir4 entre le télomère et la cassette. La proximité du télomère est nécessaire pour que la cassette soit silencieuse, sauf si la concentration intracellulaire en protéines Sir3 et Sir4 est artificiellement élevée [43]. Cela suggère que le recrutement par les complexes silencer des protéines Sir3 et Sir4 n’est efficace qu’à une certaine concentration de ces protéines, et qu’en situation normale, cette concentration critique n’est assurée qu’à proximité des télomères en périphérie du noyau (Figure 3D). En effet, une cassette dont les silencer ont été inactivés par insertion de sites Gal4 redevient sensible à la répression par Sir3 et Sir4 si elle est recrutée en périphérie du noyau par une chimère Gal4 interagissant avec la membrane nucléaire interne [44]. Ce modèle établit donc une relation mécanistique et directe entre la localisation dans une région du noyau enrichie en facteurs régulateurs et la formation d’une conformation chromatinienne particulière. Forces et contraintes à l’œuvre dans l’organisation et les réorganisations dans le noyau Existe-t-il donc une organisation des chromosomes dans l’espace nucléaire qui détermine, ou contraint, un gène à occuper certaines positions, et détermine ainsi sa sensibilité à certains facteurs régulateurs? On a vu que la proximité linéaire avec les centromères ou les télomères pouvait influencer l’expression génique, mais quels degrés de liberté existe-t-il pour les gènes situés ailleurs dans le chromosome? La chromatine peut diffuser dans le noyau, mais, compte tenu de sa masse moléculaire élevée, elle diffuse beaucoup plus lentement que les protéines isolées [ 45, 46]. Cette mobilité est généralement limitée, car les mouvements d’un segment de chromatine donné semblent réduits à un espace confiné [ 45– 47] bien que, dans la levure, on ait observé que quelques régions du chromosome étaient animées de mouvements de grande amplitude dépendant d’un processus consommant de l’ATP [ 48]. Il doit donc exister des points d’ancrage distribués de façon inhomogène le long du chromosome qui restreignent les mouvements des segments de chromatine mais la nature de ces ancrages n’est pas encore identifiée. Par ailleurs, il existe une certaine précision dans l’organisation du chromosome en interphase qui semble conserver une partie de l’organisation existant dans le chromosome métaphasique [ 49, 50]. Finalement, l’organisation en territoires chromosomiques individualisés implique l’existence de contraintes qui maintiennent ensemble les segments chromatiniens d’un même chromosome [ 24]. Quelles peuvent être ces contraintes et ces points d’ancrage? D’abord, il y a l’ancrage à la périphérie de l’hétérochromatine centromérique et des télomères [ 51]. Puis, il semble qu’on puisse discerner un certain nucléosquelette à l’intérieur du noyau: les lamines ne sont pas limitées au réseau associé à la membrane nucléaire interne, et des filaments de lamine plongent aussi à l’intérieur [ 52]. De plus, de l’actine est présente dans le noyau [ 53]. Toutefois, même si la chromatine et le nucléosquelette interagissent, rien ne prouve que ce nucléosquelette organise l’architecture des chromosomes, et il semble bien plus probable que ce soit les chromosomes eux-mêmes qui portent les déterminants organisateurs de leur architecture. Des interactions entre segments de chromosomes pourraient ainsi contribuer à la structuration de l’espace nucléaire. Particulièrement, selon le bon vieil adage « qui se ressemble, s’assemble » un certain nombre de protéines interagissant avec plusieurs sites chromosomiques semblent capables d’associer ces sites entre eux. C’est le cas de la protéine Ikaros qui, comme nous l’indiquions, interagit à la fois avec des gènes qu’elle réprime et avec les satellites centromériques, et associe les segments chromosomiques correspondants [35, 37]. Dans les cellules diploïdes interphasiques de drosophile, les protéines Su(Hw) et mod(mdg4) responsables de l’activité de l’insulateur du transposon Gypsy sont localisées en quelques dizaines de focus en périphérie du noyau [54]. Le nombre de ces focus étant très inférieur au nombre de sites observés sur les chromosomes polytènes, il est probable que chacun regroupe plusieurs locus cibles de Su(Hw). En effet, il a été montré que deux régions chromosomiques éloignées d’environ 3 Mb sur le même chromosome co-localisent dans un focus Su(Hw)/Mod(mdg4) lorsqu’elles contiennent chacune une insertion Gypsy [54]. Bien que ce ne soit pas aussi clairement démontré, certaines données suggèrent que les protéines du groupe Polycomb (Pc-G), qui ont une fonction répressive, pourraient aussi organiser des interactions entre segments chromosomiques. L’hypothèse d’interactions physiques entre les cibles de Pc-G est étayée par le rôle de ces protéines dans les phénomènes de co-suppression [57] et de homing des éléments P [58]. Dans les cellules diploïdes de drosophile et chez les mammifères, les protéines Pc-G sont distribuées en focus qui pourraient peut-être correspondre au regroupement de plusieurs locus cibles [55, 56]. Finalement, la formation d’un nucléole unique à partir de plusieurs clusters de gènes ribosomiques portés par différents chromosomes implique un repositionnement de ces chromosomes sans que l’on sache encore ce qui le rend possible [59]. La distribution de certaines séquences cibles de facteur régulateurs le long du chromosome pourrait donc participer à la structuration globale du territoire chromosomique et ces facteurs ainsi influencer dans une certaine mesure l’activité d’autres gènes qui ne sont pas des cibles directes. |
On sait depuis longtemps que le noyau est compartimenté en différentes zones d’activité, mais il apparaît clairement maintenant que cette compartimentation est très dynamique car les facteurs constituant chaque compartiment s’équilibrent entre un pool nucléoplasmique libre et de larges complexes macromoléculaires. Certains de ces facteurs sont concentrés dans certaines zones même s’il semble probable que les zones de concentrations les plus élevées ne soient pas nécessairement les zones où ces facteurs sont en activité, et correspondent plutôt à des zones de stockage. On peut supposer que le regroupement de facteurs participant à des étapes successives d’un processus comme l’expression génétique dans des espaces topologiques communs améliorerait l’efficacité du système et il semble que certains facteurs régulateurs puissent favoriser de tels regroupements. Les facteurs régulateurs de l’expression génétique interagissent avec leurs gènes cibles de façon extrêmement dynamique et induisent des changements de conformation de la chromatine et de comportement des machineries de transcription qui ont généralement des durées de vie plus longues que la durée de vie des interactions qui les ont déclenchés. Toutes ces interactions ne surviennent pas de façon topologiquement aléatoire dans le noyau, et ce dernier n’est clairement pas un sac de molécules dispersées au hasard. La place dans le noyau des chromosomes eux-mêmes, qui sont de gros objets relativement peu mobiles, est la résultante de multiples contraintes encore mal définies, mais qui pourraient résulter des nombreuses interactions qui existent entre les différentes régions de ces chromosomes, et entre ces régions chromosomiques et d’autres structures nucléaires comme la membrane nucléaire interne et éventuellement un nucléosquelette interne dont l’importance reste à établir. Cette compartimenta-tion peut influencer l’expression génétique en augmentant ou en réduisant la sensibilité des gènes à des facteurs régulateurs qui ne sont pas distribués de manière uniforme dans le noyau. Les liens entre la vision moléculaire de l’expression génétique (présentée dans la première partie de cet article) et la vision cellulaire (décrite dans la deuxième partie) commencent à peine à être établis. Cependant, la biologie moléculaire intègre de plus en plus la dimension cellulaire et réciproquement, et il sera nécessaire que cette convergence s’accentue pour que l’on puisse passer d’une vision à deux dimensions à une vision à quatre dimensions, c’est-à-dire d’un schéma linéaire de ’expression génétique: séquence cible -> facteur régul+ateur -> activation de la transcription, à une vision intégrant les dimensions topologiques et cinétiques. L’importance de ces deux dernières dimensions est de plus en plus claire et il faudra certainement pouvoir les intégrer de façon quantitative dans le raisonnement pour comprendre la sélectivité des interactions qui déterminent la précision du contrôle de l’expression génétique chez les eucaryotes supérieurs. Toutefois, il nous reste encore beaucoup à comprendre pour atteindre cet objectif, particulièrement en ce qui concerne les structures chromatiniennes d’ordre supérieur, les repliements déterminant l’architecture des chromosomes interphasiques et les variations de concentration locale des composants fonctionnels des machineries de contrôle de l’expression génétique. Cela devrait nous permettre à terme de délaisser les schémas récapitulatifs remplis de petits cercles de couleur pour commencer à intégrer des chiffres et des équations, peut-être perdre un peu de poésie pour gagner un peu de rigueur. |
Le laboratoire est financé par le Cnrs et l’Association pour la Recherche sur le Cancer. Nous remercions M. Coppey, E.M. Geigl, D. Hernandez, C. Kress, et A. Teichert pour une lecture critique du manuscrit.
|
Footnotes |
1. Lemon B, Tjian R. Orchestrated response: a symphony of transcription factors for gene control. Genes Dev 2000; 14: 2551–69. 2. Glass CK, Rosenfeld MG. The coregulator exchange in transcriptional functions of nuclear receptors. Genes Dev 2000; 14: 121–41. 3. Grange T, Bertrand E, Espinas ML, et al. In vivo footprinting of the interaction of proteins with DNA and RNA. Methods 1997; 11: 151–63. 4. Orlando V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trends Biochem Sci 2000; 25: 99–104. 5. Cosma MP, Tanaka T, Nasmyth K. Ordered recruitment of transcription and chromatin remodeling factors to a cell cycle- and developmentally regulated promoter. Cell 1999; 97: 299–311. 6. Krebs JE, Fry CJ, Samuels ML, Peterson CL. Global role for chromatin remodeling enzymes in mitotic gene expression. Cell 2000; 102: 587–98. 7. Agalioti T, Lomvardas S, Parekh B, Yie J, Maniatis T, Thanos D. Ordered recruitment of chromatin modifying and general transcription factors to the IFN-beta promoter. Cell 2000; 103: 667–78. 8. Hassan AH, Neely KE, Workman JL. Histone acetyltransferase complexes stabilize SWI/SNF binding to promoter nucleosomes. Cell 2001; 104: 817–27. 9. Chen H, Lin RJ, Xie W, Wilpitz D, Evans RM. Regulation of hormone-induced histone hyperacetylation and gene activation via acetylation of an acetylase. Cell 1999; 98: 675–86. 10. Shang Y, Hu X, DiRenzo J,Lazar MA, Brown M. Cofactor dynamics and sufficiency in estrogen receptor-regulatedtranscription. Cell 2000;103: 843–52. 11. Grange T, Cappabianca L, Flavin M, Sassi H, Thomassin H. In vivo analysis of the model tyrosine aminotransferase gene reveals multiple sequential steps in glucocorticoid receptor action. Oncogene 2001; 20: 3028–38. 12. Rigaud G, Roux J, Pictet R, Grange T. In vivo footprinting of rat TAT gene: dynamic interplay between the glucocorticoid receptor and a liver-specific factor. Cell 1991; 67: 977–86. 13. Thomassin H, Flavin M, Espinas ML, Grange T. Glucocorticoid-induced DNA demethylation and gene memory during development. EMBO J 2001; 20: 1974–83. 14. Misteli T. Protein dynamics: implications for nuclear architecture and gene expression. Science 2001; 291: 843–7. 15. Phair RD, Misteli T. High mobility of proteins in the mammalian cell nucleus. Nature 2000; 404: 604–9. 16. Kimura H, Cook PR. Kinetics of core histones in living human cells: little exchange of H3 and H4 and some rapid exchange of H2B. J Cell Biol 2001; 153: 1341–53. 17. Misteli T, Gunjan A, Hock R, Bustin M, Brown DT. Dynamic binding of histone H1 to chromatin in living cells. Nature 2000; 408: 877–81. 18. Tumbar T, Sudlow G, Belmont AS. Large-scale chromatin unfolding and remodeling induced by VP16 acidic activation domain. J Cell Biol 1999; 145: 1341–54. 19. Tumbar T, Belmont AS. Interphase movements of a DNA chromosome region modulated by VP16 transcriptional activator. Nat Cell Biol 2001; 3: 134–9. 20. McNally JG, Muller WG, Walker D, Wolford R, Hager GL. The glucocorticoid receptor: rapid exchange with regulatory sites in living cells. Science 2000; 287: 1262–5. 21. Muller WG, Walker D, Hager GL, McNally JG. Large-scale chromatin decondensation and recondensation regulated by transcription from a natural promoter. J Cell Biol 2001; 154: 33–48. 22. Stenoien DL, Nye AC, Mancini MG, et al. Ligand mediated assembly and real-time cellular dynamics of estrogen receptor alpha-coactivator complexes in living cells. Mol Cell Biol 2001; 21: 4404–12. 23. Lewis JD, Tollervey D. Like attracts like: getting RNA processing together in the nucleus. Science 2000; 288: 1385–9. 24. Cremer T, CremerC. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet 2001; 2: 292–301. 25. Francastel C, Schubeler D, Martin DI, Groudine M. Nuclear compartmentalization and gene activity. Nat Rev Mol Cell Biol 2000; 1: 137–43. 26. Croft JA, Bridger JM, Boyle S, Perry P, Teague P, Bickmore WA. Differences in the localization and morphology of chromosomes in the human nucleus. J Cell Biol 1999; 145: 1119–31. 27. Verschure PJ, van Der Kraan I, Manders EM, van Driel R. Spatial relationship between transcription sites and chromosome territories. J Cell Biol 1999; 147: 13–24. 28. Dietzel S, Schiebel K, Little G, et al. The 3D positioning of ANT2 and ANT3 genes within female X chromosome territories correlates with gene activity. Exp Cell Res 1999; 252: 363–75. 29. Volpi EV, Chevret E, Jones T, et al. Largescalechromatin organization of the majorhisto compatibility complex and other regions of human chromosome 6 and its response to interferon in interphase nuclei. J Cell Sci 2000; 113: 1565–76. 30. Li G, Sudlow G, Belmont AS. Interphase cell cycle dynamics of a latereplicating, heterochromatic homogeneously staining region: precise choreography of condensation/decondensation and nuclear positioning. J Cell Biol 1998; 140: 975–89. 31. Orphanides G, Reinberg D. RNA polymerase II elongation through chromatin. Nature 2000; 407: 471–5. 32. Csink AK, Henikoff S. Genetic modification of heterochromatic association and nuclear organization in Drosophila. Nature 1996; 381: 529–31. 33. Dernburg AF, Broman KW, Fung JC, et al. Perturbation of nuclear architecture by long-distance chromosome interactions. Cell 1996; 85 : 745–59. 34. Schubeler D, Francastel C, Cimbora DM, Reik A, Martin DI, Groudine M. Nuclear localization and histone acetylation: a pathway for chromatin opening and transcriptional activation of the human beta-globin locus. Genes Dev 2000; 14: 940–50. 35. Brown KE, Guest SS, Smale ST, Hahm K, Merkenschlage M, Fisher AG. Association otranscriptionally silent genes with Ikaros complexes at centromeric heterochromatin. Cell 1997; 91: 845–54. 36. Brown KE, Baxter J, Graf D, Merkenschlager M, Fisher AG. Dynamic repositioning of genes in the nucleus of lymphocytes preparing for cell division. Mol Cell 1999; 3: 207–17. 37. Cobb BS, Morales-Alcelay S, Kleiger G, Brown KE, Fisher AG, Smale ST.Targeting of Ikaros to pericentromeric heterochromatin by direct DNA binding. Genes Dev 2000; 14: 2146–60. 38. Skok JA, Brown KE, Azuara V, et al. Nonequivalent nuclear location of immunoglobulin alleles in B lymphocytes. Nat Immunol 2001; 2: 848–54. 39. Lundgren M, Chow CM, Sabbattini P, Georgiou A, Minaee S, Dillon N. Transcription factor dosage affects changes in higher order chromatin structure associated with activation of a heterochromatic gene. Cell 2000; 103: 733–43. 40. Cockell M, Gasser SM. Nuclear compartments and gene regulation. Curr Opin Genet Dev 1999; 9: 199–205. 41. Grunstein M. Molecular model for telomeric heterochromatin in yeast. Curr Opin Cell Biol 1997; 9: 383–7. 42. Laroche T, Martin SG, Gotta M, et al. Mutation of yeast Ku genes disrupts the subnuclear organization of telomeres. Curr Biol 1998; 8: 653–6. 43. Maillet L, Boscheron C, Gotta M, Marcand S, Gilson E, Gasser SM. Evidence for silencing compartments within the yeast nucleus: a role for telomere proximity and Sir protein concentration in silencer-mediated repression. Genes Dev 1996; 10: 1796–811. 44. Andrulis ED, Neiman AM, Zappulla DC, Sternglanz R. Perinuclear localization of chromatin facilitates transcriptional silencing. Nature 1998; 394: 592–5. 45. Marshall WF, Straight A, Marko JF, et al. Interphase chromosomes undergo constrained diffusional motion in living cells. Curr Biol 1997; 7: 930–9. 46. Zink D, Cremer T. Cell nucleus: chromosome dynamics in nuclei of living cells. Curr Biol 1998; 8: R321–4. 47. Vazquez J, Belmont AS, Sedat JW. Multiple regimes of constrained chromosome motion are regulated in the interphase Drosophila nucleus. Curr Biol 2001; 11: 1227–39. 48. Heun P, Laroche T, Shimada K, Furrer P, Gasser SM. Chromosome dynamics in the yeast interphase nucleus. Science 2001; 294: 2181–6. 49. Sadoni N, Langer S, Fauth C, et al. Nuclear organization of mammalian genomes. Polar chromosome territories build up functionally distinct higher order compartments. J Cell Biol 1999; 146: 1211–26. 50. Manders EM, Kimura H, Cook PR. Direct imaging of DNA in living cells reveals the dynamics of chromosome formation. J Cell Biol 1999; 144: 813–21. 51. Marshall WF, Fung JC, Sedat JW. Deconstructing the nucleus: global architecture from local interactions. Curr Opin Genet Dev 1997; 7: 259–63. 52. Moir RD, Yoon M, Khuon S, Goldman RD. Nuclear lamins A and B1: different pathways of assembly during nuclear envelope formation in living cells. J Cell Biol 2000; 151: 1155–68. 53. Gounon P, Karsenti E. Involvement of contractile proteins in the changes in consistency of oocyte nucleoplasm of the newt Pleurodeles waltlii. J Cell Biol 1981; 88: 410–21. 54. Gerasimova TI, Byrd K, Corces VG. A chromatin insulator determines the nuclear localization of DNA. Mol Cell 2000; 6: 1025–35. 55. Messmer S, Franke A, Paro R. Analysis of the functional role of the Polycomb chromo domain in Drosophila melanogaster. Genes Dev 1992; 6: 1241–54. 56. Alkema MJ, Bronk M, Verhoeven E, et al.Identification of Bmi1-interacting proteins as constituents of a multimeric mammalian polycomb complex. Genes Dev 1997; 11: 226–40. 57. Pal-Bhadra M, Bhadra U, Birchler JA. Cosuppression in Drosophila: gene silencing of alcohol dehydrogenase by white-Adh transgenes is Polycomb dependent. Cell 1997; 90: 479–90. 58. Fauvarque MO, Dura JM. Polyhomeotic regulatory sequences induce developmental regulator-dependent variegation and targeted P-element insertions in Drosophila. Genes Dev 1993; 7: 1508–20. 59. Sullivan GJ, Bridger JM, Cuthbert A P, Newbold RF, Bickmore WA, McStayB. Human acrocentric chromosomes with transcriptionally silent nucleolar organizer regions associate with nucleoli. EMBO J 2001; 20: 2867–74. 60. Cappabianca L, Thomassin H, Pictet R, Grange T. Genomic footprinting using nucleases. Meth Mol Biol 1999; 119: 427–42. 61. Mahy NL, Perry PE, Gilchrist S, Baldock RA, Bickmore WA. Spatial organization of active and inactive genes and noncoding DNA within chromosome territories. J Cell Biol 2002; 157: 579–89. |