| |
| Med Sci (Paris). 2004 February; 20(2): 207–212. Published online 2004 February 15. doi: 10.1051/medsci/2004202207.Structure terminale des chromosomes : le « capuchon télomérique » Catherine LeBel and Raymund J. Wellinger* Département de microbiologie et d’infectiologie, Faculté de médecine, Université de Sherbrooke, 3001, 12e Avenue Nord, Sherbrooke, Québec, J1H 5N4, Canada |
Les études du généticien H.J. Muller sur les chromosomes de la drosophile ont donné naissance au concept de télomère en 1938. H.J. Muller observa que des délétions ou des inversions induites par irradiation aux rayons X de l’ensemble du génome étaient quasiment indétectables dans les régions terminales des chromosomes [
1]. Il en conclut que les extrémités des chromosomes, qu’il nomma télomères, devaient posséder une structure particulière capable de stabiliser les chromosomes. À la même époque, B. McClintock démontra que des chromosomes du maïs ayant subi des cassures double brin étaient capables de fusionner entre eux, contrairement à leurs extrémités qui demeuraient stables [
2]. Le modèle le plus récent suggère que les télomères agissent comme des structures qui protègent les chromosomes d’éventuelles fusions de leurs extrémités, de l’intervention de la machinerie de réparation (qui les considèrerait comme des cassures double brin) et de la dégradation par des exonucléases. Ces structures protectrices, que nous proposons d’appeler « capuchons télomériques », sont constituées par l’association d’ADN télomérique et de protéines spécifiques. Ce nouveau concept de capuchon protecteur des chromosomes est maintenant établi dans la communauté scientifique. Ce que l’on connaissait de la machinerie de réplication conventionnelle, incapable de synthétiser l’extrémité de molécules d’ADN linéaires comme les chromosomes eucaryotes, laissait supposer que les télomères devaient subir des pertes de leurs séquences terminales au fil des cycles de réplication. Chez les humains, on observe effectivement un raccourcissement des chromosomes dans la plupart des cellules somatiques au fil des années, et l’hypothèse selon laquelle les télomères joueraient un rôle déterminant dans la sénescence cellulaire a été proposée. Cependant, il existe une enzyme spécifique, la télomérase, d’abord identifiée chez le cilié Tetrahymena en 1985, qui peut prévenir ce raccourcissement naturel des chromosomes en ajoutant des répétitions télomériques à leurs extrémités [
3]. Cette enzyme est absente de la plupart des cellules humaines somatiques normales. En revanche, chez l’homme, elle est présente et activée dans les cellules germinales, dans la plupart des lignées de cellules transformées et dans 90 % des cellules cancéreuses primaires [
4]. Ce lien entre activation de la télomérase et carcinogenèse a suscité un grand enthousiasme dans la communauté scientifique, suggérant que des thérapies anticancéreuses fondées sur l’inhibition de la télomérase pourraient être applicables à de nombreux cancers (→). (→) m/s 2001, n° 10, p. 1076 L’intérêt porté aux télomérases a longtemps rejeté dans l’ombre les études sur la structure des télomères et du capuchon télomérique, et les mécanismes moléculaires de protection des extrémités des chromosomes sont de ce fait encore mal connus. Le séquençage de l’ADN télomérique chez différentes espèces montre que la séquence de l’ADN présent aux extrémités des chromosomes est relativement bien conservée (Figure 1A), ce qui est en faveur de son importance fonctionnelle. Dans la plupart des cas, l’ADN télomérique est constitué de courtes séquences répétées en tandem [
5]. Le nombre de répétitions varie selon les espèces, selon les individus et même entre les différents chromosomes d’une même cellule. Dans tous les organismes étudiés, le brin riche en guanines (G-riche), correspondant à l’extrémité 3’, est plus long que le brin complémentaire riche en cytosines (C-riche), créant ainsi une extension simple brin en 3’ du chromosome (Figure 1B). La drosophile, qui a servi à l’élaboration même du concept de télomère, est paradoxalement une des espèces qui font exception à ces règles générales de constitution des télomères. Elle possède en effet aux extrémités de ses chromosomes un nombre multiple de copies de rétrotransposons spécifiques des télomères. Les télomères de drosophile et les télomères réguliers d’autres organismes, dont plusieurs arthropodes, dériveraient cependant des mêmes télomères ancestraux. Les séquences télomériques « atypiques » de la drosophile résulteraient de modifications des séquences télomériques communes plutôt que de la création de novo d’une nouvelle structure télomérique.
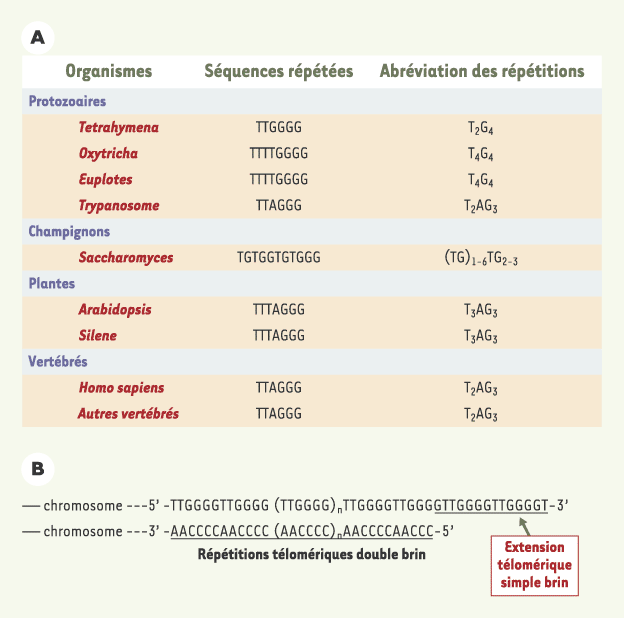
 | Figure 1.
Structure primaire des télomères.
A. Séquences télomériques des chromosomes de différents organismes [ 5,
17]. B. Structure primaire de l’ADN d’un télomère de l’organisme modèle Tetrahymena thermophila. |
L’extension simple brin serait essentielle pour assurer la fonction de capuchon protecteur des télomères au niveau des chromosomes et, par conséquent, de l’ensemble du génome, comme cela avait été proposé il y a 60 ans [
6,
7]. Dans cet article sont exposés les récents développements concernant la structure terminale de l’ADN chromosomique chez différents organismes. |
La première séquence télomérique déterminée fut celle du protozoaire cilié Tetrahymena thermophila [
8]. Les télomères de cet organisme comportent 50 jusqu’à 70 répétitions de la séquence TTGGGG/CCCCAA (T2G4/C4A2), et le brin G-riche correspond à l’extrémité 3’. La taille de l’extension 3’ simple brin est de l’ordre de 12 à 16 nucléotides (Figure 1B). Chez Tetrahymena, deux types de noyaux coexistent : le micronoyau et le macronoyau. Le micronoyau, diploïde et transcriptionnellement inactif, sert uniquement à la transmission de l’information génétique au cours de la reproduction. Le macronoyau, issu du micronoyau, est constitué de chromosomes fragmentés transcriptionnellement actifs. Diverses expériences suggèrent que des extensions G-riches sont présentes sur tous les télomères, aux deux extrémités des chromosomes macronucléaires [
9]. Ces extensions sont détectées durant tout le cycle cellulaire [
10]. La taille exacte des extensions, ainsi que leur séquence, sont contrôlées très strictement chez les ciliés. La majorité des extensions se termine par la séquence 5’-TTGGGGT-3’ et plus de 80 % de ces extensions ont 14-15 ou 20-21 nucléotides [10]. Cependant, leur taille peut varier lorsque les cellules sont soumises à certaines restrictions nutritionnelles ou lorsqu’elles sortent du cycle cellulaire. |
Levures : petites variations sur le thème Chez la levure Saccharomyces cerevisiae, il existe une certaine dynamique dans la régulation de la taille des extensions simple brin selon les phases du cycle cellulaire. Nos résultats récents suggèrent que, comme chez les ciliés, les télomères de levure possèdent une extension de 12-15 bases durant la plus grande partie du cycle cellulaire (données non publiées), à l’exception de la fin de la phase S où ils acquièrent, de façon transitoire, des extensions G-riches de plus de 30 nucléotides [
11]. Ces longues extensions sont présentes sur tous les télomères après réplication de l’ADN, c’est-à-dire aux deux extrémités de chaque chromosome [
12]. Cependant, contrairement à ce qui est observé chez les ciliés, la séquence 3’ terminale de la levure peut être différente pour chaque télomère, et, bien qu’elle corresponde toujours à la séquence consensus TG1-3, le nombre de guanines présentes dans chacune des répétitions peut varier [
13]. La dynamique des extensions simple brin au cours du cycle cellulaire et la présence d’une séquence spécifique à l’extrémité des chromosomes semblent donc être des événements moléculaires variables selon les espèces. Néanmoins, l’ensemble des résultats obtenus chez les ciliés et chez la levure indiquent que la protection des extrémités des chromosomes est assurée par une structure terminale définie, comprenant une courte extension simple brin (12-20 nucléotides) et des protéines spécifiques associées, l’ensemble formant le capuchon télomérique. |
Cellules de mammifères : la boucle terminale Dans le cas des cellules de mammifères, les séquences télomériques et les extensions simple brin sont plus longues que pour les organismes décrits ci-dessus. En effet, les télomères des cellules humaines possèdent une extension G-riche d’environ 150 à 200 bases, qui serait présente elle aussi durant toutes les phases du cycle cellulaire [
14–
16]. La présence de ces extensions aux deux extrémités d’un même chromosome fait cependant l’objet d’une controverse. En utilisant une technique appelée PENT (primer extension/nick translation), V.L. Makarov et al. ont montré la présence d’extensions G-riches sur plus de 80 % des extrémités des chromosomes humains [14]. Ce résultat est en désaccord avec le modèle de réplication conventionnelle (Figure 2), qui prédit que seulement 50 % des extrémités - celles produites par la réplication du brin retardé - devraient posséder une extension 3’ simple brin, après élimination de la dernière amorce ARN du dernier fragment d’Okazaki (hybride ARN-ADN synthétisé par la polymérase α/primase lors de la synthèse du brin retardé). Les extensions simple brin détectées aux extrémités du brin avancé pourraient résulter soit de l’action d’exonucléases, soit d’une activité télomérase. De telles activités exonucléases ont été mises en évidence chez la levure S. cerevisiae [12]. Concernant l’activité télomérase, il n’est pas exclu que cette enzyme, dont le moment d’action précis n’est pas encore bien défini, puisse agir après la réplication, à l’extrémité du brin avancé, pour créer une extension simple brin en utilisant comme substrat une très courte extension laissée par la machinerie de réplication ou bien produite par des exonucléases.
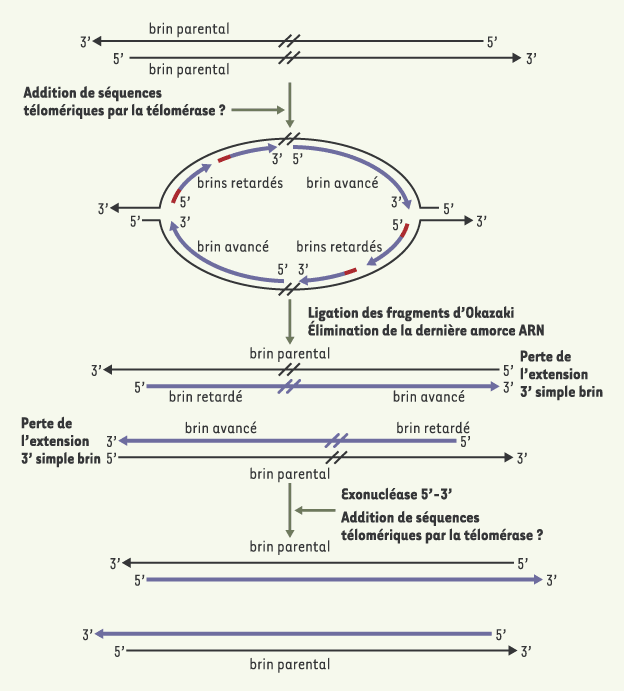
 | Figure 2.
Le problème du brin avancé lors de la réplication des télomères. L’ADN parental (en noir) est répliqué (ADN nouvellement synthétisé en bleu) par un mécanisme semi-conservatif. Les flèches indiquent la polarité de l’ADN. La réplication semi-conservative de l’ADN ne permet pas de régénérer une extension 3’ simple brin à l’extrémité du brin avancé. En revanche, à l’extrémité du brin retardé, l’élimination de la dernière amorce d’ARN (en rouge) produit une courte extension simple brin. La création du substrat de la télomérase à l’extrémité du brin avancé ne peut se faire que par l’intervention d’une exonucléase, alors que ce substrat est créé par défaut à l’extrémité du brin retardé. L’élongation se produirait, dans les deux cas, par l’intervention de la télomérase, sans que l’on sache actuellement si cet ajout a lieu avant ou après la réplication conventionnelle. |
W.E. Wright et al. ont obtenu chez l’humain des résultats plus en accord avec le modèle de réplication conventionnelle. Selon ces auteurs, les chromosomes humains de fibroblastes pourraient être asymétriques, avec une longue extension simple brin d’un côté et une très courte extension de l’autre [16]. En microscopie électronique, ces auteurs ont évalué la taille des longues extensions simple brin à environ 200 ± 75 nucléotides, mais la taille des extensions courtes demeure inconnue [16]. Des observations similaires ont été rapportées pour les télomères chez certaines plantes [17]. Ces résultats suggèrent que des extensions simple brin sont bien présentes à toutes les extrémités télomériques et que leur présence, plus que leur taille, est essentielle à la protection des chromosomes [7]. Dans ce modèle « linéaire » des extrémités des chromosomes, le capuchon télomérique est formé de l’extension d’ADN simple brin et des protéines spécifiques qui lui sont associées (Figure 3A). Cependant, en 1999, ce modèle a été radicalement modifié. Pour la première fois, des observations en microscopie électronique ont montré que les extrémités des chromosomes de mammifères sont en fait repliées sur elles-mêmes, formant une énorme boucle terminale, appelée t-loop (telomere loop) [
18]. Ces boucles sont créées du fait de l’insertion dans la portion double brin du télomère de l’extrémité 3’ simple brin (Figure 3B). Il y a ainsi déplacement d’une partie de l’ADN télomérique double brin, qui devient simple brin à l’intérieur de la boucle même, formant une D-loop (displacement loop) [18]. La formation de ces structures est permise par la présence des extensions terminales simple brin relativement longues chez les mammifères et par l’association avec certaines protéines télomériques spécifiques. Ces structures terminales permettraient aux cellules de distinguer leurs extrémités chromosomiques des cassures double brin survenant ailleurs dans l’ADN, et de les protéger, entre autres, de la réparation. Le contenu riche en guanine des extensions télomériques pourrait favoriser la formation de structures alternatives, comme des duplex ou des quadruplex de G (G-quartets ou G4-DNA) [5].
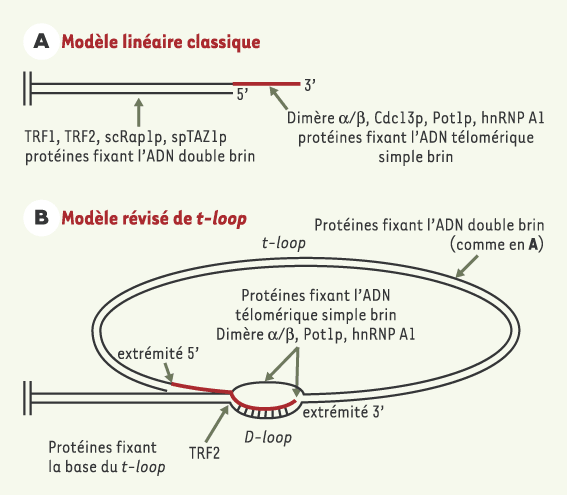
 | Figure 3.
Modèles de structure des télomères.
A. Modèle classique d’une extrémité chromosomique avec une courte extension en 3’ (en rouge) et un complexe protéique protégeant l’ADN chromosomique (modèle linéaire). B. Les télomères des mammifères se terminent par de larges boucles, les t-loops (telomere loops). Le double brin télomérique se replie sur lui-même, l’extension 3’ simple brin s’insère dans la portion double brin et engendre la D-loop (displacement loop). La protéine TRF2 se fixe à la base du D-loop in vitro. Les t-loops, en association avec un complexe protéique terminal, pourraient permettre de différencier les extrémités des chromosomes des cassures double brin dans l’ADN, et éviter ainsi la détection des télomères par la machinerie de réparation des cassures d’ADN. Les régions simple brin du t-loop et les extensions en 3’ sont indiquées en rouge. TRF1, 2 : telomeric repeat binding factor 1, 2 ; scRap1p : protéine télomérique RAP1 de Saccharomyces cerevisiae ; Cdc13p : telomere binding protein Cdc13p ; Pot1p : protection of telomeres 1 ; hnRNPA1 : heterogeneous ribonucleoprotein A1. |
Récemment, en utilisant une technique d’attachement d’oligonucléotides télomériques complémentaires à l’extension G-riche (T-OLA, telomeric-oligonucleotide ligation assay) [
19], le groupe de R. Weinberg a montré qu’il existe une corrélation entre l’érosion des extensions 3’ simple brin et l’entrée en sénescence des cellules humaines [
20]. Cette érosion semble être le résultat direct des divisions cellulaires successives plutôt qu’une conséquence de la sénescence. Cela suggère qu’une altération spécifique de la structure terminale des chromosomes, et plus particulièrement de l’extension G-riche, survient lors de la sénescence. En effet, cette altération, plutôt qu’une diminution globale de la taille des télomères, déclencherait le processus de sénescence cellulaire. |
Retour chez les ciliés : deux modes de protection ? Après la découverte des structures en boucle chez les mammifères, des structures t-loop analogues ont pu être observées aux extrémités des chromosomes micronucléaires d’Oxytricha fallax, qui possèdent de longues extensions permettant la formation de telles boucles terminales. En revanche, les extensions de l’ADN télomérique des chromosomes macronucléaires du même organisme sont trop courtes pour former des t-loops. Dans ce cas, les télomères sont sous forme linéaire simple brin et protégés par des complexes spécifiques d’hétérodimères protéiques [
21]. Ainsi, chez cet organisme, selon l’origine de l’ADN étudié, deux mécanismes différents de protection des chromosomes pourraient être utilisés par une même cellule. Des structures en t-loop ont également été observées chez Trypanosoma brucei, aux deux extrémités des chromosomes [
22]. Comme pour les cellules de mammifères, ces structures sont associées à la présence d’extensions simple brin sur les deux brins d’ADN nouvellement synthétisés. La présence de longues extensions simple brin semble donc essentielle à la création des t-loops dans tous les organismes. |
L’ensemble de ces résultats démontre l’existence de deux arrangements possibles aux extrémités des chromosomes eucaryotes : les t-loops qui requièrent de longues extensions simple brin afin de permettre la formation de boucles d’ADN, et les structures linéaires formées de courtes extensions simple brin protégées par des complexes protéiques spécifiques. Ces deux arrangements ne sont pas nécessairement exclusifs. Récemment, une nouvelle protéine télomérique protectrice a été découverte chez la levure S. pombe et chez l’homme. Cette protéine possède des caractéristiques très similaires à celles des protéines qui protègent les courtes extensions chez les ciliés et chez la levure S. cerevisiae [
23]. Les extensions impliquées dans la formation de la structure t-loop étant relativement longues, il est possible qu’une courte séquence à l’extrémité de ces extensions demeure, dans la boucle, sous forme simple brin non associée au brin complémentaire (Figure 3B). Cette courte extension simple brin pourrait recruter des protéines protectrices conservées chez les eucaryotes, et cette structure pourrait être la condition sine qua non pour la formation du capuchon télomérique. Il est donc possible que la fonction de protection des chromosomes eucaryotes associée au capuchon terminal requiert un complexe nucléoprotéique conservé au cours de l’évolution. La signification biologique et le mécanisme de formation des t-loops sont encore la source de nombreuses interrogations et il n’est pas exclu que les travaux en cours apportent encore des réponses surprenantes à ces questions. |
Les travaux de notre laboratoire sont soutenus par des financements des Instituts de recherche en santé du Canada (IRSC) et de l’Institut national du cancer du Canada (INCC). C.L. était boursière du Conseil de recherches en sciences naturelles et en génie (CRNSG) et R.J.W. est un chercheur national du Fonds de la recherche en santé du Québec (FRSQ).
|
1. Muller HJ. The remaking of chromosomes. The Collecting Net 1938; 13 : 181–95, 198. 2. McClintock B. The behavior in successive nuclear divisions of a chromosome broken at meiosis. Proc Natl Acad Sci USA 1939; 25 : 405–16. 3. Greider CW, Blackburn EH. Identification of a specific telomere terminal transferase activity in Tetrahymena extracts. Cell 1985; 43 : 405–13. 4. Kim NW, Piatyszek MA, Prowse KR, et al. Specific association of human telomerase activity with immortal cells and cancer. Science 1994; 266 : 2011–5. 5. Wellinger RJ, Sen D. The DNA structures at the ends of eukaryotic chromosomes. Eur J Cancer 1997; 33 : 735–49. 6. Sandell LL, Zakian VA. Loss of a yeast telomere: arrest, recovery, and chromosome loss. Cell 1993; 75 : 729–39. 7. Van Steensel B, Smogorzewska A, de Lange T. TRF2 protects human telomeres from end-to-end fusions. Cell 1998; 92 : 401–13. 8. Blackburn EH, Gall JG. A tandemly repeated sequence at the termini of the extrachromosomal ribosomal RNA genes in Tetrahymena. J Mol Biol 1978; 120 : 33–53. 9. Klobutcher LA, Swanton MT, Donini P, Prescott DM. All gene-sized DNA molecules in four species of hypotrichs have the same terminal sequence and an unusual 3’ terminus. Proc Natl Acad Sci USA 1981; 78 : 3015–9. 10. Jacob NK, Skopp R, Price CM. G-overhang dynamics at Tetrahymena telomeres. Embo J 2001; 20 : 4299–308. 11. Wellinger RJ, Wolf AJ, Zakian VA. Saccharomyces telomeres acquire single-strand TG1–3 tails late in S phase. Cell 1993; 72 : 51–60. 12. Wellinger RJ, Éthier K, Labrecque P, Zakian VA. Evidence for a new step in telomere maintenance. Cell 1996; 85 : 423–33. 13. Forstemann K, Hoss M, Lingner J. Telomerase-dependent repeat divergence at the 3’ ends of yeast telomeres. Nucleic Acids Res 2000; 28 : 2690–4. 14. Makarov VL, Hirose Y, Langmore JP. Long G tails at both ends of human chromosomes suggest a C strand degradation mechanism for telomere shortening. Cell 1997; 88 : 657–66. 15. McElligott R, Wellinger RJ. The terminal DNA structure of mammalian chromosomes. Embo J 1997; 16 : 3705–14. 16. Wright WE, Tesmer VM, Huffman KE, Levene SD, Shay JW. Normal human chromosomes have long G-rich telomeric overhangs at one end. Genes Dev 1997; 11 : 2801–9. 17. Riha K, McKnight TD, Fajkus J, Vyskot B, Shippen DE. Analysis of the G-overhang structures on plant telomeres : evidence for two distinct telomere architectures. Plant J 2000; 23 : 633–41. 18. Griffith JD, Comeau L, Rosenfield S, et al. Mammalian telomeres end in a large duplex loop. Cell 1999; 97 : 503–14. 19. Cimino-Reale G, Pascale E, Battiloro E, et al. The length of telomeric G-rich strand 3’-overhang measured by oligonucleotide ligation assay. Nucleic Acids Res 2001; 29 : e35. 20. Stewart SA, Ben-Porath I, Carey VJ, et al. Erosion of the telomeric single-strand overhang at replicative senescence. Nat Genet 2003; 33 : 492–6. 21. Murti KG, Prescott DM. Telomeres of polytene chromosomes in a ciliated protozoan terminate in duplex DNA loops. Proc Natl Acad Sci USA 1999; 96 : 14436–9. 22. Munoz-Jordan JL, Cross GA, de Lange T, Griffith JD. T-loops at trypanosome telomeres. Embo J 2001; 20 : 579–88. 23. Baumann P, Cech TR. Pot1, the putative telomere end-binding protein in fission yeast and humans. Science 2001; 292 : 1171–5. |