| |
| Med Sci (Paris). 2004 April; 20(4): 487–490. Published online 2004 April 15. doi: 10.1051/medsci/2004204487.Chroniques génomiques Coup de tabac sur les puces Bertrand Jordan* Marseille-Génopole, case 901, Parc Scientifique de Luminy, 13288 Marseille Cedex 9, France MeSH keywords: Conception d'appareillage, Analyse de profil d'expression de gènes, Humains, Séquençage par oligonucléotides en batterie, Reproductibilité des résultats |
Une apparente bonne santé Le secteur des puces à ADN fait preuve de dynamisme. Plusieurs fabricants mettent sur le marché des microarrays « génome entier » autorisant la mesure du niveau d’expression de l’ensemble des gènes humains. L’entreprise FeBit, dont nous avons déjà parlé dans ces colonnes [
1], commercialise enfin son instrument-miracle, le Geniom One, automatisant fabrication, hybridation et lecture de réseaux d’oligonucléotides. Enfin, les puces à protéines, après des débuts difficiles, commencent à avoir une existence réelle. Pourtant le développement commercial reste en deçà des prévisions très optimistes d’il y a deux ou trois ans 1, et le marché des microarrays à usage clinique tarde à s’ouvrir. Et, surtout, apparaissent au grand jour des interrogations sur la qualité des données engendrées par les microarrays, notamment sur la cohérence entre les résultats produits par des plates-formes différentes. |
La confrontation des plates-formes L’on s’attendrait a priori à ce que les profils d’expression obtenus à l’aide de réseaux d’oligonucléotides courts (Affymetrix), longs (Agilent), ou de produits de PCR provenant de clones d’ADNc soient très proches ; les efforts déployés par différentes équipes (notamment celle de l’European Bioinformatics Institute) pour constituer des bases de données d’expression reposent à l’évidence sur ce présupposé. Or, des articles récents mettent sérieusement en doute cette cohérence. Le plus radical est sans doute celui de l’équipe de Margaret Cam, responsable de la plate-forme microarrays au National Institute of Diabetes and Digestive and Kidney Disorders dans le cadre du National Institutes of Health (NIH) nord-américain [
2]. Après la publication de deux études montrant d’importantes différences entre les résultats de différents systèmes [
3,
4], Margaret Cam a voulu les comparer dans les meilleures conditions possibles, afin de choisir le plus performant pour son centre de ressources. La comparaison incluait le système Affymetrix (employé par un laboratoire du NIH qui en a une très bonne expérience), des réseaux d’ADNc fournis par Agilent (leurs réseaux d’oligonucléotides n’étant pas encore disponibles à l’époque) et le procédé Codelink d’Amersham (oligonucléotides 30-mères fixés dans une couche d’un gel spécifique). Les deux derniers étaient mis en œuvre, dans le laboratoire du NIH, grâce à des ingénieurs envoyés par ces deux sociétés. Les échantillons fournis étaient identiques, provenant des mêmes préparations d’ARN. |
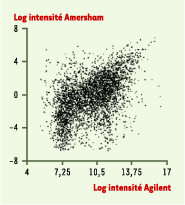
Des résultats catastrophiques Parmi les gènes représentés sur les trois types de puces d’après les indications de leurs fabricants, deux mille (2009 exactement) étaient communs aux trois plates-formes et permettaient donc une comparaison. La conclusion essentielle (et très dérangeante) de l’étude est que les indications données par les trois systèmes divergent presque totalement ! Bien que la reproductibilité technique et biologique de chaque série de mesures soit bonne (coefficient de corrélation de Pearson supérieur à 0,9), la corrélation entre les valeurs obtenues pour le même échantillon par différentes plates-formes plafonne à 0,5 dans tous les cas… Les diagrammes correspondants sont franchement mauvais. La Figure 1 montre la confrontation des données Codelink (Amersham) avec celles d’Agilent : la corrélation est à peine perceptible, et l’image est très différente de la diagonale attendue.
 | Figure 1.
Comparaison, en coordonnées logarithmiques normalisées, des valeurs mesurées par le système Codelink Amersham et la plate-forme Agilent pour l’expression des mêmes 2 009 gènes dans le même échantillon. Le résultat attendu serait un nuage de points très proches d’une diagonale : on en est loin… (adapté de [ 2]). |
Mais après tout, le but de l’exercice n’est pas tant de mesurer des niveaux d’expression que de repérer des gènes différentiellement exprimés, et l’on pourrait espérer que, malgré les différences individuelles, une certaine cohérence serait retrouvée à ce niveau-là. Hélas, ce n’est pas le cas. Selon les données obtenues avec la plate-forme Affymetrix, 34 gènes sont différentiellement exprimés entre les deux conditions examinées (culture avec ou sans sérum) ; pour Agilent, 67, et pour Amersham, 117. Mais seuls quatre d’entre eux sont communs aux trois listes ! Les comparaisons deux à deux ne sont guère plus probantes : cinq gènes communs entre Affymetrix (sur 34) et Amersham (sur 117), 23 entre Agilent (67) et Amersham… Au moins les gènes différentiels communs sont-ils régulés dans le même sens selon les différents systèmes. Mince consolation… Ces résultats sont donc extrêmement inquiétants, d’autant qu’ils ont été obtenus dans d’excellentes conditions. Les échantillons de départ étaient strictement identiques, alors que l’article de W.P. Kuo et al. [4] comparait a posteriori des données obtenues dans deux laboratoires sur les mêmes lignées - mais avec des préparations différentes et de nombreuses variations techniques. Les plates-formes, de plus, étaient mises en œuvre par des personnels bien expérimentés, et la comparaison des performances constituait l’objectif principal du travail. Les données obtenues ont visiblement surpris Margaret Cam, qui s’en explique dans une interview parue dans l’hebdomadaire professionnel BioArray News 2. Après quelques hésitations, les intéressés ont finalement décidé de publier leur étude, qui fait à juste titre un certain bruit dans le petit monde des puces à ADN : les conclusions parlent d’elles-mêmes, et elles sont sévères. |
Certaines des discordances observées pourraient s’expliquer par la détection de formes alternatives des transcrits. Selon la position des oligonucléotides (pour Affymetrix et Codelink) ou des produits de PCR (pour Agilent) par rapport aux exons du gène considéré, l’on pourrait en effet mesurer l’abondance de produits de transcription différents issus du même gène. Les auteurs n’ont pas pu examiner sérieusement cette hypothèse, car ils n’ont pas eu accès à l’ensemble des séquences des produits présents sur les microarrays (sauf pour la puce Affymetrix)… Par ailleurs, les calculs impliqués par l’acquisition des données, particulièrement complexes dans le cas d’Affymetrix, puisque chaque mesure repose sur l’analyse d’une vingtaine de plots différents, pourraient aussi jouer un rôle dans ces différences, mais la réinterprétation des valeurs brutes par deux autres logiciels n’a pas donné une meilleure cohérence. Que cela plaise ou non, les différences constatées indiquent que deux au moins des systèmes donnent des résultats inexacts… L’on ne peut à cet égard que regretter l’absence du « juge de paix », c’est-à-dire de la vérification par PCR quantitative des différentiels indiqués. Ces contrôles sont annoncés pour un prochain article ; bien que je ne sois pas un chaud partisan d’Affymetrix 3, [3], je ne serais pas étonné que cette firme soit la gagnante de la comparaison. Le système Codelink commercialisé par Amersham n’a pas fait l’objet de beaucoup de publications, et l’emploi d’un unique oligonucléotide court pour la mesure d’expression laisse un peu rêveur, même si le support et la technique de dépôt sont particulièrement travaillés. Quant aux microarrays Agilent, ils n’ont été commercialisés sous cette forme (produits de PCR à partir de clones d’ADNc) que de manière assez transitoire, avant que l’entreprise ne mette l’essentiel de ses efforts dans des réseaux fondés sur l’emploi d’oligonucléotides longs. On confronte donc ici une technologie a priori bien établie avec des méthodes soit assez nouvelles (Codelink) soit « instables » (au sens de leur pérennité) pour Agilent. Il aurait été intéressant de faire la comparaison avec des microarrays à base d’ADNc bien éprouvés, comme ceux d’un centre de ressources académique ayant une bonne expérience et de sérieuses références. Quoi qu’il en soit, Amersham, comme Agilent (et Affymetrix), vendent leurs puces en assurant qu’elles permettent des mesures d’excellente qualité pour les gènes indiqués dans leurs spécifications ; or, étant donné les discordances enregistrées, on est obligé de conclure que deux au moins des produits ne sont pas à la hauteur de ces assurances ! |
Cet article n’est pas le premier à relever de sérieuses incohérences entre les résultats fournis par les différentes plates-formes de microarrays. Les études de W.P. Kuo et al. [4] et celles R. Kothapalli et al. [
5], entre autres, arrivaient déjà à des conclusions similaires. Il restait cependant des échappatoires, comme par exemple le fait que les échantillons analysés ne soient pas strictement les mêmes, échappatoires qui n’existent pas dans le travail de l’équipe de Margaret Cam. Le titre d’un article récent, émanant de l’équipe de John Weinstein (National Cancer Institute, NIH) [
6], est plus positif : « Comparing cDNA and oligonucleotide array data: concordance of gene expression across platforms for the NCI-60 cancer cells. » Les plates-formes comparées sont le système Affymetrix (puce HU6800 représentant 6800 gènes humains) et des microarrays comportant près de 10 000 produits de PCR d’ADNc établis et validés au NIH : cette fois l’on compare effectivement deux approches bien éprouvées. L’exercice est large, portant sur l’ensemble des 60 lignées cellulaires du NCI 4. Les auteurs, disposant de l’ensemble des séquences, ont procédé à une étude informatique détaillée et ont finalement défini 1 493 gènes représentés sur les deux supports (alors qu’il y avait a priori plus de 3 000 gènes en commun). Ils éliminent ainsi l’essentiel des différences qui pourraient être dues aux événements d’épissage alternatif. Pour quelques gènes, une très bonne corrélation sur l’ensemble des 60 lignées (coefficient de Pearson supérieur à 0,9) est observée entre les deux plates-formes ; mais, pour la majorité, elle se situe plutôt autour de 0,6. Les regroupements des lignées cellulaires et, dans une moindre mesure, des gènes ont été établis à partir des deux séries de mesures (ADNc et puces à oligonucléotides) : ils sont similaires quoique non identiques. Au total, on observe effectivement une certaine cohérence entre les résultats lorsque l’on analyse avec soin un grand ensemble de mesures, mais on est tout de même loin de la concordance quasiment totale espérée a priori (et suggérée par le titre de l’article). |
Une note positive pour terminer… Une dernière publication [
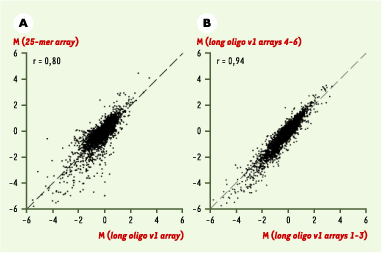
7], datant de l’été 2003, donne (enfin !) un résultat satisfaisant. Elle émane d’une équipe de l’université de San Francisco qui a comparé pour deux échantillons d’ARN humain les résultats obtenus avec le système Affymetrix (GeneChip U95A v2) et ceux fournis par des réseaux constitués d’oligonucléotides 70-mères (un par gène), vendus par l’entreprise Operon et déposés sur lame de verre par les auteurs, qui ont une bonne expérience de cette fabrication. En fait, deux versions de la collection Operon ont été utilisées ; toutes les informations de séquence étant disponibles, il s’avérait que 7344 gènes étaient communs aux trois systèmes, et c’est sur eux qu’a porté la comparaison. Les auteurs ont principalement confronté les rapports des signaux donnés par un échantillon d’ARN d’une lignée cellulaire humaine et ceux provenant d’un pool d’ARN vendu par Stratagene, et ont effectué les moyennes de six mesures pour chaque combinaison. Et, cette fois, les données obtenues montrent une corrélation d’environ 0,8, qui monte à 0,89 si l’on exclut les points correspondant à de faibles intensités (la reproductibilité, elle, est de 0,94) (Figure 2). Ce sont donc de bons résultats, et cet article rassure : dans des expériences soigneusement menées, deux plates-formes différentes donnent bel et bien des résultats cohérents. A contrario, il doit y avoir quelque chose de pourri au royaume de certains fabricants…
 | Figure 2.
A. Comparaison (pour 7 344 gènes) entre les données Affymetrix et celles obtenues avec un réseau à oligonucléotides longs. Notons que l’on compare ici les logarithmes des rapports d’intensité entre deux échantillons (0 signifie une absence d’expression différentielle) et que les données sont les moyennes de six déterminations. B. Reproductibilité des mesures comparant les moyennes des valeurs obtenues avec trois réseaux à oligonucléotides longs (1 à 3) et les trois autres (4 à 6). La corrélation est, comme attendu, meilleure que celle que l’on observe entre plates-formes différentes (adapté de [ 7]). |
|
Je ne pense bien sûr pas qu’il faille abandonner l’emploi des microarrays et l’analyse des profils d’expression - cela reste l’un des seuls outils dont nous disposions pour faire véritablement de la « biologie à grande échelle ». Mais les résultats décevants de ces comparaisons doivent inciter à la prudence, et être l’occasion pour certains industriels d’améliorer à la fois la qualité de leurs produits et les informations fournies aux utilisateurs. Les discordances observées proviennent probablement du choix d’oligonucléotides peu performants du point de vue de la sensibilité ou de la spécificité, et d’approximations ou d’erreurs dans l’étiquetage des gènes censés être reconnus. De plus, certains détectent sans doute des variants d’épissage différents d’une plate-forme à l’autre. Cela étant, la course à la puce « génome entier » mentionnée au début de cet article suscite une certaine inquiétude : est-on sûr que tous ces oligonucléotides reconnaissent réellement le gène qu’ils sont censés détecter, et lui seul ? Ont-ils vraiment été testés un à un, l’expérience montrant qu’aucun programme ne prédit leur performance de manière totalement fiable ? Il ne servira à rien d’archiver soigneusement les données d’expression si ces résultats se rapportent à des transcrits mal définis et différents d’une série d’expériences à l’autre… La validation des résultats, l’analyse statistique et la comparaison systématique des informations obtenues avec différentes plates-formes - en un mot, la « qualité » - doivent rester (ou parfois devenir) un souci constant afin de permettre à cet extraordinaire outil qu’est la puce à ADN de tenir ses promesses. La connaissance des séquences déposées ou synthétisées sur les puces 5, la confirmation des résultats les plus importants par PCR quantitative et, pour les études cliniques, la validation des signatures d’expression obtenues par un test portant sur une série totalement indépendante d’échantillons biologiques sont et restent à cet égard indispensables. |
Footnotes |
1. Jordan B. Puces-Actualités. Med Sci (Paris) 2002; 18 : 287–301. 2. Tan PK, Downey TJ, Spitznagel EL Jr, et al. Evaluation of gene expression measurements from commercial microarray platforms. Nucleic Acids Res 2003; 31 : 5676–84. 3. Nimgaonkar A, Sanoudou D, Butte AJ, et al. Reproducibility of gene expression across generations of Affymetrix microarrays. BMC Bioinformatics 2003; 4 : 27. 4. Kuo WP, Jenssen TK, Butte AJ, et al. Analysis of matched mRNA measurements from two different microarray technologies. Bioinformatics 2002; 18 : 405–12. 5. Kothapalli R, Yoder SJ, Mane S, Loughran TP Jr. Microarray results : how accurate are they ? BMC Bioinformatics 2002; 3 : 22. 6. Lee JK, Bussey KJ, Gwadry FG, et al. Comparing cDNA and oligonucleotide array data: concordance of gene expression across platforms for the NCI-60 cancer cells. Genome Biol 2003; 4 : R82. 7. Barczak A, Rodriguez MW, Hanspers K, et al. Spotted long oligonucleotide arrays for human gene expression analysis. Genome Res 2003; 13 : 1775–85. |