Les lymphomes diffus à grandes cellules de phénotype B (LDGC) constituent 30 % à 40 % des lymphomes observés dans les pays occidentaux. Ils sont individualisés dans la classification OMS des hémopathies qui reconnaît le caractère très hétérogène de cette entité. L’index pronostique international (IPI) incluant des facteurs cliniques simples (âge, stade, performance status, nombre de sites extra-ganglionnaires atteints) et un facteur biologique (taux de LDH, lactate déshydrogénase) est utilisé par les cliniciens depuis 1993. Il constitue un élément essentiel dans le choix de la stratégie thérapeutique. Toutefois, cet index ne rend compte que partiellement de la grande hétérogénéité de ce type de lymphome. Récemment, l’hétérogénéité des LDGC a été en partie comprise à la suite de l’étude des profils d’expression génique en micro-arrray [ 1– 3]. On distingue ainsi au moins deux sous-groupes de LDGC :
- le type germinal center B-cell like (GCB), caractérisé par l’expression de gènes codant pour des molécules (BCL-6, CD10 notamment) habituellement exprimées dans les lymphocytes B des centres germinatifs ;
- le type activated B-cell like (ABC), exprimant des gènes surexprimés en cas d’activation in vitro des lymphocytes B (tels que BCL-2) et des gènes de différenciation plasmocytaire (MUM-1, FOXP1 ou XBP-1). Le phénotype GCB est de meilleur pronostic que le phénotype ABC, et ce indépendamment de l’IPI.
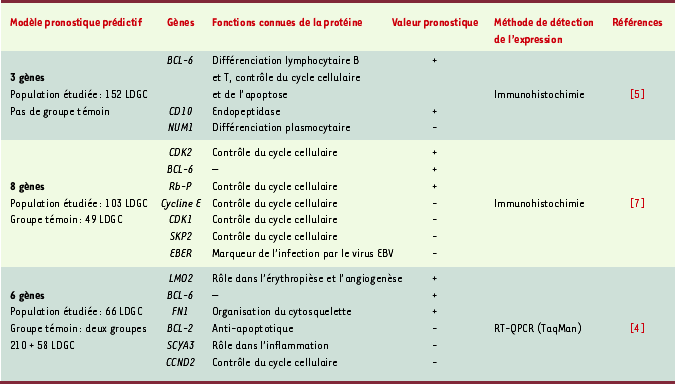
L’étude des profils d’expression génomique, onéreuse, non standardisée, est pour le moment réservée à la recherche, et n’est pas applicable « au lit du malade ». L’objectif de travaux récemment publiés a été d’utiliser la somme considérable d’informations obtenues par micro-array pour n’en garder que les données les plus pertinentes, et susceptibles d’être obtenues par des techniques de routine à partir de la tumeur. I.S. Lossos et al. [ 4] ont ainsi étudié la valeur pronostique, dans un groupe de 66 LDGC, de l’expression de 36 gènes, qui, pour la majorité d’entre eux, avaient été précédemment analysés par la technique de micro-array. L’expression de ces 36 gènes a été évaluée comparativement à deux gènes témoins (GAPDH et PGK1) par PCR quantitative en temps réel (technologie TaqMan), technique maintenant bien standardisée et reproductible. Six gènes (Tableau I), dont l’impact pronostique était le plus discriminant en analyse univariée, ont ainsi été retenus et ont permis de construire un modèle pronostique prédictif, indépendant de l’IPI. On peut regretter que la validation du modèle prédictif ait été réalisée dans les groupes étudiés précédemment en micro-array [ 2, 3] ayant mis en évidence l’implication des 6 gènes, et non dans un groupe totalement indépendant. De même, le choix d’inclure deux gènes cibles de BCL-6 (CCDN2 et SCYA3/MIP1 sont réprimés par BCL-6) est discutable dans ce contexte d’expression « minimaliste » souhaitée par les auteurs.
| Tableau I.
Modèles prédictifs pronostiques dans les lymphomes diffus à grandes cellules de phénotype B fondés sur l’expression d’un nombre restreint de gènes et obtenus à partir des données des micro-arrays. + : expression associée à un pronostic favorable ; - : expression associée à un pronostic défavorable ; LDGC : lymphome diffus à grandes cellules de phénotype B. |
Deux études récentes méritent d’être également rapportées dans ce contexte car elles sont un pas supplémentaire vers l’application en routine des profils d’expression génique.
- Dans une étude portant sur 152 LDGC, C.P. Hans et al. [ 5] ont testé la validité de la classification moléculaire obtenue à partir des profils d’expression génique en utilisant une technique simple d’immuno-histochimie sur coupe en paraffine. À partir d’un algorithme classant les lymphomes en type GCB ou non-GCB, en fonction de l’expression de BCL-6, CD10 et MUM-1, les auteurs confirment la pertinence et l’impact pronostique des deux sous-groupes précédemment définis par micro-array. Ainsi, l’analyse par immuno-histochimie montre une survie à 5 ans de 76 % dans le groupe GCB (CD10+, BCL6±) et de 34 % dans le groupe non-GCB (CD10-, BCL6±, MUM1+). Le phénotype GCB ou non-GCB est encore dans cette étude un facteur pronostique indépendant de l’IPI. Une étude comportant un effectif plus faible (42 LDGC) vient de confirmer la valeur pronostique du phénotype GCB défini par l’expression de CD10 et/ou de BCL-6 et par l’absence d’expression de marqueurs d’activation (CD138 et MUM1) [ 6].
- Dans une série de 152 LDGC, A.I. Saez et al. [ 7] ont testé la valeur pronostique de l’expression de 52 molécules détectées en immuno-histochimie. Huit des 52 mo-lécules ayant le plus d’impact sur la survie globale et la survie sans échec en analyse univariée, incluant notamment BCL-6 (Tableau I), ont été retenues et permettent d’établir un score pronostique capable de prédire la survie des patients, indépendamment de l’IPI.
L’enjeu de ces études ne réside pas seulement dans une meilleure compréhension de la physiopathologie des LDGC. Ces études pourraient permettre d’établir un « IPI biologique » incluant les caractéristiques biologiques de la tumeur les plus pertinentes pour leur signification pronostique.
Toutefois, un point majeur concernant ces résultats doit être souligné. Toutes ces études ont été réalisées à partir de patients traités de façon homogène par un traitement de type CHOP ou CHOP-like, chimiothérapie de référence jusqu’à il y a peu, mais qui est devenu maintenant historique. En effet, l’immuno-chimiothérapie combinant une chimiothérapie classique à un anticorps monoclonal (constitué pour l’essentiel par le Rituximab, anticorps humanisé dirigé contre l’antigène lymphocytaire CD20) a fait la preuve de sa supériorité en termes de survie globale et de survie sans rechute et constitue un nouveau gold standard [ 8]. Le mode d’action original de l’anticorps, potentialisé par la chimiothérapie, fait que ce traitement modifie l’impact pronostique de certaines caractéristiques biologiques de la tumeur. C’est ainsi que l’expression habituellement péjorative de BCL-2 voit sa valeur pronostique disparaître dès lors que les patients sont traités par le Rituximab [ 9]. Quel est l’impact du phénotype GCB ou non-GCB pour les patients recevant un traitement combiné ? L’absence d’expression de BCL-6 est-elle toujours défavorable dans cette situation ? Ces questions sont actuellement sans réponse et soulignent la relativité des facteurs pronostiques qui sont étroitement associés aux évolutions thérapeutiques. Finalement, l’indice pronostique moderne idéal, qui reste à définir, utilisable pour de nouveaux essais thérapeutiques, devra être un indice combiné. Il pourrait associer les données cliniques (l’IPI) et un indice biologique simple applicable en routine, reflétant les caractéristiques moléculaires de la prolifération et être validé dans une population de patients traités par une combinaison Rituximab/chimiothérapie. Un autre intérêt majeur pour les cliniciens de l’étude des profils d’expression est la possibilité de définir de nouvelles cibles thérapeutiques. Ainsi, la surexpression de certains gènes, mise en évidence par micro-array, tel que le gène codant pour la protéine kinase PKCβ, s’associe au caractère chimiorésistant de certains LDGC [3]. Des essais de phase II, utilisant un inhibiteur de la PKCβ, ont été mis en route depuis cette observation, dans les LDGC et les lymphomes à cellules du manteau. L’expression de la protéine est évaluée par simple immuno-histochimie et sera corrélée à la réponse thérapeutique. Ces essais pourraient constituer dans un proche avenir l’une des premières retombées thérapeutiques de l’étude des profils d’expression génique dans les lymphomes.