| |
| Med Sci (Paris). 2005 January; 21(1): 66–72. Published online 2005 January 15. doi: 10.1051/medsci/200521166.Les noyaux suprachiasmatiques : une horloge circadienne composée Hugues Dardente and Nicolas Cermakian* Centre de recherche de l’Hôpital Douglas, Laboratoire de Chronobiologie moléculaire, Université McGill, 6875, boulevard LaSalle, Montréal, Québec, H4H 1R3 Canada |
Un individu isolé, sans aucun repère temporel, n’en continue pas moins de présenter une rythmicité évidente de diverses fonctions physiologiques telles que l’alternance veille/sommeil ou la température corporelle. La persistance de ces rythmes en conditions d’isolement temporel suivant une période proche de 24 heures les définit comme étant des rythmes circadiens [
1]. Comme de nombreux animaux, végétaux, champignons et bactéries, l’homme possède une horloge endogène qui lui permet de maintenir et d’ajuster ses rythmes de façon très précise. C’est cette même horloge qui est responsable des perturbations qu’entraînent les périples entre fuseaux horaires. Ces perturbations ne sont que transitoires, le temps pour l’horloge de se remettre à l’heure : c’est le phénomène d’entraînement. Celui-ci requiert des facteurs synchroniseurs : le plus efficace est la lumière (facteur photique), mais il en existe d’autres, tels que la température et le rythme de prise alimentaire. L’horloge principale des mammifères se situe dans les noyaux suprachiasmatiques (NSC) de l’hypothalamus [1]. Leur rôle est illustré dans la Figure 1, qui prend comme exemple le rythme d’activité locomotrice chez un rongeur.
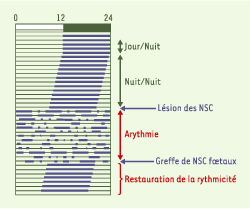
 | Figure 1.
Propriétés fondamentales de l’horloge des NSC : entraînement et « libre-cours ». La cage de l’animal est équipée d’une roue à laquelle il a un accès permanent. La roue est reliée à un système d’enregistrement informatique. Le schéma obtenu est appelé « actogramme ». La barre supérieure indique la condition (artificielle) journalière d’éclairement dans laquelle est maintenu le rongeur. Elle consiste en 12 heures de lumière (de 0 à 12, rectangle blanc) et 12 heures d’obscurité (de 12 à 24, rectangle vert). Chaque ligne de l’actogramme correspond à un jour. Lorsque l’animal court dans sa roue, l’activité est enregistrée : elle est symbolisée ici par les barres noires. Lorsque l’animal (ici un rongeur nocturne) est maintenu dans ce régime d’alternance de 12 heures de jour et 12 heures d’obscurité (cinq premières lignes de cet actogramme), sa période d’activité dans la roue est limitée à la période nocturne. L’alternance de périodes de repos (durant le jour) et d’activité (durant la nuit) suit une période d’exactement 24 heures : on dit que cette activité est « entraînée » par l’alternance jour-nuit. L’animal est ensuite soumis à une obscurité constante, il ne dispose donc plus de repère temporel. Dans ces conditions, l’activité demeure organisée, avec des périodes de repos et d’activité. Toutefois, elle semble dériver vers la gauche ; chaque jour, ce rongeur se met à courir dans sa roue un peu plus tôt que la veille : on appelle cette dérive un « libre-cours ». Elle démontre que l’animal possède un système endogène de mesure du temps, dont la période est légèrement différente de 24 heures (ici, inférieure à 24 heures). L’activité locomotrice, comme tous les autres rythmes, d’ailleurs, requiert donc des « donneurs de temps » (principalement la lumière) afin d’assurer une synchronisation exacte avec les cycles extérieurs de 24 heures. La lésion des noyaux suprachiasmatiques (NSC) entraîne la perte d’une activité temporellement organisée, démontrant ainsi que les NSC constituent le substrat neuro-anatomique de la rythmicité. Enfin, la greffe de NSC fœtaux à un animal dont les NSC sont lésés permet de rétablir une activité temporelle organisée. |
Nous nous proposons ici de résumer les connaissances concernant les mécanismes moléculaires de l’horloge, de les reconsidérer sous les angles de la diversité et de l’organisation cellulaire des NSC, et des implications de celles-ci en termes d’entraînement et de distribution du message temporel, et de replacer les NSC dans le contexte plus large de l’organisme, où ils semblent jouer, au sein d’une pléiade d’autres oscillateurs, un rôle de chef d’orchestre. |
L’horloge moléculaire des NSC L’utilisation d’une stratégie de génétique inverse (du phénotype au génotype) a permis, en s’appuyant sur le rythme d’activité locomotrice (trait phénotypique indicateur du fonctionnement et de la phase de l’horloge, Figure 1), d’isoler le premier mutant murin de l’horloge. Le gène, identifié par clonage positionnel, fut baptisé Clock (Clk) [
2] (→). Tirant partie de connaissances acquises chez la drosophile, trois orthologues murins du gène Period (Per) de la drosophile furent également clonés [2]. La protéine CLK (produit du gène Clk) possède un domaine bHLH (basic helix-loop-helix) et deux domaines PAS (PER-ARNT-SIM, les trois premières protéines possédant ce domaine à avoir été identifiées). Les domaines bHLH et PAS pouvant être impliqués dans la dimérisation entre protéines, il semblait possible que CLK possède un partenaire. Celui-ci, un autre facteur bHLH-PAS nommé BMAL1 (brain and muscle ARNT-like protein 1), fut rapidement identifié [2]. Les protéines PER possèdent également un domaine PAS et peuvent s’hétérodimériser avec les produits des gènes Cryptochromes 1-2, les protéines CRY1 et CRY2.
(→) m/s 2000, n° 4, p. 504
La production d’animaux dont les différents gènes ont été invalidés a permis de démontrer leur implication dans l’horloge circadienne : ces animaux présentent des phénotypes allant d’une perturbation modeste des rythmes à une arythmie totale [
3]. Une étude approfondie a permis d’établir un modèle précisant les contributions de ces gènes dans l’horloge (Figure 2). L’hétérodimère CLK/BMAL1 se lie à des séquences E-box dans les promoteurs de gènes cibles, incluant les gènes Per et Cry [3]. Les protéines PER et CRY forment ensuite des complexes qui peuvent inhiber la transcription induite par CLK/BMAL1, et donc leur propre expression. Cela crée une boucle de rétroaction négative, qui est à la base des oscillations des taux d’ARNm codant pour les protéines PER et CRY. Depuis l’établissement de ce modèle de base, de nombreux autres acteurs ont été identifiés [3,
4] : les récepteurs nucléaires Rev-Erbα et RORα, qui répriment et activent, respectivement, le gène Bmal1, et en contrôlent ainsi l’expression rythmique ; Timeless, dont la protéine peut interagir avec les CRY et réprimer Bmal1 ; Npas2 et Bmal2, des homologues de Clk et de Bmal1, respectivement ; les gènes Dec1-2, éléments présomptifs de la boucle négative. Enfin, les interactions entre les différents partenaires ainsi que les modalités de leur translocation nucléaire et de leur stabilité impliquent diverses modifications post-traductionnelles, telles que des phosphorylations et des ubiquitinylations [2]. Il ne fait aucun doute que le nombre d’acteurs devrait encore aller croissant dans les années à venir, amenant à réviser encore le modèle actuel.
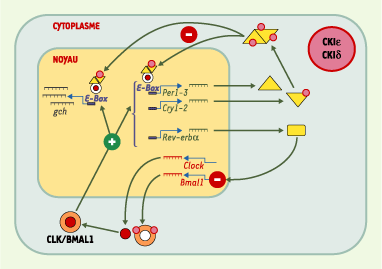
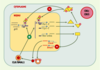 | Figure 2.
Vue simplifiée des mécanismes moléculaires de l’horloge circadienne. Un hétérodimère CLK/BMAL1 est transloqué dans le noyau où il agit comme stimulateur de la transcription de gènes possédant des E-box. Les gènes Period (Per1-3), Cryptochromes (Cry1-2) et Rev-erbα (codant pour un membre de la famille des récepteurs nucléaires orphelins) possèdent des E-box dans leur promoteur. Les protéines PER et CRY s’hétérodimérisent, sont à leur tour transloquées dans le noyau et viennent réprimer l’activité transcriptionnelle du dimère CLK/BMAL1. La protéine REV-ERBα agit comme inhibiteur de la transcription de Bmal1. Les translocations nucléaires ainsi que la stabilité des dimères sont dépendantes de modifications post-traductionnelles, en particulier de phosphorylations par diverses kinases (notamment les caséine kinases (CK) Iδ et Iε). Le dimère CLK/BMAL1 stimule également la transcription de nombreux autres gènes possédant des E-box ; ces gènes sont dits contrôlés par l’horloge (gch) et assurent les sorties physiologiques de celle-ci. Par souci de simplification, les rôles (encore à préciser) des protéines RORalpha, DEC1-2, TIM, NPAS2 et BMAL2 dans l’horloge ont été omis (voir texte pour des précisions). |
|
Les NSC : une horloge hétérogène et dynamique ? Entraînement de l’horloge moléculaire Bien que l’horloge puisse fonctionner de façon autonome (c’est-à-dire sans repère temporel), sa période est ajustée à 24 heures par différents facteurs, dont la lumière, afin d’adapter les rythmes aux conditions environnementales. Comment la lumière réajuste-t-elle l’horloge moléculaire ? Dans la rétine, les photorécepteurs visuels (cônes et bâtonnets), ainsi que des cellules ganglionnaires contenant un photopigment récemment identifié, la mélanopsine, semblent jouer un rôle. Cependant, cônes, bâtonnets et mélanopsine doivent tous trois être supprimés pour abolir toute réponse de l’horloge à la lumière [
5]. Dans les NSC eux-mêmes, la transcription des gènes Per1 et Per2 peut être activée par la lumière, mais uniquement lorsque celle-ci est présentée durant la nuit [ 2]. Or, c’est justement pendant la nuit que la lumière peut décaler la phase de l’horloge biologique, ce qui se traduit par des modifications comportementales (Figure 3).
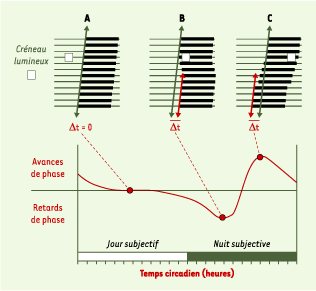
 | Figure 3.
Analyse de l’entraînement des rythmes : la courbe de réponse de phase. Comme cela est montré dans la Figure 1, l’horloge des NSC continue de fonctionner en l’absence de repère temporel (obscurité constante). Sa période est alors différente (bien que proche) de 24 heures. Comme elle demeure néanmoins organisée suivant une alternance rythmique, on peut définir, en se fondant sur un marqueur du fonctionnement de l’horloge tel que l’activité locomotrice, un jour et une nuit, dits « subjectifs » puisque l’animal est en condition d’isolement temporel. Le temps n’est plus nycthéméral, mais circadien. Il est alors possible de tester les effets d’une brève application de lumière (créneau lumineux, carré blanc entouré de vert) à différents temps circadiens sur la phase du rythme d’activité étudié. A. Lorsque le créneau lumineux est appliqué durant le jour subjectif, la phase du rythme est identique avant et après l’application de ce créneau (flèche verte) : en conséquence, la différence temporelle (Δt) est égale à 0. Reportée sur un graphique exprimant la différence de phase en fonction du temps circadien auquel est appliqué le stimulus lumineux, cette valeur est donc sur une ligne horizontale indiquant l’absence d’effet sur la phase. B. Si maintenant le créneau est appliqué au début de la nuit subjective, lorsque l’animal court dans sa roue (barre noire), on observe une modification de la phase du rythme au cours des jours suivants. L’activité locomotrice commence plus tard qu’attendu. La différence temporelle (Δt) entre la phase attendue (flèche verte) et celle observée (flèche rouge) est reportée sur la partie comprise sous la ligne horizontale ; elle correspond à un retard de phase. C. À l’inverse, en fin de nuit, le même stimulus lumineux entraîne dans les jours qui suivent son application un début anticipé de l’activité de roue. On a donc une avance de phase dont la durée Δt est reportée dans la partie supérieure. La répétition de ces stimulus à tous les points du rythme circadien permet ainsi d’établir une courbe, la courbe de réponse de phase. Elle témoigne d’une remise à l’heure de l’horloge biologique, en réponse à un stimulus (ici la lumière) en fonction du moment où celui-ci est appliqué. |
Cette induction des gènes Per fait intervenir le glutamate et le PACAP (pituitary adenylate cyclase activating polypeptide), issus des projections rétiniennes sur les NSC (pour revue, voir [
6]), et requiert le facteur de transcription CREB (cAMP response element binding protein) [
7]. D’autres facteurs que la lumière, regroupés sous le terme de « facteurs non photiques »(par exemple, la présentation d’une nouvelle roue, certains neuromédiateurs), peuvent également synchroniser les NSC [
8]. À l’inverse du facteur photique, la sensibilité aux facteurs non photiques est largement limitée au jour. Ainsi, alors que la lumière durant la nuit induit l’expression de Per1 et de Per2, les facteurs non photiques appliqués durant le jour entraînent une diminution de l’expression de ces deux mêmes gènes [8]. Per1 et Per2 semblent donc être la cible des facteurs synchroniseurs, qu’ils soient photiques ou non, dans l’horloge moléculaire. Toutefois, la série d’événements intervenant entre l’effet des facteurs photiques et non photiques sur les expressions de Per1 et de Per2 et les modifications de phase des rythmes demeure mal comprise. Brève histologie des NSC Les NSC constituent une petite structure paire, située au-dessus du chiasma optique, dans l’hypothalamus antérieur [ 1]. Chez le rat, chacun des NSC compte à peine 10 000 neurones, parmi les plus petits du cerveau (7 à 10 µm de diamètre), et mesure environ 700 µm suivant l’axe rostrocaudal, et 350 µm pour les axes médiolatéral et dorsoventral. Les NSC présentent une organisation caractéristique retrouvée chez presque tous les mammifères étudiés, y compris l’humain. Schématiquement, on peut définir une partie dorsomédiane également appelée shell (coquille), à l’origine de la plupart des efférences des NSC, et une partie ventrolatérale, le core (cœur), impliquée dans l’intégration des signaux photiques et non photiques puisqu’elle reçoit les afférences des NSC (Figure 4).
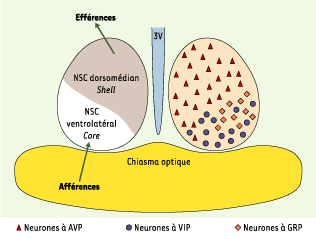
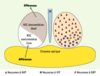 | Figure 4.
Organisation morphofonctionnelle et neurochimique simplifiée des NSC. Les NSC sont une structure paire située dans l’hypothalamus, au-dessus du chiasma optique et de part et d’autre du troisième ventricule (3V). On distingue, d’après leur phénotype neuropeptidique, différents types de neurones (à droite) : à vasopressine (AVP), à peptide vasoactif intestinal (VIP) et à peptide libérant la gastrine (GRP). Ces types cellulaires sont distribués de façon dissymétrique dans les NSC et permettent de définir une partie dorsomédiane (shell) et une partie ventrolatérale (core) (à gauche). Cette distinction est retrouvée sur le plan de l’organisation fonctionnelle des NSC : le core reçoit les afférences (photiques et non photiques), alors que du shell part la majorité des projections. |
Le shell est constitué de neurones exprimant la vasopressine (AVP), alors que le core renferme des neurones exprimant deux autres neuropeptides : le peptide intestinal vasoactif (VIP) et le peptide libérant la gastrine (GRP) (Figure 4). Comme le suggère son nom, la partie shell enveloppe la partie core suivant les plans rostrocaudal et médiolatéral. Pour cette raison, les parties les plus rostrales et caudales des NSC ne contiennent que des neurones à AVP. D’autres neurones, tels que ceux exprimant la somatostatine, sont également présents dans les NSC. Une grande majorité des neurones des NSC exprime la glutamate décarboxylase, et utiliserait donc le neurotransmetteur inhibiteur GABA (acide γ-amino butyrique) [
9], alors que d’autres neurones utilisent un neurotransmetteur excitateur, le glutamate [
10]. Une horloge hétérogène et dynamique De nombreuses études réalisées au cours des années 1980 à 1995 se sont attachées à caractériser la répartition des neuropeptides dans les NSC, leur expression au cours d’un cycle circadien et les effets de la lumière sur ces profils d’expression (pour revue, voir [
11]). En revanche, il n’a guère été tenu compte de cette organisation dans les années suivantes, au cours desquelles de grands progrès ont été réalisés dans la compréhension des bases moléculaires de l’horloge. Des résultats récents invitent à reconsidérer les données moléculaires dans le contexte neurochimique des NSC. Le neurone constitue la plus petite unité rythmique, comme le démontre la persistance du rythme d’activité électrique de neurones dissociés des NSC [
12]. Toutefois, in vivo, la précision et la robustesse du rythme dépendent d’un fonctionnement synchronisé de toutes ces unités [
13–
15]. La façon la plus simple de concevoir l’horloge est de la considérer comme identique pour chaque unité cellulaire des NSC. En d’autres termes, chaque neurone renferme une horloge semblable et toutes les horloges, c’est-à-dire chaque neurone, présentent une phase semblable. La réalité est toutefois plus complexe. En fait, la proportion de neurones présentant un rythme d’activité électrique est fonction du type cellulaire [
16] : elle est plus grande pour les neurones qui expriment l’AVP que pour ceux qui expriment le VIP. Ces données électrophysiologiques sont appuyées par des études sur la distribution des ARNm des gènes de l’horloge au cours d’un cycle circadien. En effet, lorsqu’ils sont à leur maximum d’expression, durant le jour, les ARNm des gènes Per1-2 sont principalement observés dans la partie dorsomédiane (neurones à AVP) des NSC chez le hamster doré [
17], le rat [
18,
19] et la souris [
20] ; la proportion de neurones à VIP exprimant ces gènes est quant à elle beaucoup plus faible dans toutes ces espèces. Toutefois, les neurones des parties ventrolatérale et dorsomédiane présentent une expression rythmique suivant la même phase, du moins chez le rat [19]. L’utilisation de techniques telles que l’hybridation in situ non radioactive [19,
21] ou l’emploi d’animaux transgéniques exprimant le gène rapporteur luciférase sous le contrôle du promoteur du gène Per1 [15] ont permis de décomposer la dynamique de l’expression des gènes Per au cours d’un cycle circadien. Tout se passe comme si une partie des neurones des NSC initiait l’expression de ces gènes puis, de proche en proche, la conférait aux neurones adjacents, conduisant ultimement à une expression ubiquitaire de ces gènes dans les NSC, lors du pic. Lorsque les neurones sont suivis individuellement, on s’aperçoit d’ailleurs qu’ils présentent des phases assez variables (c’est-à-dire que le pic des oscillations ne survient pas au même moment), certains neurones étant même complètement déphasés [16,
22] ; de plus, certaines régions semblent contenir des neurones arythmiques [17,
23]. Les NSC fonctionnent donc comme un ensemble très hétérogène, peut-être hiérarchisé : la production des rythmes des NSC en tant que tissu est la résultante d’une moyenne des activités des différents compartiments, voire des milliers d’oscillateurs qui les composent. Cela fut joliment souligné par l’emploi de souris chimères [
24] possédant des quantités variables, mais définies, de neurones ayant une horloge soit fonctionnelle (avec des copies du gène Clk sauvage), soit invalidée (avec des copies du gène Clk mutant). Le phénotype des souris chimères dépend de la proportion de neurones mutants : plus celle-ci est importante, moins la rythmicité est soutenue. La compartimentation des NSC semble importante en termes d’entraînement photique, puisque l’induction des gènes Per1 et Per2 est elle-même topographiquement différente : alors que l’induction de Per2 survient dans presque la totalité de la partie ventrolatérale des NSC (région présentant une innervation rétinienne), l’induction de Per1 est beaucoup plus limitée [18,
25] et semble restreinte aux neurones exprimant le GRP, suggèrant un rôle important de ce neuropeptide dans l’entraînement photique [18,
26]. Ces profils d’induction de Per1 et de Per2, phénotypiquement limités et différentiels, donnent une idée de la complexité des mécanismes mis en jeu lors de la synchronisation des NSC et invitent à une considération plus importante de l’organisation neurochimique des NSC. La nécessité d’une communication entre les parties ventrolatérale et dorsomédiane dans la remise à l’heure de l’horloge a été proposée récemment [
27] : elle ouvre de nouvelles perspectives. À l’appui de ces données sur l’importance des subdivisions des NSC, un rôle fondamental des neuropeptides eux-mêmes dans le maintien de la rythmicité (d’un point de vue moléculaire comme phénotypique) et l’entraînement photique a été mis en évidence récemment chez des souris invalidées, soit pour l’un des récepteurs du VIP présent dans les NSC, le récepteur VPAC2 [
28,
29], soit pour le précurseur du VIP lui-même [
30]. D’autres molécules, dont le GABA [
31,
32], sont impliquées dans la synchronisation intercellulaire des NSC. Plus généralement, l’activité électrique synaptique [16], les jonctions intercellulaires [32], des molécules d’adhérence cellulaire [
33] et des interactions temporellement dynamiques entre les neurones et la glie [
34] sont également mises en jeu. Combien d’horloges dans les NSC ? Des données récentes indiquent que les parties shell et core des NSC hébergent vraisemblablement deux oscillateurs distincts, pouvant sous certaines conditions fonctionner de manière indépendante, bien qu’ils soient normalement synchronisés in vivo [
35,
36]. Ces résultats viennent en appui de données, obtenues in vitro, démontrant que les rythmes de sécrétion d’AVP et de VIP peuvent être découplés [
37,
38]. Chaque NSC serait donc, hypothétiquement, composé de deux oscillateurs couplés. Enfin, les deux NSC peuvent également être découplés (du moins chez le hamster doré), donnant lieu à deux rythmes distincts d’activité locomotrice résultant d’expressions antiphasiques des gènes de l’horloge dans chaque NSC [
39]. Cela nous donne donc, arithmétiquement, quatre oscillateurs relativement indépendants, mais néanmoins couplés, chacun étant constitué de multiples unités rythmiques (les neurones). Le chemin de la compréhension du fonctionnement intégré des NSC et de son entraînement est décidément long et pavé de surprises… |
Les horloges périphériques Longtemps considérés comme nécessaires et suffisants dans la genèse et le maintien des rythmes biologiques, les NSC ont un rôle moins clair aujourd’hui. D’autres tissus expriment de façon rythmique les gènes de l’horloge [
40], et cette rythmicité peut également être induite dans des cellules non nerveuses en culture [40]. Ces études ont provoqué de profonds bouleversements : les NSC n’ont pas le privilège de la rythmicité. L’utilisation de rats transgéniques avec un rapporteur luciférase sous le contrôle du promoteur de Per1 a permis de démontrer que de nombreuses régions du cerveau, ainsi que des tissus non neuronaux, présentent des oscillations pendant quelques jours lorsqu’ils sont mis en culture [
41]. In vivo, cependant, il semble que les NSC jouent un rôle d’oscillateur principal contrôlant les autres oscillateurs du système circadien [
42]. En effet, la lésion des NSC entraîne une disparition apparente des rythmes dans les autres tissus [
43,
44] : les NSC, s’ils ne sont pas les seuls à exprimer une rythmicité, apparaissent donc indispensables à la fonction des autres oscillateurs. Ces oscillateurs subordonnés seraient moins robustes, et leur rythmicité moins soutenue que celle des NSC. Toutefois, l’utilisation de souris knock-in pour le gène Per2 (exprimant une protéine de fusion PER2-luciférase) pointe certains biais expérimentaux des études antérieures [
45]. Celles-ci utilisaient des animaux différents à chaque temps du jour, et ne tenaient donc pas compte de la possibilité que l’absence de rythmicité soit en fait la résultante d’une moyenne des déphasages entre individus. S.L. Yoo et al. [45], en suivant pendant plusieurs jours les mêmes tissus de souris aux NSC lésés, ont observé une rythmicité persistante assortie, toutefois, de périodes variables entre individus et d’une profonde désynchronisation entre les organes d’un même animal. Ainsi, de générateurs de la rythmicité, les NSC se trouvent convertis en chef d’orchestre assurant la coordination de toutes les partitions [45,
46]. Bien qu’attirantes, ces données devront encore être confortées. |
L’emploi des techniques de biologie moléculaire (clonage) et d’outils génétiques (animaux mutants, invalidés et transgéniques) durant la dernière décennie a permis des avancées considérables dans la compréhension des mécanismes moléculaires de l’horloge circadienne, concrétisées par l’établissement d’un modèle qui en précise les rouages. Plus récemment, l’attention s’est portée sur une étude plus intégrée du fonctionnement de cette horloge, des voies de son entraînement et de la façon dont elle distribuait le temps aux différents tissus, qu’ils soient nerveux ou périphériques. Toutes ces études indiquent que les NSC sont en fait une horloge composée, hétérogène, constituée de sous-ensembles et de compartiments, reflets de son organisation cytoarchitectonique et neurochimique, ayant chacun un rôle dédié. La mise à l’heure du temps biologique implique une coordination entre toutes ces sous-unités, ainsi que la participation de nombreux facteurs et mécanismes cellulaires. Une compréhension beaucoup plus poussée des acteurs et des cascades d’événements impliqués semble aujourd’hui un préalable nécessaire pour espérer expliquer comment les NSC assurent le contrôle de la synchronisation des fonctions physiologiques. ¹ |
Nous remercions les autres membres du laboratoire pour leurs précieux conseils sur ce texte. H. Dardente est boursier post-doctoral de la Fondation pour la Recherche Médicale (France). La recherche dans le laboratoire de N. Cermakian est financée par les Instituts de recherche en santé du Canada, le Conseil de la recherche en sciences naturelles et en génie, le Fonds de la recherche en santé du Québec et le Programme scientifique des frontières humaines.
|
1. Dunlap JC, Loros JJ, DeCoursey PJ. Chronobiology : biological timekeeping. Sunderland, MA : Sinauer Associates, 2003 : 406 p. 2. Reppert SM, Weaver DR. Coordination of circadian timing in mammals. Nature 2002; 418 : 935–41. 3. Cermakian N, Boivin DB. A molecular perspective of human circadian rhythm disorders. Brain Res Brain Res Rev 2003; 42 : 204–20. 4. Emery P, Reppert SM. A rhythmic ror. Neuron 2004; 43 : 443–6. 5. Hattar S, Lucas RJ, Mrosovsky N, et al. Melanopsin and rod-cone photoreceptive systems account for all major accessory visual functions in mice. Nature 2003; 424 : 76–81. 6. Hannibal J. Neurotransmitters of the retino-hypothalamic tract. Cell Tissue Res 2002; 309 : 73–88. 7. Travnickova-Bendova Z, Cermakian N, Reppert SM, Sassone-Corsi P. Bimodal regulation of mperiod promoters by creb-dependent signaling and clock/bmal1 activity. Proc Natl Acad Sci USA 2002; 99 : 7728–33. 8. Challet E, Pevet P. Interactions between photic and nonphotic stimuli to synchronize the master circadian clock in mammals. Front Biosci 2003; 8 : S246–57. 9. Moore RY, Speh JC. Gaba is the principal neurotransmitter of the circadian system. Neurosci Lett 1993; 150 : 112–6. 10. Perreau-Lenz S, Kalsbeek A, Garidou ML, et al. Suprachiasmatic control of melatonin synthesis in rats : inhibitory and stimulatory mechanisms. Eur J Neurosci 2003; 17 : 221–8. 11. Dardente H, Menet JS, Challet E, et al. Daily and circadian expression of neuropeptides in the suprachiasmatic nuclei of nocturnal and diurnal rodents. Brain Res Mol Brain Res 2004; 124 : 143–51. 12. Welsh DK, Logothetis DE, Meister M, Reppert SM. Individual neurons dissociated from rat suprachiasmatic nucleus express independently phased circadian firing rhythms. Neuron 1995; 14 : 697–706. 13. Liu C, Weaver DR, Strogatz SH, Reppert SM. Cellular construction of a circadian clock : period determination in the suprachiasmatic nuclei. Cell 1997; 91 : 855–60. 14. Herzog ED, Aton SJ, Numano R, et al. Temporal precision in the mammalian circadian system : a reliable clock from less reliable neurons. J Biol Rhythms 2004; 19 : 35–46. 15. Yamaguchi S, Isejima H, Matsuo T, et al. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science 2003; 302 : 1408–12. 16. Nakamura W, Honma S, Shirakawa T, Honma K. Regional pacemakers composed of multiple oscillator neurons in the rat suprachiasmatic nucleus. Eur J Neurosci 2001; 14 : 666–74. 17. Hamada T, LeSauter J, Venuti JM, Silver R. Expression of period genes : rhythmic and nonrhythmic compartments of the suprachiasmatic nucleus pacemaker. J Neurosci 2001; 21 : 7742–50. 18. Dardente H, Poirel VJ, Klosen P, et al. Per and neuropeptide expression in the rat suprachiasmatic nuclei : compartmentalization and differential cellular induction by light. Brain Res 2002; 958 : 261–71. 19. Yan L, Okamura H. Gradients in the circadian expression of per1 and per2 genes in the rat suprachiasmatic nucleus. Eur J Neurosci 2002; 15 : 1153–62. 20. LeSauter J, Yan L, Vishnubhotla B, et al. A short half-life gfp mouse model for analysis of suprachiasmatic nucleus organization. Brain Res 2003; 964 : 279–87. 21. Hamada T, Antle MC, Silver R. Temporal and spatial expression patterns of canonical clock genes and clock-controlled genes in the suprachiasmatic nucleus. Eur J Neurosci 2004; 19 : 1741–8. 22. Herzog ED, Geusz ME, Khalsa SB, et al. Circadian rhythms in mouse suprachiasmatic nucleus explants on multimicroelectrode plates. Brain Res 1997; 757 : 285–90. 23. Jobst EE, Allen CN. Calbindin neurons in the hamster suprachiasmatic nucleus do not exhibit a circadian variation in spontaneous firing rate. Eur J Neurosci 2002; 16 : 2469–74. 24. Low-Zeddies SS, Takahashi JS. Chimera analysis of the clock mutation in mice shows that complex cellular integration determines circadian behavior. Cell 2001; 105 : 25–42. 25. Yan L, Takekida S, Shigeyoshi Y, Okamura H. Per1 and per2 gene expression in the rat suprachiasmatic nucleus : circadian profile and the compartment-specific response to light. Neuroscience 1999; 94 : 141–50. 26. Karatsoreos IN, Yan L, LeSauter J, Silver R. Phenotype matters : Identification of light-responsive cells in the mouse suprachiasmatic nucleus. J Neurosci 2004; 24 : 68–75. 27. Yan L, Silver R. Differential induction and localization of mper1 and mper2 during advancing and delaying phase shifts. Eur J Neurosci 2002; 16 : 1531–40. 28. Harmar AJ, Marston HM, Shen S, et al. The vpac(2) receptor is essential for circadian function in the mouse suprachiasmatic nuclei. Cell 2002 109 : 497–508. 29. Hughes AT, Fahey B, Cutler DJ, et al. Aberrant gating of photic input to the suprachiasmatic circadian pacemaker of mice lacking the vpac2 receptor. J Neurosci 2004; 24 : 3522–6. 30. Colwell CS, Michel S, Itri J, et al. Disrupted circadian rhythms in vip- and phi-deficient mice. Am J Physiol Regul Integr Comp Physiol 2003; 285 : R939–49. 31. Liu C, Reppert SM. Gaba synchronizes clock cells within the suprachiasmatic circadian clock. Neuron 2000; 25 : 123–8. 32. Shinohara K, Hiruma H, Funabashi T, Kimura F. Gabaergic modulation of gap junction communication in slice cultures of the rat suprachiasmatic nucleus. Neuroscience 2000; 96 : 591–6. 33. Prosser RA, Rutishauser U, Ungers G, et al. Intrinsic role of polysialylated neural cell adhesion molecule in photic phase resetting of the mammalian circadian clock. J Neurosci 2003; 23 : 652–8. 34. Serviere J, Lavialle M. Astrocytes in the mammalian circadian clock : putative roles. Prog Brain Res 1996; 111 : 57–73. 35. Nagano M, Adachi A, Nakahama K, et al. An abrupt shift in the day/night cycle causes desynchrony in the mammalian circadian center. J Neurosci 2003; 23 : 6141–51. 36. de la Iglesia HO, Cambras T, Schwartz WJ, Diez-Noguera A. Forced desynchronization of dual circadian oscillators within the rat suprachiasmatic nucleus. Curr Biol 2004; 14 : 796–800. 37. Shinohara K, Honma S, Katsuno Y, et al. Two distinct oscillators in the rat suprachiasmatic nucleus in vitro. Proc Natl Acad Sci USA 1995; 92 : 7396–400. 38. Shirakawa T, Honma S, Katsuno Y, et al. Synchronization of circadian firing rhythms in cultured rat suprachiasmatic neurons. Eur J Neurosci 2000; 12 : 2833–8. 39. De la Iglesia HO, Meyer J, Carpino A Jr, Schwartz WJ. Antiphase oscillation of the left and right suprachiasmatic nuclei. Science 2000; 290 : 799–801. 40. Balsalobre A. Clock genes in mammalian peripheral tissues. Cell Tissue Res 2002; 309 : 193–9. 41. Yamazaki S, Numano R, Abe M, et al. Resetting central and peripheral circadian oscillators in transgenic rats. Science 2000; 288 : 682–5. 42. Pando MP, Morse D, Cermakian N, Sassone-Corsi P. Phenotypic rescue of a peripheral clock genetic defect via scn hierarchical dominance. Cell 2002; 110 : 107–17. 43. Akhtar RA, Reddy AB, Maywood ES, et al. Circadian cycling of the mouse liver transcriptome, as revealed by cdna microarray, is driven by the suprachiasmatic nucleus. Curr Biol 2002; 12 : 540–50. 44. Sakamoto K, Nagase T, Fukui H, et al. Multitissue circadian expression of rat period homolog (rper2) mrna is governed by the mammalian circadian clock, the suprachiasmatic nucleus in the brain. J Biol Chem 1998; 273 : 27039–42. 45. Yoo SH, Yamazaki S, Lowrey PL, et al. Period2 : luciferase real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc Natl Acad Sci USA 2004; 101 : 5339–46. 46. Teboul M, Delaunay F. Ni maître ni esclave chez les horloges biologiques. Med Sci (Paris) 2004; 20 : 628–9. |