| |
| Med Sci (Paris). 2005 February; 21(2): 170–174. Published online 2005 February 15. doi: 10.1051/medsci/2005212170.Anomalies de la transcription et diagnostic en génétique constitutionnelle Claude Houdayer1* and Dominique Stoppa-Lyonnet 1Service de Génétique oncologique, Inserm U.509, Pathologie moléculaire des cancers, Institut Curie, 26, rue d’Ulm, 75248 Paris Cedex 05, France |
Les anomalies d’épissage représentent 10 % des mutations rapportées dans la Human Gene Mutation Database [
1], ce qui est déjà certainement sous-estimé, car seules les mutations affectant les sites donneurs et accepteurs d’épissage (voir plus loin) sont prises en compte. De plus, cette valeur moyenne peut atteindre près de 50 % pour certains gènes comme NF1 [
2] ou ATM [
3]. La mise en évidence des anomalies d’épissage, et plus largement de la transcription, est donc incontournable en diagnostic. Elle repose sur les connaissances validées en termes d’épissage et de régulation de l’expression génique. |
Épissage et séquences consensus en cis
L’épissage est le processus complexe par lequel les cellules eucaryotes produisent un ARN messager (ARNm) mature à partir d’un pré-ARNm. Il nécessite la reconnaissance des exons, l’excision des introns, puis l’union des exons pour former un transcrit mature. Cette reconnaissance est assurée par des séquences génomiques consensus en cis, dont les plus connues sont les sites donneurs, les sites accepteurs d’épissage et le site de branchement (Figure 1). Les altérations de ces sites et leurs conséquences sont maintenant bien mises en évidence (Figure 2). Grandes délétions, insertions ou délétions d’une ou plusieurs bases et modifications nucléotidiques sont à même d’altérer la fonction de ces sites, avec des conséquences diverses : (1) abolition du site physiologique avec saut de l’exon concerné (Figure 2A) ; (2) abolition du site physiologique avec révélation d’un site dit cryptique, qui prend alors le relais du site sauvage : en fonction de la localisation du site cryptique, il s’ensuit une délétion exonique (Figure 2B) ou une rétention intronique (Figure 2C) avec, respectivement, synthèse d’une protéine déficitaire en acides aminés, ou ayant incorporé des acides aminés supplémentaires ; (3) la combinaison saut d’exon et utilisation d’un site cryptique est également possible. Notons que les modifications nucléotidiques peuvent ne pas toucher ces séquences consensus, mais néanmoins altérer l’épissage en créant un site cryptique qui sera utilisé préférentiellement au site sauvage, avec des conséquences similaires.
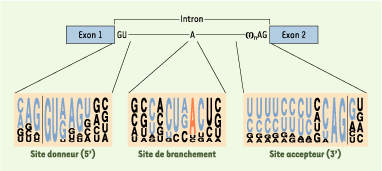
 | Figure 1.
Les sites consensus d’épissage « traditionnels ». Le site donneur correspond à la charnière exon/intron. Le site de branchement, riche en bases pyrimidiques et qui contient un A est situé à une trentaine de bases en amont du site accepteur, qui correspond à la charnière intron/exon. Pour les séquences consensus, la taille des nucléotides est proportionnelle à leur fréquence estimée par alignement de 1 683 séquences humaines introniques. Lors de l’épissage, le site donneur vient se lier au site de branchement (formation du splicéosome) pour aboutir à la libération de l’intron sous forme de lasso et à la production de l’ARN messager (ARNm) avec deux exons raboutés (d’après [
5]). |
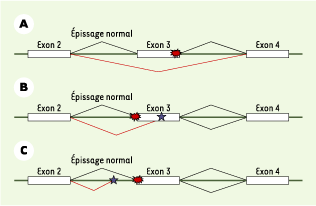
 | Figure 2.
Représentation de quelques anomalies d’épissage. Les exons sont représentés par des rectangles, les introns par un trait vert. La position de la mutation est indiquée par un symbole rouge ; les traits rouges schématisent l’anomalie de la transcription. A. Altération du site donneur de l’exon 3, saut de l’exon 3. B. Abolition du site accepteur de l’exon 3, avec révélation d’un site cryptique exonique (étoile bleue) : délétion exonique partielle. C. Abolition du site accepteur de l’exon 3, avec révélation d’un site cryptique intronique (étoile bleue) : rétention intronique. |
Récemment, de nouvelles séquences consensus ont émergé, principalement les ESE (exonic splicing enhancers) et les ESS (exonic splicing silencers). Ce sont des séquences exoniques qui favoriseraient (ESE), ou réprimeraient (ESS) l’épissage de l’exon qui les contient, et ce par interaction avec des facteurs de transcription spécifiques. L’existence des ESE était en fait pressentie depuis longtemps [
4], mais s’ils sont devenus d’une actualité incontournable, c’est principalement grâce aux travaux de l’équipe d’Adrian Krainer qui a amélioré leur définition et montré leur importance en pathologie humaine [5]. Ainsi, on a longtemps pensé que l’impact délétère de la mutation BRCA1 Cys64Gly résidait dans la synthèse d’une protéine BRCA1 avec un domaine RING finger altéré. Il s’avère, en fait, que le retentissement majeur de cette mutation est une anomalie de l’épissage par altération d’un ESE, avec production d’un messager aberrant hors cadre qui serait ensuite détruit par NMD (nonsense mediated decay) (voir plus loin) [
6]. Cela étant posé, les connaissances de ces séquences sont à approfondir, comme le montre le débat sur SMN2 (survival motor neuron 2), dont l’anomalie d’épissage serait due soit à l’inactivation d’un ESE [
7] soit, au contraire, à l’activation d’un ESS [
8]. ESE et ESS ont leurs homologues introniques (ISE et ISS), mais leurs séquences consensus sont moins bien définies et leur implication en pathologie n’a été qu’exceptionnellement rapportée [
9]. Plus complexes, les composite exonic regulatory element of splicing pourraient combiner des propriétes activatrices et inhibitrices de l’épissage [
10]. Dans ces anomalies de l’épissage, la notion de traduction n’importe plus, seule la composition en nucléotides compte : ainsi, une modification nucléotidique d’apparence bénigne (neutre par exemple) peut abolir, créer ou renforcer une séquence consensus et perturber l’épissage normal [3, 10,
11]. C’est également le cas - exemplaire - des microsatellites, largement distribués dans le génome, et dont la variation de taille peut retentir sur l’expression : (1) en modifiant les distances entre les séquences d’épissage en cis ; (2) en formant des structures secondaires ; (3) en se liant à des facteurs protéiques modifiant l’épissage [
12,
13]. |
Anomalies du promoteur et niveau d’expression C’est le second aspect de la transcription « appréhendable » en diagnostic. En effet, une simple modification nucléotidique sur le promoteur d’un gène peut, par plusieurs mécanismes, retentir sur son niveau d’expression, jusqu’à être délétère. Tout d’abord, elle peut altérer un site consensus de liaison aux facteurs de transcription nucléaires, et de ce fait, diminuer, voire éteindre, l’expression génique [
14–
16]. Par ailleurs, elle peut créer un codon d’initiation de la traduction prématuré (uAUG) et permettre ainsi l’ouverture d’un cadre de lecture en amont du cadre physiologique (upstream open reading frame, uORF). Il en résultera des produits de taille variée, actifs ou non, mais surtout, l’utilisation d’un uAUG peut inhiber la traduction du messager sauvage en empêchant le ribosome d’atteindre l’AUG physiologique [
17]. Les modifications nucléotidiques du promoteur peuvent également avoir un effet délétère en altérant sa structure. Il peut alors se créer une structure secondaire qui fera obstacle au passage du ribosome, abaissant le niveau d’expression ou, au contraire, déstabilisant une structure secondaire nécessaire à l’interaction avec des protéines de régulation [
18]. Enfin, le gène d’intérêt peut être intact, mais son expression altérée via des effets à distance comme des modifications de structure chromatinienne [
19] ou la rupture de séquences régulatrices [
20,
21]. |
Neutraliser le messager altéré : le NMD (nonsense mediated decay)
Le diagnostic des anomalies de la transcription est compliqué par l’instabilité des ARNm portant des mutations tronquantes, ou NMD (nonsense mediated decay). Sans qu’il s’agisse d’une règle absolue [
22], le NMD élimine le messager portant le codon stop prématuré, empêchant ainsi la traduction d’une protéine tronquée potentiellement délétère par effet dominant négatif. Cet effet, probablement bénéfique in vivo, induit un problème majeur pour le diagnostic, car l’anomalie est masquée in vitro. |
Anomalies de la transcription : interprétation biologique L’interprétation de ces anomalies est parfois délicate, car elles sont associées à une pénétrance et à une expressivité variable selon les individus porteurs. Une même anomalie d’épissage de RB1 (retinoblastoma 1) peut rendre compte de formes bilatérales-unilatérales de rétinoblastomes, voire de rétinomes1 (RB1 mutation database, disponible sur http://www.d-lohmann.de/Rb/mutations.html). L’interprétation du caractère causal est encore plus complexe quand il existe des membres non atteints dans la famille. Une autre difficulté de l’analyse des transcrits est l’existence d’un épissage alternatif physiologique, qu’il est indispensable de connaître, afin de ne pas interpréter comme délétère un saut d’exon(s) reflétant, en fait, un transcrit alternatif. L’étude de plusieurs contrôles normaux est donc indispensable. Enfin, et en toute rigueur, l’impact sur l’épissage des mutations rompant le cadre de lecture devrait être étudié, car les conséquences phénotypiques seront peut-être différentes selon qu’elles entraînent un NMD, le saut d’un exon en phase, ou une protéine tronquée par échappement au NMD. La prise en compte des anomalies de la transcription pour le diagnostic génétique est donc aussi importante que délicate. Deux stratégies sont possibles pour les caractériser. L’option « racine », initiale, dépend du choix de l’acide nucléique étudié. Il peut s’agir d’ADN ou d’ARN. |
C’est le plus couramment utilisé en diagnostic. En effet, l’ADN est une molécule robuste, facile à extraire. L’analyse du gène devra comprendre le promoteur, les parties codantes et les jonctions introns/exons, en explorant environ 120 pb dans l’intron, afin de couvrir au mieux les possibles sites cryptiques d’épissage [
23]. Malheureusement, toute anomalie délétère située hors des zones étudiées passera inaperçue, comme, par exemple, des altérations introniques profondes, dont l’existence est pourtant démontrée [
24]. Mais la difficulté majeure de l’abord ADN réside dans l’interprétation de certaines modifications nucléotidiques identifiées. Ainsi, près de 30 % des modifications identifiées sur le gène BRCA1 ne fournissent pas d’interprétation lisible en génomique [
25]. Il s’agit de modifications introniques ou exoniques, dont le retentissement est inconnu (unknown variants), mais qui sont, de fait, candidates à des altérations de la transcription. Leur grand nombre rend une analyse ARN systématique incompatible avec un diagnostic de routine. En revanche, on peut modéliser in silico leur impact afin de débusquer une éventuelle anomalie d’épissage (Tableau I, Figure 3). L’expérience nous a montré que les matrices dédiées aux sites d’épissage « classiques » sont performantes (car ces sites sont bien connus), mais elles sont aussi parfois prises en défaut, car elles ne détectent pas l’anomalie ou, au contraire, prédisent une anomalie inexistante ([10] et données non publiées) : il ne s’agit donc pas d’une arme absolue, mais d’un outil indicatif.
 | Figure 3.
Résultats de la modélisation de la mutation g.56903C → G/Leu220Val sur l’exon 7 du gène RB1. Le nucléotide muté, dans l’exon, est indiqué en lettre capitale bleue. La numérotation indiquée pour Start et End débute au 1er nucléotide des séquences testées. Les sites donneurs identifiés sont soulignés sur les séquences. Splice Site Prediction prédit la création d’un site cryptique pour la séquence mutée, ce qui est confirmé par MaxEntScan. L’analyse ARN a effectivement montré l’utilisation de ce site cryptique. |
 | Tableau I.
Logiciels de reconnaissance et d’analyse des séquences consensus d’épissage. Les différents sites d’épissage recherchés ont des séquences consensus relativement conservées, ce qui autorise leur identification par différents algorithmes. Ces algorithmes permettent également d’évaluer quantitativement l’impact de modifications nucléotidiques au sein de ces séquences car un score est attribué pour chaque base, pour chaque position, par comparaison avec l’hypothèse la plus probable. Concernant la liste présentée (non exhaustive) d’« outils web » de prédiction, il est possible d’interroger simultanément ces différents sites, ce qui simplifie grandement la procédure (données disponibles sur demande). Il existe d’autres logiciels dédiés à l’identification des séquences réglant la transcription [
32,
33]. |
Quant aux matrices dédiées aux sites émergents (ESE finder, rescue ESE), elles sont à manier avec encore plus de précautions, car l’analyse des exons identifie beaucoup d’ESE présomptifs, souvent pour les mêmes facteurs de transcription. L’altération d’un ESE prédite par le logiciel peut correspondre à une réalité in vivo, mais il est également possible qu’il s’agisse d’un artéfact de modélisation (ESE identifié à tort), que le gène d’intérêt n’utilise pas l’ESE incriminé, ou que cet ESE « défaillant » soit secouru par un ESE voisin. En dépit de ces inconvénients, l’analyse in silico a toute sa place dans le processus diagnostique, comme élément d’orientation vers les études complémentaires sur ARN. |
La complexité de l’interprétation des unknown variants et la couverture imparfaite du gène d’intérêt seraient donc en faveur d’une approche diagnostique à partir de l’ARN, dont la puissance a été démontrée [2, 3,
26]. En effet, les mutations non caractérisées par l’approche ADN génomique seront identifiées, du moins si elles retentissent sur l’épissage. Quant au NMD, qui a longtemps été un obstacle à la stratégie ARN, son effet serait prévenu par l’ajout de puromycine [
27]. D’un point de vue pratique, il serait souhaitable de travailler directement sur l’ARN extrait du sang total pour être au plus près des conditions physiologiques du patient. Malheureusement, l’utilisation de lymphocytes circulants reste limitante, car elle n’est pas forcément un bon reflet du tissu d’intérêt. Le traitement de l’échantillon dépendra ensuite de l’objectif : (1) l’étude du niveau d’expression des deux allèles conduirait à recueillir le sang total sur un mélange de stabilisation pour ARN, sensé conserver dans le prélèvement le niveau d’expression physiologique des ARN ; (2) la caractérisation d’un messager anormal ferait préférer un traitement du prélèvement par la puromycine pour se prémunir d’un possible NMD. Cependant, l’approche ARN nécessite un prélèvement et du matériel particuliers. Par ailleurs, il peut être nécessaire d’établir une lignée lymphoblastoïde pour avoir une source d’ARN suffisante, ce qui implique un délai et un surcoût important. De plus, il n’est pas rare d’identifier des transcrits anormaux, dont la réalité in vivo peut être discutable, car induits, par exemple, par les conditions de culture. Enfin, l’anomalie dépistée en ARN doit être caractérisée en génomique, ce qui pose parfois un problème. Pour ces raisons, l’approche ARN n’est pas adaptée aux exigences d’un diagnostic de routine de première intention. Aujourd’hui, cette stratégie est surtout rapportée pour de petites séries, dans le cadre de la recherche. |
Conclusions et perspectives En raison de leur forte contribution au spectre mutationnel, les anomalies de la transcription prennent une place sans cesse grandissante en clinique, tant du point de vue de leur fréquence que de leur variété. Aujourd’hui, leur identification dans le cadre diagnostique, en génétique constitutionnelle, débutera raisonnablement par une approche sur ADN génomique qui sera, en fonction des résultats obtenus, suivie ou non d’une étude ARN. L’indication d’une étude ARN est à discuter devant la mise en évidence d’un unknown variant d’interprétation délicate et devant la non-mise en évidence d’anomalies génomiques. Le contexte clinique et les résultats de l’analyse in silico permettent d’éclairer la stratégie et l’interprétation du biologiste. Les anomalies de la transcription représentent un exemple parfait d’une problématique se situant à la frontière du médical et du fondamental. En effet, de nouveaux mécanismes de régulation de l’expression sont régulièrement découverts, qu’il faudra un jour faire basculer dans le domaine diagnostique. Ainsi, des travaux récents montrent que l’expression d’un gène peut être éteinte par méthylation, elle-même induite par la transcription d’un ARN antisens [
28]. La régulation de l’expression génique serait sous le contrôle de nombreuses séquences en cis [
29] dont la nature et les altérations seront importantes à caractériser en diagnostic. Par exemple, l’insertion de séquences LINE1 complètes dans les introns supprimerait l’expression du gène concerné. Connaissant la fréquence de ces séquences dans le génome, il pourrait s’agir d’un mécanisme de régulation commun [
30] dont le dysfonctionnement, en conséquence, représenterait un mécanisme délétère important. Le biologiste se doit également d’accompagner l’évolution des outils de bio-informatique dont l’apport va grandissant dans l’interprétation des anomalies moléculaires et protéiques. Enfin, au-delà de la transcription, il faut se pencher sur les mécanismes de régulation post-transcriptionnelle dont l’implication en pathologie humaine peut parfois être appréhendée au niveau génomique [
31]. |
Footnotes |
1. Stenson PD, Ball EV, Mort M, et al. Human gene mutation database (HGMD) : 2003 update. Hum Mutat 2003; 21 : 577–81. 2. Ars E, Serra E, Garcia J, et al. Mutations affecting mRNA splicing are the most common molecular defects in patients with neurofibromatosis type 1. Hum Mol Genet 2000; 9 : 237–47. 3. Teraoka SN, Telatar M, Becker-Catania S, et al. Splicing defects in the ataxia-telangiectasia gene, ATM : underlying mutations and consequences. Am J Hum Genet 1999; 64 : 1617–31. 4. Cooper TA, Mattox W. The regulation of splice-site selection, and its role in human disease. Am J Hum Genet 1997; 61 : 259–66. 5. Cartegni L, Chew SL, Krainer AR. Listening to silence and understanding nonsense : exonic mutations that affect splicing. Nat Rev Genet 2002; 3 : 285–98. 6. Yang Y, Swaminathan S, Martin BK, Sharan SK. Aberrant splicing induced by missense mutations in BRCA1 : clues from a humanized mouse model. Hum Mol Genet 2003; 12 : 2121–31. 7. Cartegni L, Krainer AR. Disruption of an SF2/ASF-dependent exonic splicing enhancer in SMN2 causes spinal muscular atrophy in the absence of SMN1. Nat Genet 2002; 30 : 377–84. 8. Kashima T, Manley JL. A negative element in SMN2 exon 7 inhibits splicing in spinal muscular atrophy. Nat Genet 2003; 34 : 460–3. 9. Cogan JD, Prince MA, Lekhakula S, et al. A novel mechanism of aberrant pre-mRNA splicing in humans. Hum Mol Genet 1997; 6 : 909–12. 10. Pagani F, Stuani C, Tzetis M, et al. New type of disease causing mutations : the example of the composite exonic regulatory elements of splicing in CFTR exon 12. Hum Mol Genet 2003; 12 : 1111–20. 11. Chao HK, Hsiao KJ, Su TS. A silent mutation induces exon skipping in the phenylalanine hydroxylase gene in phenylketonuria. Hum Genet 2001; 108 : 14–9. 12. Hefferon TW, Groman JD, Yurk CE, Cutting GR. A variable dinucleotide repeat in the CFTR gene contributes to phenotype diversity by forming RNA secondary structures that alter splicing. Proc Natl Acad Sci USA 2004; 101 : 3504–9. 13. Buratti E, Brindisi A, Pagani F, Baralle FE. Nuclear factor TDP-43 binds to the polymorphic TG repeats in CFTR intron 8 and causes skipping of exon 9 : a functional link with disease penetrance. Am J Hum Genet 2004; 74 : 1322–5. 14. Sakai T, Ohtani N, McGee TL, et al. Oncogenic germ-line mutations in Sp1 and ATF sites in the human retinoblastoma gene. Nature 1991; 353 : 83–6. 15. Ohtani-Fujita N, Fujita T, Takahashi R, et al. A silencer element in the retinoblastoma tumor-suppressor gene. Oncogene 1994; 9 : 1703–11. 16. Price P, Wong AM, Williamson D, et al. Polymorphisms at positions -22 and -348 in the promoter of the BAT1 gene affect transcription and the binding of nuclear factors. Hum Mol Genet 2004; 13 : 967–74. 17. Liu L, Dilworth D, Gao L, et al. Mutation of the CDKN2A 5’ UTR creates an aberrant initiation codon and predisposes to melanoma. Nat Genet 1999; 21 : 128–32. 18. Cazzola M, Skoda RC. Translational pathophysiology : a novel molecular mechanism of human disease. Blood 2000; 95 : 3280–8. 19. Marlin S, Blanchard S, Slim R, et al. Townes-Brocks syndrome : detection of a SALL1 mutation hot spot and evidence for a position effect in one patient. Hum Mutat 1999; 14 : 377–86. 20. Bedell MA, Jenkins NA, Copeland NG. Good genes in bad neighbourhoods. Nat Genet 1996; 12 : 229–32. 21. Pfeifer D, Kist R, Dewar K, et al. Campomelic dysplasia translocation breakpoints are scattered over 1 Mb proximal to SOX9 : evidence for an extended control region. Am J Hum Genet 1999; 65 : 111–24. 22. Holbrook JA, Neu-Yilik G, Hentze MW, Kulozik AE. Nonsense-mediated decay approaches the clinic. Nat Genet 2004; 36 : 801–8. 23. Roca X, Sachidanandam R, Krainer AR. Intrinsic differences between authentic and cryptic 5’ splice sites. Nucleic Acids Res 2003; 31 : 6321–33. 24. Harland M, Mistry S, Bishop DT, Bishop JA. A deep intronic mutation in CDKN2A is associated with disease in a subset of melanoma pedigrees. Hum Mol Genet 2001; 10 : 2679–86. 25. Abkevich V, Zharkikh A, Deffenbaugh AM, et al. Analysis of missense variation in human BRCA1 in the context of interspecific sequence variation. J Med Genet 2004; 41 : 492–507. 26. Messiaen LM, Callens T, Mortier G, et al. Exhaustive mutation analysis of the NF1 gene allows identification of 95 % of mutations and reveals a high frequency of unusual splicing defects. Hum Mutat 2000; 15 : 541–55. 27. Andreutti-Zaugg C, Scott RJ, Iggo R. Inhibition of nonsense-mediated messenger RNA decay in clinical samples facilitates detection of human MSH2 mutations with an in vivo fusion protein assay and conventional techniques. Cancer Res 1997; 57 : 3288–93. 28. Tufarelli C, Stanley JA, Garrick D, et al. Transcription of antisense RNA leading to gene silencing and methylation as a novel cause of human genetic disease. Nat Genet 2003; 34 : 157–65. 29. Wittkopp PJ, Haerum BK, Clark AG. Evolutionary changes in cis and trans gene regulation. Nature 2004; 430 : 85–8. 30. Han JS, Szak ST, Boeke JD. Transcriptional disruption by the L1 retrotransposon and implications for mammalian transcriptomes. Nature 2004; 429 : 268–74. 31. Hudder A, Werner R. Analysis of a Charcot-Marie-Tooth disease mutation reveals an essential internal ribosome entry site element in the connexin-32 gene. J Biol Chem 2000; 275 : 34586–91. 32. Bulyk ML. Computational prediction of transcription-factor binding site locations. Genome Biol 2003; 5 : 201. 33. Wasserman WW, Sandelin A. Applied bioinformatics for the identification of regulatory elements. Nat Rev Genet 2004; 5 : 276–87. |