| |
| Med Sci (Paris). 2005 March; 21(3): 253–260. Published online 2005 March 15. doi: 10.1051/medsci/2005213253.
Épissage alternatif, pathologie et thérapeutique moléculaire
Laurent Corcos1* and Stéphanie Solier2,3 1 Inserm U.613, Faculté de Médecine, 22, avenue Camille Desmoulins, 29238 Brest Cedex 3, France 2Inserm U.517, Faculté de Médecine et de Pharmacie, 7, boulevard Jeanne d’Arc, 21000 Dijon 3Hématologie clinique, CHRU Le Bocage, 2, boulevard Jeanne d’Arc, 21000 Dijon, France |
L’épissage des pré-ARN messagers concerne au moins 95 % des transcrits et constitue un préalable à l’exportation des messagers matures dans le cytoplasme, siège de la synthèse protéique. Il s’effectue de façon coordonnée avec la transcription et se déroule en deux étapes successives impliquant deux réactions de transestérification. Trois déterminants de séquence sont requis (Figure 1A) : les sites d’épissage 5’ (site donneur) et 3’ (site accepteur) situés aux jonctions exon-intron, respectivement en 5’ et 3’ de l’intron, et le site de branchement en amont du site 3’ accepteur. Cette réaction (Figure 1B) a lieu au sein du splicéosome, un complexe dont l’assemblage et le fonctionnement requièrent plus de 150 polypeptides et cinq ribonucléoprotéines, les snRNP (small nuclear ribonucleoproteins), constituées d’une molécule d’ARN, l’ARNsn (snRNA), complexée avec sept protéines Sm ou apparentées et quelques autres protéines spécifiques [
1]. Le splicéosome a deux fonctions : la reconnaissance des sites d’épissage et les étapes couplées d’élimination des introns et de re-ligation des exons. Plusieurs familles de protéines jouent un rôle majeur parmi lesquelles les protéines SR (riches en sérines et en arginines), hnRNP (heterogenous nuclear ribonucleoproteins) et CELF (CUG-binding protein and embryonic lethal abnormal vision-type RNA-binding protein 3-like factors) [
2] (Figure 2). Un répertoire de séquences cis joue également un rôle déterminant lors de l’épissage [
3] (Figure 2).
 | Figure 1.
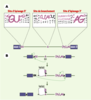
Épissage des pré-ARN messagers. A. Profil des séquences consensus au site d’épissage 5’, au site de branchement et au site d’épissage 3’. À chaque position, la fréquence des 4 nucléotides A, C, G et T est représentée par la taille des caractères. La fréquence a été déterminée à partir de 1 683 séquences humaines [
44]. Les séquences les plus conservées sont les dinucléotides GU et AG, respectivement en 5’ et en 3’ de l’intron, et le nucléotide A au niveau du site de branchement. Les rectangles représentent les exons, et les traits pleins les introns. (Py) n : étendue de polypyrimidines. B. Réaction d’excision-épissage d’un pré-ARNm. L’épissage d’ARN prémessager se déroule en deux étapes successives, impliquant chacune une réaction de trans-estérification. Trois éléments sont essentiels à ces réactions : les sites d’épissage 5’ (ou site donneur) et 3’ (ou site accepteur), situés à la jonction exon-intron, respectivement en 5’ et 3’ de l’intron, et le site de branchement, en amont du site 3’ accepteur. Lors de la première étape, le groupement 2’ hydroxyle de l’adénosine du site de branchement attaque le phosphate du site d’épissage 5’. Cette réaction engendre un exon 5’ libre et un lariat intermédiaire. Lors de la deuxième étape, le 3’ hydroxyle de l’exon 5’ attaque le phosphate du site d’épissage 3’, produisant ainsi l’ARNm et le lariat d’intron. |
 | Figure 2.
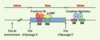
Schéma positionnant les principales protéines sur le pré-ARNm, ainsi que les séquences en cis les plus déterminantes pour la réaction d’épissage. La fonction des protéines SR (serine-rich) (représentées en orange) peut être réglée au niveau transcriptionnel, en fonction du tissu ou du stade de développement, par épissage alternatif, par phosphorylation-déphosphorylation, ce qui influence leur localisation subcellulaire et leur activité [
45]. Elles sont stockées dans des compartiments du noyau appelés speckles ou SFC (splicing factors compartments) [
46]. Elles doivent être recrutées par le complexe d’épissage et dirigées à la périphérie des SFC vers les sites de transcription. Une fois engagées dans le splicéosome, elles sont les cibles de phosphatases Ser/Thr, PP1, PP2A, PP2Cγ, dont certaines font partie intégrante du splicéosome [
47]. Les protéines hnRNP (heterogeneous nuclear ribonucleoproteins) (représentées en jaune) figurent parmi les protéines les plus abondantes du noyau ; certaines d’entre elles sont confinées dans le nucléoplasme (hnRNP C et U), tandis que d’autres (hnRNP A, B, K, J) font la navette entre le noyau et le cytoplasme [
48]. L’ESE (exonic splicing enhancer) (représenté en violet au niveau de l’exon) est le siège de nombreuses mutations qui modifient le site de liaison de protéines SR et conduisent à l’exclusion d’exon. Dans le cas de la myopathie de Becker, la mutation non-sens Glu1211X, touchant un ESE, conduit à une manifestation morbide modérée, tandis que la plupart des autres mutations non-sens entraînent la production de protéines tronquées, à l’origine d’un phénotype très sévère. Un algorithme de prédiction d’ESE fonctionnels est disponible en ligne ( http://exon.cshl.edu/ESE/index.html). L’ESS (exonic splicing silencer) (représenté en vert au niveau de l’exon) agit, entres autres, par liaison à hnRNP I (ou PTB) et hnRNP A/B ou H. HnRNP A1 peut également induire la répression par liaison à un site exonique et par le recrutement coopératif d’autres hnRNP, ce qui neutralise l’activité d’enhancers proximaux, voire l’assemblage du splicéosome. L’ISE (intronic splicing enhancer) et l’ISS (intronic splicing silencer) (représentés respectivement en violet ou en vert au niveau de l’intron) auraient des rôles antagonistes, lesquels peuvent s’exercer au sein d’éléments introniques bipartites. |
En 2004, environ 9,5 % des mutations répertoriées dans HGMD (human gene mutation database) affecteraient l’épissage (http://archive.uwcm.ac.uk/uwcm/mg/ docs/hohoho.html). Les mutations de type I couvrent environ 60 % des mutations et touchent des séquences de sites d’épissage invariantes, ce qui abolit complètement la phase de définition de l’exon [
4]. Les mutations de type II touchent les motifs variables. Dans le cas de la maladie de Menkes (défaut d’absorption hépatique du cuivre), une mutation d’un motif invariant du site donneur de l’exon 6 conduit à une forme très sévère, tandis qu’une délétion de 3 paires de bases (pb) du motif variable du même site donneur d’épissage conduit à une exclusion de l’exon 6 dans une fraction des transcrits et est associée à une forme moins agressive. Une mutation unique dans le dernier nucléotide de l’exon 18 du gène CFTR (cystic fibrosis transmembrane conductance regulator), associée à la mucoviscidose, crée un site cryptique conduisant à l’inclusion d’un nouvel exon de 84 pb. Le niveau de sévérité des atteintes pulmonaire et de l’épididyme est corrélé au taux d’inclusion de cet exon aberrant. |
On estime que 10 000 à 20 000 des gènes humains sont sujets à un épissage alternatif (EA) [
5,
6]. L’analyse de représentativité de fragments de transcrits au sein des banques d’EST (expressed sequence tags) montre, en effet, que chaque ARN prémessager peut faire l’objet, en moyenne, de six à huit épissages distincts, et cela pour l’ensemble des eucaryotes analysés. En outre, une analyse, avec les banques d’EST, de 475 séquences protéiques variantes retrouvées chez des sujets porteurs de maladies suggère qu’au moins un tiers de ces protéines est issu d’un épissage alternatif [
7]. Des données récentes indiquent que 70 % à 88 % des événements d’EA changeraient la nature de la protéine, et seulement 19 % conduiraient à la production de protéines tronquées [
8]. Cette dernière observation résulte en grande partie du fait que l’EA est souvent associé à l’insertion de codons stop prématurés (situés en amont de la position occupée par le codon stop habituellement utilisé), ce qui peut être à l’origine de la production de protéines tronquées, mais surtout du mécanisme de nonsense-mediated mRNA decay [
9]. Ce mécanisme est en effet très souvent observé si le codon stop est situé à plus de 50-55 nucléotides en amont de la dernière jonction exon-exon, et conduit à diminuer considérablement la stabilité des transcrits correspondants, donnant ainsi rarement lieu à la synthèse d’une protéine abondante. Ce mécanisme souffre de peu d’exceptions et est interprété comme une surveillance physiologique destinée à prévenir la production de protéines tronquées potentiellement délétères. Enfin, l’épissage alternatif peut être influencé par la transcription [
10,
11]. Les différents types d’EA sont illustrés sur la Figure 3.
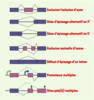 | Figure 3.
Représentation des différents types d’épissage alternatif. En bleu les exons constitutifs, en violet les exons alternatifs, et en traits pleins les introns. Différentes combinaisons d’assemblage des exons, voire de rétention d’intron sont possibles. En outre, l’utilisation de promoteurs transcriptionnels peut dicter les choix d’épissage. Enfin, l’épissage alternatif peut conduire à l’utilisation de signaux de sites de polyadénylation distincts. |
L’analyse de maladies d’origine génétique a été longtemps focalisée sur la recherche de mutations affectant la région codante des protéines. Plus récemment, un très large ensemble de mutations régulatrices a été répertorié. Ces mutations sont associées à la production de transcrits moins abondants, aberrants ou nouveaux, codant ou non pour des protéines normales ou modifiées. La majorité des mutations morbides touchent davantage les séquences de régulation que les régions codantes [2,
12,
13] et affectent également des protéines d’épissage [
14] (Tableaux I et II).
 | Tableau I.
Exemples de gènes portant des mutations d’épissage associées à des maladies chez l’homme (adapté de [ 13]). * OMIM : online Mendelian inheritance in man ( http://www.ncbi.nlm.nih.gov/entrez/query.fcgi ?db=OMIM). |
 | Tableau II.
Exemples de gènes de protéines d’épissage impliquées dans des maladies chez l’homme (adapté de [ 14]). |
|
Identification des événements d’épissage alternatif Une étape déterminante dans la compréhension des mécanismes et du rôle de l’EA tient à l’identification, in extenso, des événements d’épissage. Des algorithmes spécifiques ont été développés et plusieurs approches expérimentales à haut débit ont été explorées (Figure 4). L’utilisation de sondes oligonucléotidiques fixées sur des filtres de forte densité a permis l’hybridation d’ARNm à des taux reflétant la spécificité tissulaire d’expression des gènes cibles [
15]. Des méthodes plus sophistiquées utilisant des fibres optiques ont également été proposées [
16]. D’autres méthodes destinées à identifier l’ensemble des transcrits alternatifs d’un gène donné ont été également rapportées [
17,
18]. Un ensemble de sites web dédiés à l’EA est listé par L.F. Larreau et al. [
19].
 | Figure 4.
Identification des événements d’épissage alternatif. A. Utilisation de sondes oligonucléotidiques fixées sur des filtres à forte densité. Dans cet exemple, le pré-ARNm contient 4 exons, dont un exon alternatif (E2 en violet). Cinq types de sondes sont utilisées : 1, 2, 3, 1-2, 1-3 (par exemple, la sonde 1 s’hybride au niveau de l’exon 1, la sonde 1-2 s’hybride au niveau de la jonction entre exon 1 et exon 2…). Les sondes oligonucléotidiques sont fixées sur des filtres par leur région 3’ (symbolisée par un rond noir). Les ARN ou les produits de RT-PCR marqués sont ensuite hybridés sur ces filtres. L’ARN qui contient les exons 1, 2 et 3 s’est hybridé avec les sondes 1, 2, 3, et 1-2, tandis que l’ARN ne contenant pas l’exon 2 s’est hybridé avec les sondes 1, 3, et 1-3. L’hybridation est représentée par le petit ovale jaune. B. Hybridation moléculaire. À partir d’ADN génomique, une série de sondes (flèches vertes) est produite. Elles sont ensuite hybridées avec des populations d’ARNm ou d’ADN complémentaire. L’hybridation n’a lieu qu’avec les séquences exoniques (adapté de [ 15]). |
|
Exemples de maladies dues à des défauts d’épissage Maladies d’origine génétique La démence frontotemporale et le parkinsonisme liés au chromosome 17 sont dus à des mutations du gène MAPT (microtubuleassociated protein tau) codant pour la protéine tau requise pour l’assemblage des microtubules. Des événements d’EA touchant les exons 2, 3 et 10 conduisent à la production d’isoformes aux extrémités amino- et carboxyterminales variables. Le rapport des isoformes de tau 4R (présence de l’exon 10)/3R (absence de l’exon 10) est déterminant pour spécifier le phénotype (il est d’environ 1 dans le cerveau normal) ; ce rapport est étroitement contrôlé par l’activité d’un ensemble de séquences ESE (exonic splicing enhancer), ESS (exonic splicing silencer), ISS (intronic splicing silencer) et une structure en épingle qui piège le site 5’ d’épissage. Les amyotrophies spinales (spinal muscular atrophy, SMA) sont dues à des mutations du gène SMN (survival motoneuron, SMN1). Quatre-vingt-treize pour cent des patients sont porteurs d’une délétion homozygote qui emporte au moins l’exon 7, le reste des patients ayant une délétion hétérozygote avec présence sur l’autre allèle d’une mutation non-sens ou faux-sens. Ce gène est dupliqué, et une copie très homologue (SMNc ou SMN2) est présente chez 95 % de la population [3]. Le type 2 de la dystrophie myotonique est dû à une expansion de séquences CCTG dans l’intron 1 du gène ZNF9. C’est la longueur, dans l’ARN, du nombre de répétitions de CUG ou CCUG qui spécifie le degré de l’atteinte. Ces expansions de séquences seraient responsables d’une perte de l’activité d’épissage due à l’activation de la liaison spécifique de protéines des familles mbnl (muscleblind) sur ces séquences, conduisant à un phénomène de séquestration de ces protéines sur ces sites. Un phénotype comparable est d’ailleurs obtenu chez des souris dont le gène mbnl a été invalidé. Ce recrutement protéique incorrect conduirait à des épissages aberrants de plusieurs transcrits dont la troponine T cardiaque, le récepteur de l’insuline, le canal chlore spécifique du muscle, la protéine tau et une protéine apparentée à la myotubularine. Des mutations inactivant l’assemblage ou la fonction des snRNP ont également été trouvées. Dans le cas de la rétinite pigmentaire, trois gènes codant pour des protéines du splicéosome, responsables de formes autosomiques dominantes, ont été identifiés (PRPF31, HPRP3 et PRPC8). Les mutations de PRPF31 incluent des insertions, des délétions, des mutations faux-sens et des mutations de sites d’épissage. La protéine correspondante est associée aux snRNP U4/U6 et U5 dont elle permet la trimérisation. La protéine HPRP3 est également un composant du complexe U4/U6 snRNP, au sein duquel elle serait capable de recruter HPRP4. Les mutations d’HPRP3 associées à des cas sporadiques de la maladie sont faux-sens ou non-sens dans deux codons conservés de l’exon 11. La protéine PRPC8 est également un constituant du splicéosome et le siège d’un ensemble de mutations toutes regroupées dans une région très conservée du dernier exon. Modifications d’épissage associées à la tumorigenèse Plusieurs observations ont permis d’associer des modifications d’épissage à la tumorigenèse [ 3]. D’une part, l’abondance relative de protéines SR augmente entre les stades prénéoplasique et métastatique dans un modèle de tumorigenèse mammaire chez la souris [
20]. D’autre part, la transition, par augmentation de l’épissage alternatif, d’une forme de faible affinité vers une forme de forte affinité du récepteur 1 du FGF ( fibroblast growth factor), est associée à la progression tumorale des astrocytomes [
21]. Cet EA est favorisé en présence de la protéine PTB (polypyrimidine tract binding protein) dont le taux est augmenté. Le gène CD44 est impliqué dans l’adhérence et la migration cellulaires et peut coder pour des formes variantes de CD44, CD44v, associées à de nombreux cancers [
22]. Le gène suppresseur de tumeur BRCA1 code pour une protéine nucléaire, mais, dans certains cancers du sein, un variant d’épissage dépourvu de l’exon 11 code pour une protéine dépourvue de signal de localisation nucléaire, suggérant la possibilité d’une perte de fonction de la protéine [
23]. |
Compensations moléculaires La meilleure compréhension des mécanismes de l’EA et l’identification des transcrits qu’il produit ont permis de développer des stratégies visant à modifier le taux de transcrits alternatifs, pour favoriser la synthèse de protéines « thérapeutiques » ou bloquer la production de protéines « indésirables » [14,
24]. Surexpression de protéines stimulant l’épissage Les surexpressions de hnRNPA1 et du facteur d’épissage adénoviral E4-ORF6 permettent l’exclusion de l’exon 18 inséré dans un minigène CFTR, conduisant à une surproduction de transcrits normaux dépourvus de l’exon 18 [
25]. La surexpression du facteur d’épissage Htra2-b1 permet une surproduction de transcrits normaux (incluant l’exon 7) du gène SMN2, de nature à complémenter le défaut chez les patients SMA porteurs d’allèles délétères du gène SMN1 [
26]. Les protéines SC35 et hnRNPA1 ont des effets opposés sur l’inclusion de l’exon 9 dans un minigène Casp-2 et sur l’induction de l’apoptose [
27]. Modification d’activité des protéines d’épissage L’inhibition pharmacologique de la kinase Clk1/Sty, qui phosphoryle les protéines SR et augmente l’activité de la protéine d’épissage ASF/SF2 (alternative splicing factor/splicing factor 2), modifie l’activité d’épissage de transcrits sélectionnés [
28]. Des composés synthétiques de type PNA (peptide nucleic acid) sont capables de recruter la machinerie d’épissage [
29], et le butyrate ou le valproate de sodium induisent une augmentation du taux des transcrits SMN2, aboutissant à une rémission partielle dans un modèle murin d’atrophie musculaire spinale caractérisée par une perte d’expression, en rapport avec une mutation, du gène SMN1 [
30]. L’aclarubicine, un inhibiteur de la topo-isomérase II, induit une redistribution des protéines SR, ce qui stimule également l’inclusion de l’exon 7 du gène SMN2 [
31]. Des inhibiteurs des topo-isomérases I (camptothécine) et II (étoposide) induisent l’EA (en faveur de l’inclusion de l’exon 9) du pré-ARNm codant pour la pro-caspase-2 [
32,
33]. En outre, les hormones stéroïdiennes stimulent également l’EA par l’intermédiaire du recrutement de transactivateurs spécifiques [
34]. Blocage ou facilitation d’épissage avec des oligonucléotides Des oligonucléotides destinés à inhiber l’inclusion de l’exon 23 muté du gène de la dystrophine dans le modèle murin des souris mdx (porteuses d’une mutation non-sens dans l’exon 23 du gène de la dystrophine, aboutissant à une protéine tronquée qui ne peut se localiser correctement au niveau du sarcolemme des fibres musculaires) permettent la synthèse et la localisation correctes de la dystrophine [
35]. Récemment, une restauration phénotypique complète de fonction de la dystrophine a été obtenue chez ces souris, après injection de virus AAV ( adeno-associated virus), permettant la production d’un transcrit élaboré de façon à induire l’exclusion de l’exon 23 porteur de la mutation et correspondant à une région répliquée non essentielle à la fonction de la protéine [
36]. Des oligonucléotides antisens bifonctionnels, couvrant un domaine de ciblage couplé à une partie peptidique (PNA) contenant des ESE constituées de répétitions arginine-sérine, ont été utilisés [
37,
38]. Le ciblage des transcrits BRCA1 et SMN2 par la méthode ESSENCE (exon-specific splicing enhancement by small chimeric effectors) a été réalisé avec succès [ 38]. Une variante de cette méthode, TOES (targeted oligonucleotide enhancer of splicing), consistant en l’inclusion d’un domaine effecteur contenant une séquence cible de fixation de protéines d’épissage comme ASF/SF2, a permis le traitement de fibroblastes de patients SMA, avec un degré de correction (inclusion de l’exon 7 du gène SMN2) de l’ordre de 70 % [ 14]. Une approche similaire a été réalisée avec un oligonucléotide bifonctionnel capable de recruter la protéine hnRNPA1 et d’augmenter le rapport transcrits Bcl-xS/transcrits Bcl-xL, dans le but de promouvoir le potentiel pro-apoptotique [
39] (Figure 5). L’interférence par l’ARN s’est également montrée efficace [
40].
 | Figure 5.
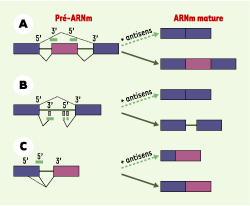
Blocage ou facilitation d’épissage avec des oligonucléotides dirigés contre des motifs d’épissage Les oligonucléotides antisens sont représentés en vert. Ils permettent l’exclusion d’un exon (A), bloquent les sites cryptiques d’épissage 5’ ou 3’ aberrants (B), forcent la sélection d’un site d’épissage 5’ particulier (C). En bleu les exons constitutifs, en violet les exons alternatifs, en traits pleins les introns (exemples des gènes β -globine et CFTR [
49], du gène DMD [
50] et du gène Bcl-X [
51]) (adapté de [ 49]). |
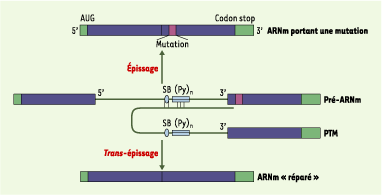
Le trans-épissage Cette approche, appelée SMART (spliceosome-mediated RNA transsplicing), repose sur le ciblage des pré-ARNm [ 14] (Figure 6). Elle a permis la correction du saignement, associé à la production de facteur VIII, dans un modèle de souris hémophiles [
41]. Le trans-épissage a été également utilisé pour cibler des transcrits matures avec des ribozymes de groupe I [
42]. Une troisième approche repose sur l’utilisation d’une endonucléase d’épissage des ARNt. L’endonucléase permet le clivage de l’ARN cible et de l’ARN correcteur apporté, avant que les étapes de cis- et de trans-épissage ne se déroulent. L’expression de l’endonucléase et de la séquence de ciblage a permis de reprogrammer un gène cellulaire dans des fibroblastes de souris [
43]. 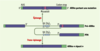 | Figure 6.
Le trans-épissage. Dans cet exemple, le pré-ARNm comprend deux exons, et porte une mutation (représentée en violet) dans le deuxième exon, conduisant à une protéine non fonctionnelle. L’utilisation de PTM (pre-trans-splicing molecule) permet la suppression de la mutation en remplaçant l’exon muté. Le PTM se lie au pré-ARNm par appariement de séquences, ce qui masque en partie le site 3’ d’épissage. La réaction d’épissage normale est donc bloquée et le domaine de trans-épissage du PTM peut jouer le rôle d’un site 3’ d’épissage. Le PTM apporte l’exon dépourvu de mutation, ce qui permet la réparation de l’ARNm et la production d’une protéine fonctionnelle. (Py)n : étendue de polypyrimidines, SB : site de branchement. |
À la lumière de ces différentes approches, il est concevable d’imaginer des combinaisons entre ces stratégies : par exemple, le cumul d’agents pharmacologiques agissant sur le taux ou l’activité des protéines SR et topo-isomérases avec des expériences de trans-épissage. La sélection forcée de promoteurs transcriptionnels, par l’utilisation de ligands de récepteurs nucléaires, pourrait également constituer une approche prometteuse permettant de diriger des épissages alternatifs spécifiques [34]. |
Outre l’identification de nouvelles cibles thérapeutiques potentielles, la recherche systématique des événements d’EA pourrait permettre d’obtenir des canevas de référence quant au caractère prédictif potentiel, par exemple, d’un état précancéreux, ou de l’efficacité d’un traitement. Les possibilités nouvelles évoquées par les succès expérimentaux récents de compensation fonctionnelle suscitent de réels espoirs thérapeutiques. Les difficultés prévisibles appartiennent au registre des étapes requises pour le développement de médicaments. À celles-ci, s’ajoutent les obstacles inhérents aux approches de thérapie génique et cellulaire en termes d’efficacité, de « durabilité », et surtout d’innocuité à long terme. De plus, les « niches » constituées par les maladies génétiques rares risquent de représenter une priorité toute relative pour l’industrie pharmaceutique. En revanche, tous les intervenants en santé humaine seront sans aucun doute très motivés par le défi de l’élaboration de molécules à visée anticancéreuse. C’est probablement dans ce domaine que les attentes seront les plus nombreuses et les plus difficiles à satisfaire.

|
Nous remercions vivement le Dr Jamal Tazi (CNRS, Montpellier) pour sa lecture du manuscrit. Merci également pour leur soutien à l’Inserm, la Ligue Nationale contre le Cancer, la Ligue contre le Cancer de Saône-et-Loire et le Conseil régional de Bourgogne
|
1. Nilsen TW. The spliceosome : the most complex macromolecular machine in the cell ? BioEssays 2003; 25 : 1147–9. 2. Caceres JF, Kornblihtt AR. Alternative splicing : multiple control mechanisms and involvement in human disease. Trends Genet 2002; 18 : 186–93. 3. Faustino NA, Cooper TA. Pre-mRNA splicing and human disease. Genes Dev 2003; 17 : 419–37. 4. Nissim-Rafinia M, Kerem B. Splicing regulation as a potential genetic modifier. Trends Genet 2002; 18 : 123–7. 5. Black DL. Mechanisms of alternative pre-messenger RNA splicing. Annu Rev Biochem 2003; 72 : 291–336. 6. Modrek B, Resch A, Grasso C, Lee C. Genome-wide detection of alternative splicing in expressed sequences of human genes. Nucleic Acids Res 2001; 29 : 2850–9. 7. Hanke J, Brett D, Zastrow I, et al. Alternative splicing of human genes : more the rule than the exception ? Trends Genet 1999; 15 : 389–90. 8. Modrek B, Lee C. A genomic view of alternative splicing. Nat Genet 2002; 30 : 13–9. 9. Nagy E, Maquat LE. A rule for termination-codon position within intron-containing genes : when nonsense affects RNA abundance. Trends Biochem Sci 1998; 23 : 198–9. 10. Proudfoot N. Connecting transcription to messenger RNA processing. Trends Biochem Sci 2000; 25 : 290–3. 11. Logette E, Wotawa A, Solier S, et al. The human caspase-2 gene : alternative promoters, pre-mRNA splicing and AUG usage direct isoform-specific expression. Oncogene 2003; 22 : 935–46. 12. Cartegni L, Chew SL, Krainer AR. Listening to silence and understanding nonsense : exonic mutations that affect splicing. Nat Rev Genet 2002; 3 : 285–98. 13. Brinkman BM. Splice variants as cancer biomarkers Clin Biochem 2004; 37 : 584–94. 14. Garcia-Blanco MA, Baraniak AP, Lasda EL. Alternative splicing in disease and therapy. Nat Biotechnol 2004; 22 : 535–46. 15. Roberts GC, Smith CW. Alternative splicing : combinatorial output from the genome. Curr Opin Chem Biol 2002; 6 : 375–83. 16. Yeakley JM, Fan JB, Doucet D, et al. Profiling alternative splicing on fiber-optic arrays. Nat Biotechnol 2002; 20 : 353–8. 17. Zhu J, Shendure J, Mitra RD, Church GM. Single molecule profiling of alternative pre-mRNA splicing. Science 2003; 301 : 836–8. 18. Jin P, Fu GK, Wilson AD, et al. PCR isolation and cloning of novel splice variant mRNAs from known drug target genes. Genomics 2004; 83 : 566–71. 19. Lareau LF, Green RE, Bhatnagar RS, Brenner SE. The evolving roles of alternative splicing. Curr Opin Struct Biol 2004; 14 : 273–82. 20. Stickeler E, Kittrell F, Medina D, Berget SM. Stage-specific changes in SR splicing factors and alternative splicing in mammary tumorigenesis. Oncogene 1999; 18 : 3574–8. 21. Yamaguchi F, Saya H, Bruner JM, Morrison RS. Differential expression of two fibroblast growth factor-receptor genes is associated with malignant progression in human astrocytomas. Proc Natl Acad Sci USA 1994; 91 : 484–8. 22. Sneath RJ, Mangham DC. The normal structure and function of CD44 and its role in neoplasia. Mol Pathol 1998; 51 : 191–200. 23. Thakur S, Zhang HB, Peng Y, et al. Localization of BRCA1 and a splice variant identifies the nuclear localization signal. Mol Cell Biol 1997; 17 : 444–52. 24. Nissim-Rafinia M, Kerem B. Splicing regulation as a potential genetic modifier. Trends Genet 2002; 18 : 123–7. 25. Nissim-Rafinia M, Chiba-Falek O, Sharon G, et al. Cellular and viral splicing factors can modify the splicing pattern of CFTR transcripts carrying splicing mutations. Hum Mol Genet 2000; 9 : 1771–8. 26. Hofmann Y, Lorson CL, Stamm S, et al. Htra2-beta 1 stimulates an exonic splicing enhancer and can restore full-length SMN expression to survival motor neuron 2 (SMN2). Proc Natl Acad Sci USA 2000; 97 : 9618–23. 27. Wu JY, Tang H, Havlioglu N. Alternative pre-mRNA splicing and regulation of programmed cell death. Prog Mol Subcell Biol 2003; 31 : 153–85. 28. Pilch B, Allemand E, Facompre M, et al. Specific inhibition of serine- and arginine-rich splicing factors phosphorylation, spliceosome assembly, and splicing by the antitumor drug NB-506. Cancer Res 2001; 61 : 6876–84. 29. Cartegni L, Krainer AR. Correction of disease-associated exon skipping by synthetic exon-specific activators. Nat Struct Biol 2003; 10 : 120–5. 30. Chang JG, Hsieh-Li HM, Jong YJ, et al. Treatment of spinal muscular atrophy by sodium butyrate. Proc Natl Acad Sci USA 2001; 98 : 9808–13. 31. Andreassi C, Jarecki J, Zhou J, et al. Aclarubicin treatment restores SMN levels to cells derived from type I spinal muscular atrophy patients. Hum Mol Genet 2001; 10 : 2841–9. 32. Wotawa A, Solier S, Logette E, et al. Differential influence of etoposide on two caspase-2 mRNA isoforms in leukemic cells. Cancer Lett 2002; 185 : 181–9. 33. Solier S, Lansiaux A, Logette E, et al. Topoisomerase I and II inhibitors control caspase-2 pre-messenger RNA splicing in human cells. Mol Cancer Res 2004; 2 : 53–61. 34. Aubœuf D, Honig A, Berget SM, O’Malley BW. Coordinate regulation of transcription and splicing by steroid receptor coregulators. Science 2002; 298 : 416–9. 35. Lu QL, Mann CJ, Lou F, et al. Functional amounts of dystrophin produced by skipping the mutated exon in the mdx dystrophic mouse. Nat Med 2003; 9 : 1009–14. 36. Goyenvalle A, Vulin A, Fougerousse F, et al. Rescue of dystrophic muscle through U7 snRNA-mediated exon skipping. Science 2004; 306 : 1746–9. 37. Skordis LA, Dunckley MG, Yue B, et al. Bifunctional antisense oligonucleotides provide a trans-acting splicing enhancer that stimulates SMN2 gene expression in patient fibroblasts. Proc Natl Acad Sci USA 2003; 100 : 4114–9. 38. Cartegni L, Krainer AR. Correction of disease-associated exon skipping by synthetic exon-specific activators. Nat Struct Biol 2003; 10 : 120–5. 39. Villemaire J, Dion I, Elela SA, Chabot B. Reprogramming alternative pre-messenger RNA splicing through the use of protein-binding antisense oligonucleotides. J Biol Chem 2003; 278 : 50031–9. 40. Celotto AM, Graveley BR. Exon-specific RNAi : a tool for dissecting the functional relevance of alternative splicing. RNA 2002; 8 : 718–24. 41. Chao H, Mansfield SG, Bartel RC, et al. Phenotype correction of hemophilia A mice by spliceosome-mediated RNA trans-splicing. Nat Med 2003; 9 : 1015–9. 42. Rogers CS, Vanoye CG, Sullenger BA, George AL Jr. Functional repair of a mutant chloride channel using a trans-splicing ribozyme. J Clin Invest 2002; 110 : 1783–9. 43. Deidda G, Rossi N, Tocchini-Valentini GP. An archaeal endoribonuclease catalyzes cis- and trans- nonspliceosomal splicing in mouse cells. Nat Biotechnol 2003; 21 : 1499–504. 44. Burge CB, Tuschl T, Sharp PA. Splicing of precursors to mRNAs by the spliceosomes. In : Gesteland RF, Cech TR, Atkins JF, eds. The RNA world II. Cold Spring Harbor-New York : Cold Spring Harbor Laboratory Press, 1999 : 525–60. 45. Sanford JR, Bruzick JP. Developmental regulation of SR protein phosphorylation and activity. Genes Dev 1999; 13 : 1513–8. 46. Mistelli T, Spector DL. The cellular organization of gene expression. Curr Opin Cell Biol 1998; 10 : 323–31. 47. Murray MV. Role of phosphorylation in pre-mRNA splicing. Front Horm Res 1999; 25 : 83–100. 48. Dreyfuss G, Matunis MJ, Piñol-Roma S, Burd CG. HnRNP proteins and the biogenesis of mRNA. Annu Rev Biochem 1993; 63 : 289–321. 49. Sazani P, Kole R. Therapeutic potential of antisense oligonucleotides as modulators of alternative splicing. J Clin Invest 2003; 112 : 481–6. 50. Mann CJ, Honeyman K, Cheng AJ, et al. Antisense-induced exon skipping and synthesis of dystrophin in the mdx mouse. Proc Natl Acad Sci
USA 2001; 98 : 42–7. 51. Mercatante DR, Mohler JL, Kole R. Cellular response to an antisense-mediated shift of Bcl-x pre-mRNA splicing and antineoplastic agents. J Biol Chem 2002; 277 : 49374–82. |