| |
| Med Sci (Paris). 2005 June; 21(6-7): 573–577. Published online 2005 June 15. doi: 10.1051/medsci/2005216-7573.Insubordination et sollicitude du chromosome X humain Simone Gilgenkrantz* 9, rue Basse, 54330 Clereysur- Brenon, France MeSH keywords: Animaux, Chats, Chromosomes humains, Femelle, Humains, Mâle, Chromosome X, Chromosome Y |
Depuis la découverte du mécanisme de l’inactivation, qui se fait au hasard sur l’un des deux X dans les cellules féminines, conférant à la femme un statut de « mosaïque physiologique », le chromosome X nous a réservé bien des surprises. Son séquençage, qui vient d’être publié dans le numéro du 17 mars 2005 de la revue Nature, couplé à une étude sur l’inactivation et sur les gènes échappant à celle-ci, nous montre une fois de plus que le chromosome X, dans sa structure et son comportement, échappe aux règles qui régissent les autosomes et le chromosome Y. |
Le chromosome X enfin séquencé Après celle du chromosome Y, publiée en 2000, la séquence du chromosome X vient de paraître [
1]. Elle a nécessité le travail de plus d’une centaine de chercheurs dans une vingtaine d’équipes coordonnées par Mark Ross au Wellcome Trust Sanger Institute (Hinston, Royaume-Uni). Elle est désormais à la disposition de la communauté scientifique et peut être consultée dans diverses banques de données (Figure 1).  | Figure 1.
La séquence du chromosome X peut être consultée dans diverses banques de données.
|
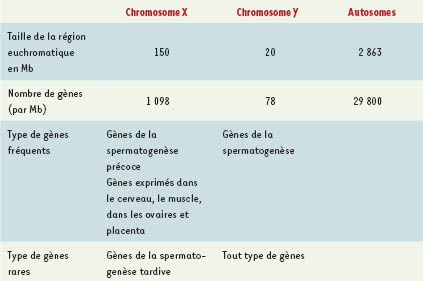
Les 99,3 % de la région euchromatique séquencés contiennent 1 098 gènes. Bien que le chromosome X ne contienne que 4 % de l’ensemble des gènes humains, 10 % des maladies à transmission mendélienne répertoriées concernent des gènes portés par le chromosome X, avec une transmission dite liée à l’X, récessive ou dominante, parfois létale chez le garçon. Les causes moléculaires de 168 phénotypes liés à l’X ont été identifiées et ces recherches sont extrêmement riches en enseignement pour la compréhension de nombreux processus pathologiques, en particulier dans les retards mentaux [
2]. Comparativement aux onze autosomes déjà séquencés (5, 6, 7, 10, 13, 14, 19, 20, 21, 22), le chromosome X est pauvre en gènes, et ceux-ci sont plutôt petits : longueur moyenne de 49 kb contre 57 pour le chromosome 13, par exemple. Pourtant c’est lui qui possède le gène le plus long, le gène DMD codant pour la dystrophine, qui s’étend sur 2 220 223 pb. Concordant avec cette faible densité en gènes, la fréquence des îlots CpG est faible : 5,25 par Mb, ce qui correspond à la moitié de la densité moyenne. Quant aux séquences répétées, elles correspondent à 56% de la séquence euchromatique. Toutefois, leur représentation diffère de celle des autosomes : peu de SINE (short interspersed nuclear element), mais surtout il existe une grande quantité de LINE (long interspersed element) de la famille L1 qui constituent 29% de la séquence de l’X contre 17% en moyenne pour les autosomes et dont l’éventuelle signification sera discutée ultérieurement (Tableau I).  | Tableau I.
La séquence du chromosome X, quelques chiffres.
|
|
Au cours de l’évolution des espèces, la sexualisation a évolué différemment pour les mammifères et les oiseaux. Tous les mammifères (protothériens, euthériens et placentaires) ont deux chromosomes sexuels hétéromorphes X et Y, le mâle étant XY et la femelle XX, alors qu’un phénomène approximativement inverse s’est produit chez les oiseaux avec un mâle ZZ et une femelle ZW (→)
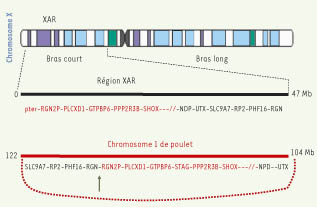
(→) m/s 2004, n° 11, p.1004). Auparavant, c’est-à-dire il y a environ 300 millions d’années, la détermination sexuelle dépendait d’événements environnementaux, comme la température d’incubation des œufs, par exemple. Puis, selon la loi de Susumu Ohno, le père de l’inactivation de l’X (1959), les régions concernant la détermination du sexe se seraient concentrées sur une paire d’autosomes et une barrière se serait établie entre eux, empêchant les recombinaisons. À la suite de mutations successives, le protochromosome Y aurait perdu en taille et en contenu génétique alors que le protochromosome X conservait sa taille ainsi que la plupart de ses gènes. La comparaison de séquences avec le génome du poulet, désormais possible, vient confirmer ces hypothèses et fournir des notions complémentaires : la quasi totalité des séquences codantes des bras longs du chromosome X correspond à une région de 20 Mb environ sur les bras courts du chromosome 4 du poulet. La plupart des séquences des bras courts de l’X (Xp11.3-pter), y compris la région PAR1 (pseudoautosomique), correspondent à un segment de 1q aviaire. De plus, une région dite XAR (X added region) a été ajoutée il y a environ 150 millions d’années sur l’X des mammifères euthériens, avant leur radiation. D’après la disposition des gènes chez le poulet, il semble que cette région résulte de deux réarrangements ainsi que d’une translocation, à moins qu’on accepte l’hypothèse d’un modèle plus parcimonieux : la recombinaison de l’X avec un chromosome en anneau (Figure 2).  | Figure 2.
Homologie entre la région XAR de l’X humain et le chromosome 1 du poulet. Un chromosome en anneau hypothétique est représenté avec l’ordre des gènes observé chez le poulet par l’arc de cercle en rouge. Une recombinaison d’un tel anneau à l’extrémité du chromosome X en un point indiqué par la flèche verte pourrait expliquer la formation de la zone XAR en une seule étape (d’après [
5]). |
Par la suite, l’évolution s’est poursuivie chez les mammifères. L’étude des homologies de séquences entre l’X canin et l’X humain montre une linéarité presque complète. En revanche, la différence notable entre l’X murin et l’X humain – absence de 9 Mb sur l’X des rongeurs d’une région Xp comprenant la région PAR1 – résulte, selon toute probabilité, d’une délétion survenue ultérieurement dans cette espèce. La comparaison de l’homologie entre X et Y avait permis de conclure à quatre régions physiques correspondant à des étapes évolutives, et donc interprétées comme quatre strates. Mais une réévaluation des strates 3 et 4 a montré qu’une partie distale correspondrait à une cinquième strate dont la formation a dû se produire il y a environ 30 millions d’années. |
Les relations du couple X et Y Depuis leur individualisation, l’histoire évolutive des chromosomes sexuels a différé notablement de celle des autosomes. Le chromosome Y a subi une pression sélective lui conférant presque exclusivement des fonctions de différenciation masculine et testiculaires. L’analyse de la région mâle spécifique (MSY) montre qu’il contient plusieurs douzaines de gènes absents sur l’X [
3]. De même, chez tous les mammifères, l’X a aussi concentré des gènes bénéfiques pour les femelles, tels que ceux s’exprimant dans les ovaires ou dans le placenta, en même temps qu’il s’appauvrissait en gènes intervenant plus tardivement dans la spermatogenèse. Mais la pression de sélection qui s’est exercée sur l’X est plus subtile. Comme il est présent à l’état hémizygote chez les mâles, le chromosome X a dû sélectionner des gènes récessifs bénéfiques pour eux, car se manifestant plus facilement que sur les autosomes. Viennent en premier lieu les gènes de la famille MAGE (melanoma antigen) qui codent pour des antigènes de tumeurs reconnus in vitro par des lymphocytes T. Alors qu’il en existe seulement 4 dans l’ensemble du génome, le chromosome X en possède 32 à lui tout seul. Membres du groupe codant pour des antigènes de cancer testiculaire (CT), ils sont exprimés dans divers cancers. Ces antigènes devraient fournir des cibles de choix pour la production de vaccins puisqu’ils sont reconnus par le système immunitaire. Dans les tissus normaux, leur expression est presque exclusivement testiculaire. Une telle concentration de gènes codant pour des antigènes CT sur le chromosome X humain doit être le reflet de cette tendance à retenir les allèles récessifs favorables aux mâles au cours de l’évolution. Il est à noter que l’X murin a, lui aussi, un nombre important de gènes MAGE. Mais le chromosome X humain semble aussi enrichi en gènes s’exprimant dans le cerveau et dans les muscles, favorables aux mâles en ce sens qu’ils s’exprimeraient mieux à l’état hémizygote. Donc, en considérant la constitution actuelle des gonosomes, il semble qu’ils ont évolué sous l’effet de forces antagonistes (Tableau II) et que le chromosome X s’est constamment révélé plein de sollicitude à l’égard de la masculinité [
4, 5].  | Tableau II.
Étude comparative montrant les forces évolutives affectant le contenu en gènes des gonosomes.
|
|
L’inactivation de l’X : une discrimination négative L’inactivation d’un des deux X se produit chez les femelles de mammifères, ce qui équilibre le dosage génique entre mâles (hémizygotes) et femelles. Mais les phénomènes épigénétiques qui se produisent au cours de l’installation et du maintien de l’inactivation ne sont pas encore complètement connus. Dans les embryons femelles, il est initié très tôt par le centre inactivatieur (XIC) et le gène XIST (X
inactive specific transcrit). Ce gène fait partie des gènes d’ARN non codant qui sont estimés, d’après plusieurs approches complémentaires, à 173 sur le chromosome X. Il contrôle l’inactivation de l’X chez la femme. Situé en Xq13, il comporte 32 103 pb. Son transcrit non traduit est particulièrement long et agit en cis. Il existe cependant des transcrits plus courts résultant d’épissages alternatifs, surtout dans la région 3’ du gène. Les transcrits du gène XIST ne deviennent stables que sur l’un des deux X. Ils revêtent l’ensemble du chromosome et entraînent l’extinction transcriptionnelle des gènes. Mais ce phénomène, nous le verrons plus loin, comporte beaucoup d’exceptions. Pour propager l’inactivation tout le long des 155 Mb de l’X, des relais existent sans doute. L’hypothèse d’une participation des séquences LINE à ces relais est renforcée par la manière dont ces L1 sont réparties : nombreuses dans les régions où les gènes sont majoritairement éteints, elles deviennent plus rares dans les régions où les gènes échappent à l’inactivation. Dans la région XAR, ajoutée tardivement, leur fréquence n’est pas plus élevée que dans les autosomes. |
Le caractère imprévisible du profil d’inactivation de l’X À chaque étape de l’étude de l’inactivation, force est de constater que le chromosome X échappe à des règles simples. On sait depuis longtemps que l’inactivation est aléatoire : l’X inactivé est tantôt l’X paternel, tantôt l’X maternel. Statistiquement, on aurait pu s’attendre à une répartition 50/50. Il n’en est rien. Dans des conditions normales, pour un tissu donné, l’inactivation peut varier de 20 à 80 % pour le chromosome d’origine paternelle ou maternelle. Une fois ce pourcentage établi, il est définitif. Un exemple très séduisant est celui des chattes tricolores, qui sont à la fois rousses, noires et blanches parce que les gènes des couleurs du pelage (O/o) sont portés par l’X chez ce félin, dont la fourrure n’est jamais la même d’une chatte à l’autre (Figure 3). Chez les femmes, cette variation peut entraîner des manifestations cliniques de maladies liées à l’X qui seront d’autant plus sévères que l’X porteur du gène anormal est activé: par exemple une femme vectrice peut présenter des signes de myopathie quand l’X porteur du gène DMD muté est majoritairement actif dans ses cellules musculaires.  | Figure 3.
Lola, une chatte à robe calico. L’aspect tigré de type «écaille de tortue» est dû à l’allèle sauvage A. Il est modifié par un gène épistasique (avec les allèles O et o) qui est porté par le chromosome X. Du fait de l’inactivation au hasard des X, la fourrure qui en résulte est unique. |
Mais le laxisme du chromosome X va plus loin. Après avoir cru que la totalité des gènes portés par l’X inactif (sauf ceux de la région PAR) était réduite au silence, on s’est aperçu que bon nombre de gènes continuaient à s’exprimer. Dans le même numéro de Nature, l’étude sur le profil d’expression de l’X inactivé révèle l’étendue et la variabilité de ces exceptions [4]. Sur un système introduisant uniquement l’X humain inactif dans des cellules de souris, Laura Carrell et Huntington Willard ont analysé 95 % des gènes portés par l’X. Si 75% sont réduits au silence de façon permanente, 15 % échappent toujours à l’inactivation. Ces gènes ne sont pas disséminés sur le chromosome X, mais se trouvent surtout à la partie distale du bras court, dans une région correspondant à la cinquième strate, la plus récente. La propension des gènes à échapper à l’inactivation semble diminuer avec l’ancienneté des strates, et la concentration en L1 est inversement corrélée à la proportion de gènes échappant à l’inactivation. Quant aux 10 % de gènes restant, leur profil d’inactivation est variable. Cette variabilité ne dépend pas de la culture : un même X donne toujours les mêmes réponses, mais les 40 chromosomes X analysés révèlent une grande hétérogénéité. Il existe donc chez les femmes une variabilité dans l’expression des gènes sur l’X inactif qui n’avait encore jamais été soupçonnée à ce point. L’existence de double dose pour certains gènes pose le problème d’une éventuelle maladie associée. Enfin, pour compliquer le tout, parmi les gènes échappant à l’inactivation, en particulier ceux qui appartiennent à la région PAR1 et qui sont donc censés toujours s’exprimer, il en est qui ne s’expriment pas complètement, certains n’atteignant même pas 25 % de l’expression complète. |
Le séquençage et l’analyse du profil d’inactivation du chromosome X pose plus de questions qu’il n’en résout. La concentration de certains gènes favorables en hémizygotie démontre la sollicitude de l’X vis-à-vis de la masculinité et de son compagnon, le chromosome Y ; sollicitude aussi dans l’extinction transcriptionnelle visant à obtenir une équivalence homme/femme dans le dosage des gènes portés par l’X. Mais avec tant d’insubordination, tant d’exceptions que le profil d’expression de chaque cellule XX en devient imprévisible. Nous le savions déjà : la femme… ni tout à fait la même, ni tout à fait une autre… |
1. Ross MT, Grafham DV, Coffey AJ, et al. The DNA sequence of the X human chromosome Nature 2005; 434 : 325–37. 2. Ropers HH, Hamel BC. X-linked mental retardation. Nat Rev Genet 2005; 6 : 46–57. 3. Skaletsky H, Kuroda-Kawaguchi T, Minx P, et al. The male-specific region of the human Y chromosome is a mosaic of discrete sequence classes. Nature 2003; 423 : 825–37. 4. Carrel L, Willard HF. X-inactivation profile reveals extensive variability in X linked gene expression in females. Nature 2005; 434 : 400–4. 5. Vallender EJ, Pearson NM, Lahn BT. The X chromosome : not just her brother’s keeper. Nat Genet 2005; 37 : 343–5. 6. Gunter C. She moves in mysterious ways. Nature 2005; 434 : 279–80. |