| |
| Med Sci (Paris). 2005 November; 21(11): 927–933. Published online 2005 November 15. doi: 10.1051/medsci/20052111927.Maladies multifactorielles : un cauchemar pour le généticien Josué Feingold* Inserm U.393, Hôpital Necker Enfants-Malades, 149, rue de Sèvres, 75015 Paris, France |
Neel écrivait en 1965 que le diabète sucré, maladie multifactorielle «modèle», était un cauchemar pour le généticien [
1]. Cela reste vrai quarante ans après pour l’ensemble des maladies multifactorielles, malgré l’accroissement considérable des connaissances en génétique. Dans cet article, nous tenterons d’analyser pourquoi l’étude génétique des maladies multifactorielles demeure si difficile. Auparavant, nous situerons ce groupe de maladies par rapport aux autres maladies génétiques, et ferons un rappel concernant la méthodologie d’étude de ces affections. Le lecteur peut également se reporter à la très bonne revue générale critique des maladies à hérédité complexe ou maladies multifactorielles de Campion, publiée en 2001 dans médecine/sciences [
2]. |
Classification des maladies génétiques On considère actuellement, peut-être de façon trop schématique, qu’il existe quatre grands groupes de maladies génétiques : les maladies héréditaires, à transmission mendélienne, les maladies mitochondriales, dont l’hérédité est maternelle, les maladies par aberration chromosomique et, enfin, les maladies multifactorielles, dont la répartition chez les apparentés ne suit pas les lois de Mendel. Le progrès des connaissances a été considérable dans les trois premiers groupes. En revanche, et malgré de très nombreux travaux réalisés par des laboratoires de qualité, peu de gènes impliqués dans les maladies multifactorielles ont été identifiés, contrairement au grand nombre de gènes identifiés dans les maladies héréditaires. Les maladies multifactorielles, encore appelées maladies polygéniques ou à hérédité complexe, sont dues à de nombreux facteurs génétiques et de milieu. Fait important, les allèles impliqués ne sont, dans la très grande majorité des cas, pas délétères, mais confèrent seulement une susceptibilité accrue à la maladie. Ce mode héréditaire concerne une grande partie des maladies communes telles que les diabètes de type 1 et 2, les maladies cardiovasculaires, certains cancers fréquents, des maladies neurologiques et psychiatriques, des malformations congénitales, l’asthme et les maladies auto-immunes. Leur étude comporte trois étapes : montrer que la maladie est familiale, montrer que cette tendance familiale est due à des facteurs génétiques et, enfin, identifier les gènes impliqués. |
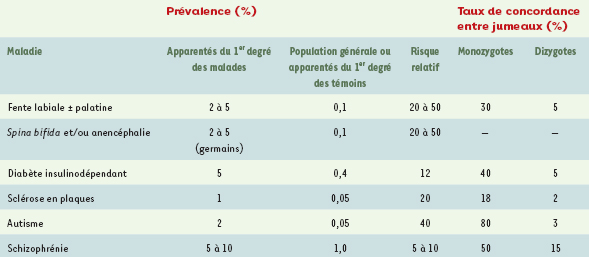
Pour démontrer qu’une maladie est familiale, on peut par exemple montrer que chez les apparentés du premier degré des malades (parents, frères, sœurs ou enfants), la maladie est plus fréquente que dans la population générale ou que chez les apparentés du premier degré de témoins sains (Tableau I ; des observations analogues sont rapportées pour de nombreux cancers et maladies cardiovasculaires). Pour faire ces comparaisons, il est nécessaire de tenir compte de l’âge, du sexe, et même de l’ethnie des apparentés des sujets malades ou témoins. Si la fréquence de la maladie est plus faible chez les apparentés du deuxième degré (oncles, tantes ou grands-parents), voire du troisième degré (cousins germains), elle est toujours supérieure à celle observée dans la population générale.  | TableauI.
Prévalence (%) de quelques maladies multifactorielles chez les apparentés du premier degré des malades et des témoins, et dans la population générale. Taux de concordance (%) chez les jumeaux. Le risque relatif (RR) est le rapport entre la prévalence de la maladie chez les apparentés et celle de la population générale (ou chez les apparentés des témoins) ; il se calcule en tenant compte du lien de parenté. Pour certains auteurs, un RR élevé serait en faveur d’une composante génétique importante ; en fait, il dépend en grande partie de la prévalence de la maladie, d’autant plus élevé que celle-ci est faible. Le RR permet de chiffrer le risque de récurrence de la maladie chez un apparenté du malade. Le risque relatif «familial» est appelé λr (relatives) ou λs (sibs) par certains auteurs. |
Démontrer un excès de cas familiaux dans une maladie ne fait toutefois que suggérer l’intérêt de la recherche d’une composante génétique: cette concentration peut en effet être due au milieu familial commun, ce qui constitue une des grandes difficultés de la génétique humaine, un milieu familial (hérédité socioculturelle) pouvant simuler une hérédité biologique. Ce type d’étude peut cependant prendre en compte un facteur environnemental dont on connaît le rôle étiologique: dans les familles de sujets atteints du cancer du poumon lié au tabac, par exemple, on a montré qu’il existait chez les apparentés un excès de sujets atteints de ce cancer, même parmi les non-fumeurs. Cette étude montre qu’il existe à côté du tabac un autre facteur étiologique, vraisemblablement génétique. |
Recherche d’une composante génétique Deux types d’études, de réalisation plus ou moins difficile, permettent de mettre en évidence l’existence d’une composante génétique. Étude des jumeaux C’est la plus ancienne des méthodes utilisées pour démontrer qu’une composante génétique est à l’origine de certaines maladies. Elle consiste à comparer le taux de concordance d’une maladie chez les jumeaux monozygotes (MZ) ou dizygotes (DZ) (Tableau I). Les jumeaux MZ, issus du même œuf, ont le même patrimoine génétique: toute discordance (un jumeau malade, l’autre sain) est par conséquent d’origine environnementale. Les jumeaux DZ, issus de deux œufs différents, ont la moitié de leur patrimoine génétique en commun: les discordances sont donc d’origine génétique ou environnementale. Si l’on suppose que les différences environnementales concernant les deux types de jumeaux sont les mêmes (ce qui n’est d’ailleurs pas toujours vrai), les différences de taux de concordance observées entre les jumeaux MZ et DZ ont une origine génétique. On comparera le plus souvent des jumeaux MZ à des jumeaux DZ de même sexe, car de nombreuses maladies sont plus fréquentes dans l’un des deux sexes. Enfin, il faut noter que des mutations somatiques peuvent rendre des jumeaux monozygotes discordants. Étude des enfants adoptés En séparant l’enfant de ses parents biologiques, l’adoption dissocie la composante génétique de la composante environnementale familiale postnatale. Un des types d’enquête consiste à comparer la fréquence de la maladie chez les parents biologiques et les parents adoptant, selon que l’enfant adopté est malade ou non. Ces enquêtes, difficiles à mener, ont essentiellement concerné les maladies psychiatriques. Une étude a ainsi montré qu’environ 20 % des parents biologiques d’un sujet atteint de maladie maniacodépressive étaient également malades, contre 2 % des parents biologiques de sujets adoptés sains; en revanche, la fréquence de la maladie était la même chez les parents ayant adopté des sujets atteints ou non. |
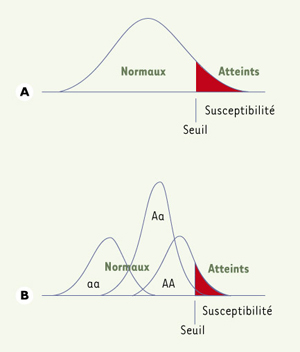
Modèles génétiques, analyse de ségrégationet identification des gènes Modèles génétiques Les deux modèles les plus classique sont le modèle polygénique ou multifactoriel et le modèle mixte. Dans le premier, on suppose que la susceptibilité à la maladie est sous la dépendance de nombreux gènes (hérédité polygénique) et de facteurs de milieu, dont l’effet individuel est petit : aucun des gènes impliqués n’est obligatoire. Il en résulte une distribution gaussienne (courbe en forme de cloche) de la susceptibilité en population générale (Figure 1A). Dès que la susceptibilité dépasse un certain seuil chez un individu, la maladie apparaît. Il s’agit donc d’une hérédité polygénique à seuil, qui intègre les facteurs de milieu dans l’étiologie de la maladie: facteurs de milieur «généraux», qui touchent les individus d’une façon aléatoire, ou «familiaux», qui accentuent la ressemblance entre individus apparentés. Ce modèle permet d’expliquer pourquoi on trouve une proportion plus grande de sujets malades parmi les sujets apparentés à l’individu atteint: ces individus ont une susceptibilité moyenne supérieure à celle de la population générale, d’où un décalage, vers la droite, de la distribution de leur susceptibilité. 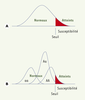 | Figure 1.
Modèles génétiques. A.
Modèle polygénique à seuil. B.
Modèle mixte (commentaires dans le texte). |
Dans le second modèle génétique, dit «mixte» (Figure 1B), la susceptibilité est sous le contrôle d’un gène majeur, dont l’action est toutefois modulée par un système polygénique et des facteurs de milieu ; à la limite, la composante polygénique peut être absente. Analyse de ségrégation Elle est le principal outil statistique pour analyser le mode héréditaire d’un trait pathologique ou non. Dans le cas des maladies multifactorielles, elle permet de montrer si la répartition familiale de la maladie est compatible avec la ségrégation d’un gène majeur, d’estimer ses caractéristiques (mode de transmission, pénétrance des différents génotypes, fréquences alléliques), de montrer la présence ou non d’une composante polygénique et d’estimer la fréquence des cas sporadiques. Si différentes méthodes d’analyse ont été décrites, le modèle régressif proposé par Bonney [
3,
4] semble le mieux adapté à l’étude des maladies multifactorielles: il est fondé sur une relation de régression entre le phénotype observé et des variables explicatives, comme l’effet d’un gène majeur ou les corrélations familiales résiduelles liées à des facteurs génétiques ou environnementaux. L’utilisation de l’analyse de ségrégation n’a pas donné tous les résultats qu’on pouvait espérer, notamment en raison d’une simplification obligatoire des modèles testés par rapport à la réalité biologique. Cependant, certains résultats d’un grand intérêt ont été obtenus : l’étude des répartitions familiales et l’analyse de ségrégation ont permis de montrer qu’une petite fraction de certaines maladies multifactorielles (cancers du sein ou du colon, diabètes de type 2, démence de type Alzheimer) avait un mode héréditaire monofactoriel. L’analyse de ségrégation a également montré que la susceptibilité à certaines maladies infectieuses dépendait en partie d’un gène majeur : c’est notamment le cas de la susceptibilité à l’infection par l’herpès virus 8 [
5]. Localisation chromosomique et identification des gènes de susceptibilité Ces travaux se sont révélés difficiles. Dans la majorité des études, on vise à localiser les gènes de susceptibilité sans faire au préalable d’analyse de ségrégation. Par leur effet, ces gènes sont comparables à ceux qui sont associés aux caractères quantitatifs : il s’agit donc de QTL ( quantitative trait loci). Si une analyse de ségrégation a toutefois montré qu’un gène majeur était impliqué, on peut essayer de le localiser par la méthode classique des lod-scores, largement utilisée dans l’étude des maladies monofactorielles. Dans ces cas, un «tour du génome» est réalisé en utilisant des marqueurs anonymes de l’ADN, microsatellites et SNP (single nucleotide polymorphims), ces derniers étant extrêmement fréquents et de plus en plus utilisés. Cependant, le mode héréditaire de la maladie est souvent trop imprécis pour utiliser la méthode des lod-scores (toute erreur sur le mode de transmission entraîne en effet une perte de puissance du test [
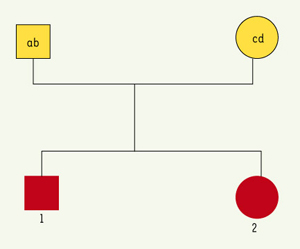
6]). Les méthodes alors utilisées sont dites non paramétriques, car elles ne font aucune hypothèse a priori sur le mode héréditaire de la maladie. Deux méthodes, complémentaires, sont très utilisées (pour revue, voir [2]): la méthode des paires de germains atteints (analyse de liaison entre la maladie et un marqueur) a pour but de détecter la présence d’un facteur de susceptibilité dans la région du marqueur ; la méthode de comparaison des fréquences alléliques, génotypiques ou phénotypiques entre des cas et des contrôles (étude d’association entre la maladie et un marqueur) vise, quant à elle, à mettre en évidence une différence dans les risques associés aux allèles, génotypes ou phénotypes du marqueur ; le risque peut être augmenté ou diminué (facteur de susceptibilité ou de résistance à la maladie). Dans ces méthodes non paramétriques, on peut soit étudier des gènes candidats qui, par leur fonction, peuvent être impliqués dans la maladie, soit faire «un tour du génome» en utilisant des marqueurs anonymes. L’étude de régions candidates suggérées par les modèles animaux peut également se révéler utile. Méthode «paire de germains atteints» (affected sib pair analysis) La méthode consiste à comparer, chez des paires de germains atteints de la même maladie, le nombre d’allèles du marqueur génétique transmis en même temps par le père et la mère. Si le marqueur étudié est proche, voire dans un gène de susceptibilité impliqué dans la maladie, les paires de germains malades présenteront un excès d’allèles du marqueur en commun : cet excès s’explique par la liaison ( linkage) existant entre le locus marqueur et le locus impliqué dans la maladie. En l’absence de liaison, les probabilités que les deux germains atteints partagent 0, 1 ou 2 allèles identiques parentaux (IBD, identity by descent) sont respectivement de 1/4, 1/2, 1/4. Ces proportions sont faciles à estimer si les quatre allèles parentaux sont différents (Figure 2). 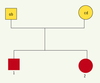 | Figure 2.
Méthode des paires de germains atteints. Le génotype des parents pour le marqueur est ab ou cd. Dans l’hypothèse d’une indépendance entre la maladie et le marqueur, chacun des germains atteints 1 et 2 peut présenter pour ce marqueur le génotype ac, ad, bc et bd avec la probabilité 1/4; dans un quart des cas, les deux germains auront les deux allèles en commun (IBD: identity by descent=2), dans la moitié un seul (IBD=1) et, dans un quart des cas, aucun allèle en commun (IBD=0). |
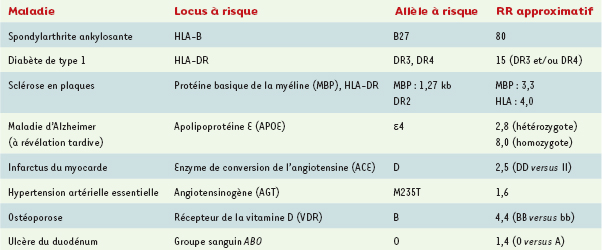
C’est ainsi que l’étude du système HLA dans la sclérose en plaques a montré la répartition suivante des paires de germains atteints : 40 % ont deux allèles identiques, 43 % un allèle identique et 17 % aucun allèle partagé, ce qui démontre une liaison entre cette maladie et le système HLA. Des méthodes prenant en compte l’ensemble des malades d’une famille ont été décrites. Association entre marqueurs génétiques et maladies Cette méthode compare la fréquence (phénotypique ou génique) des allèles d’un marqueur génétique chez des malades et des témoins soigneusement appariés, notamment sur l’origine géographique, l’âge ou le sexe. L’excès ou le déficit de fréquence d’un allèle chez les malades signe une association ; la confirmation par d’autres études permet de conclure à l’identification d’un allèle de susceptibilité ou de résistance à la maladie étudiée, en tenant compte du nombre de comparaisons effectuées dans l’analyse statistique des résultats. En ce qui concerne le marqueur identifié, il est soit directement impliqué dans la maladie (il s’agit alors d’un variant fonctionnel), soit, au contraire, en déséquilibre gamétique (déséquilibre de liaison) avec le gène. La création d’un témoin fictif interne, constitué par les allèles non transmis par les parents du sujet atteint, permet d’éviter les biais dus au groupe témoin : elle oblige cependant à étudier le sujet atteint et ses deux parents. Le test de déséquilibre de transmission (TDT, transmission desequilibrium test), proche du précédent et très utilisé, nécessite également l’étude du sujet atteint et de ses deux parents. Il compare les fréquences avec lesquelles un allèle particulier est transmis par un parent hétérozygote à l’enfant atteint: si l’allèle est associé à la maladie, il est transmis dans plus de la moitié des cas, et l’on peut conclure à l’association. Ce test étudie en même temps l’association et la liaison génétique ( linkage). Des exemples de gènes de susceptibilité sont présentés dans le Tableau II.  | Tableau II.
Exemples de gènes de susceptibilité à différentes maladies mutifactorielles.
|
Quelle que soit la stratégie d’identification des gènes impliqués dans les maladies multifactorielles, elle repose sur l’étude de gènes candidats, sélectionnés en raison de leur fonction, potentiellement impliquée dans la maladie, ou sur la base de résultats obtenus dans des modèles animaux. On étudie parfois aussi un caractère situé en amont de la maladie (phénotype intermédiaire), pour rendre l’analyse plus simple (cas de l’hypercholestérolémie étudiée pour rechercher les gènes impliqués dans l’infarctus du myocarde). En revanche, il est extrêmement difficile d’identifier les gènes de susceptibilité dans les régions chromosomiques repérées par analyse de liaison: ces régions sont effectivement relativement étendues, les allèles de susceptibilité ne sont pas des allèles délétères et, enfin, de nombreux résultats ne sont pas confirmés [
7]. L’identification par clonage positionnel est, quant à elle, très rare; on peut toutefois citer celle du gène CARD15 (NOD2) dans la maladie de Crohn [
8] et celle de la région régulatrice des gènes PARK2 et PACRG dans la susceptibilité à la lèpre [
9]. Caractéristiques des gènes de suceptibilité Si les études d’identification des gènes ont parfois permis de révéler des formes mendéliennes de maladies pourtant multifactorielles dans leur très grande majorité, elles ont également permis de mettre en évidence un certain nombre de caractéristiques des gènes de susceptibilité. Ainsi, les allèles impliqués dans la susceptibilité à une maladie ne sont pas des mutations délétères, mais appartiennent à des systèmes polymorphes; il existe cependant des situations intermédiaires entre mutations délétères et polymorphismes, c’est-à-dire entre hérédité mendélienne et hérédité multifactorielle. Il faut également reconnaître qu’il est parfois difficile de différencier une maladie mendélienne à pénétrance faible, pouvant être modulée par un système polygénique, d’une maladie multifactorielle oligogénique: la maladie de Hirschprung est un exemple [
10,
11]. Les allèles de susceptibilité à une maladie ont également pour caractéristique d’être parfois des allèles de résistance pour une autre pathologie. Enfin, ces allèles sont rarement ceux des gènes responsables des formes mendéliennes de la maladie considérée. En tout état de cause, il est notable que le nombre de gènes de susceptibilité identifiés est relativement faible, malgré la réalisation de très nombreuses études. |
Les difficultés de l’étude de la composante génétique des maladies multifactorielles De récents articles ont souligné que les résultats de très nombreuses études de liaison [7] ou d’association [
12] n’ont pu être confirmés. Les causes de ces échecs ont récemment été analysées, et des pistes de recherches pour tenter de sortir de cette situation ont été préconisées [
13]. Les diverses causes de cet échec, qui reste tout de même partiel, sont dominées par l’hétérogénéité génétique des maladies multifactorielles [13,
14]. Problèmes d’ordre statistique et épidémiologique Chaque analyse statistique d’une étude nécessite de nombreuses comparaisons, ce qui pose le problème du seuil de signification. L’utilisation de la correction de Bonferroni a pour conséquence d’entraîner une augmentation, parfois importante, de résultats faussement négatifs. La faiblesse de l’effet de chaque gène de susceptibilité rend difficile sa mise en évidence avec un seuil de signification acceptable, c’est-à-dire nettement inférieur au «0,05 classique». Pour remédier à cette difficulté, différentes stratégies sont utilisées, notamment celle de n’étudier que la moitié de l’échantillon, la seconde servant à confirmer ou infirmer les résultats significatifs, le seuil de signification de la première analyse pouvant être élevé (≥0,10). La méta-analyse de l’ensemble des résultats publiés est également de plus en plus utilisée. D’un point de vue épidémiologique, les échantillons de malades étudiés ne sont pas toujours comparables sur le plan de la gravité de la maladie, ou de son stade évolutif, ce qui peut entraîner des résultats contradictoires : ainsi, un premier échantillon ne comportant que des formes graves servira à rechercher des facteurs génétiques étiologiques et pronostiques considérés comme étiologiques; un second échantillon, rassemblant pour sa part différentes formes de la maladie, servira uniquement à la recherche de facteurs étiologiques. La comparaison des résultats de ces études risque de révéler des discordances. Hétérogénéité génétique des maladies multifactorielles D’abondants travaux ont montré que l’hétérogénéité génétique, allélique ou génique des maladies monogéniques était très importante. Il est tout à fait vraisemblable qu’il en est de même dans les maladies multifactorielles : pour cette raison, il n’est pas fréquent de retrouver dans de nouvelles études des résultats déjà publiés, qu’il s’agisse d’études de liaison ou d’association [ 7, 12]. L’hétérogénéité allélique peut rendre difficile l’interprétation des études d’association. Des allèles différents d’un même gène peuvent être associés à la susceptibilité à une maladie. De plus, la fréquence de ces allèles peut être très différente d’une population à l’autre. Enfin, ces marqueurs génétiques peuvent ne pas être des allèles de susceptibilité, mais être ou ne pas être en déséquilibre de liaison avec le ou les variants fonctionnels selon la population, d’où la possibilité de résultats contradictoires. L’hétérogénéité génique est vraisemblablement très importante. À titre d’exemple, on a longtemps pensé que les diabètes insulinodépendant et non insulinodépendant étaient deux formes d’une même maladie multifactorielle ; ce sont les études génétiques qui ont montré qu’il s’agissait de deux maladies qui n’avaient pas la même composante génétique. Prenons un modèle théorique simple: une maladie multifactorielle peut être en rapport avec des allèles de susceptibilité des gènes G1, G2, G3 chez certains malades, et avec des allèles des gènes G4, G5, G6, G7 chez d’autres. Une combinaison des deux groupes de gènes de susceptibilité ne peut être associée à la maladie, ce qui entraîne une difficulté à trouver des résultats concordants : au phénotype qui définit la maladie correspond deux maladies différentes, voire plus. Si l’hétérogénéité génétique est totale (ou quasi totale) pour les diabètes, une hétérogénéité génétique, certes partielle, commence à être connue pour certaines maladies : le gène NOD15, par exemple, est associé à la maladie de Crohn en Europe, alors qu’il ne l’est pas au Japon [
15]. On peut conclure que l’identification des gènes de susceptibilité nécessitera une étude fine du phénotype «maladie» pour tenter de différencier ses différentes formes. Si cela se révèle insuffisant, la description des différentes maladies devra prendre en compte les gènes associés à la maladie, même si les résultats ne sont pas concordants. Pour diminuer les effets de l’hétérogénéité génétique, certains auteurs étudient des populations isolées, ou qui l’étaient à l’origine (études réalisées en Islande ou en Finlande, notamment). À ce jour, l’étude de ce type de population n’a pas permis d’identifier beaucoup de gènes de maladies multifactorielles. D’une façon apparemment «contradictoire», des auteurs préconisent d’ailleurs plutôt l’étude de populations mélangées [
16]. À l’origine des maladies multifactorielles, un génotype favorable devenu défavorable par le progrès En 1962, J.Neel [
17] a émis l’hypothèse que certains génotypes favorables à l’espèce humaine durant des millénaires seraient devenus défavorables du fait du changement de milieu dans lequel vit l’homme, du moins dans les pays industrialisés. Ainsi, l’augmentation de la fréquence du diabète non insulinodépendant serait dû au fait qu’un génotype ayant permis une utilisation optimale des aliments pendant les périodes de pénurie alimentaire et de famine (génotype «d’épargne»), ce qui a été le cas pendant des millénaires, est devenu défavorable dans les populations à régime alimentaire abondant ; des considérations analogues peuvent s’appliquer à l’obésité. Quant à l’hypertension artérielle, le changement d’environnement concerne le chlorure de sodium : de fait, le régime alimentaire de l’homme fut très pauvre en sel pendant une très longue période de son histoire, d’où la sélection des gènes impliqués dans sa rétention ; les régimes actuels, très riches en sel, ne sont pas adaptés à notre constitution génétique. Pour l’asthme et les maladies auto-immunes, c’est la quasi disparition des maladies infectieuses qui pourrait expliquer l’augmentation de leurs fréquences : le système immunitaire ne serait plus adapté, en partie, aux conditions de vie actuelle, au moins en ce qui concerne ces maladies. La conséquence de tout cela est que les allèles impliqués dans ces maladies sont fréquents, voire très fréquents. On attend chez les malades des fréquences encore plus élevées de ces allèles, ce qui n’est toutefois pas facile à mettre en évidence : on peut néanmoins citer le variant M235T du gène de l’angiotensinogène, qui a une fréquence de 0,76 (chez les Japonais) et 0,91 chez les sujets hypertendus, et le variant «proline» en position 12 du gène PPARγ, dont la fréquence dans la population est de 0,85 et qui augmente légèrement le risque de diabète non insulinodépendant [
18,
19]. L’hypothèse de Neel peut s’appliquer, comme on l’a vu, à de nombreuses maladies communes. Elle montre que les génotypes associés à ces pathologies sont très «sensibles» au milieu. Celui-ci, «favorable» ou «défavorable» selon les périodes, peut être différent d’une population à l’autre, d’autant que ces populations peuvent aussi avoir des histoires génétiques différentes (dérive génétique, migration) : une des conséquences possibles de cela est que les gènes de susceptibilité peuvent différer d’une population à l’autre, entraînant des phénomènes d’hétérogénéité génétique au sein d’une même maladie. |
Malgré quelques succès, il faut reconnaître que le nombre de gènes de susceptibilité identifiés dans les maladies multifactorielles est relativement petit par rapport au nombre de recherches réalisées. Il faut en outre rappeler que la valeur prédictive positive des allèles identifiés est faible, ne permettant pas de dépister des populations à risque [
20]. Cependant, chez le sujet malade, quelques rares marqueurs génétiques peuvent être utilisés comme critère diagnostique : un des meilleurs exemples est celui du HLA-B27 dans la spondylarthrite ankylosante. De plus, la recherche des gènes de susceptibilités doit être poursuivie, avec de nouvelles approches qu’il faudra imaginer, car leur identification devrait permettre également de mieux connaître la physiopathologie de ces maladies, avec à la clé le développement de traitements plus efficaces comportant, peut-être, moins d’effets secondaires. |
1. Neel JV, Fajan SS, Conn JW, Davidson RT. Diabetes mellitus. In: Genetics and the epidemiology of chronic diseases. Amsterdam: Excerpta Medica, 1965: 105–32. 2. Campion D. Dissection génétique des maladies à hérédité complexe. Med Sci (Paris) 2001; 17 : 1139–48. 3. Bonney GE. Regressive logistic models for familial disease and other binary traits. Biometrics 1986; 42 : 611–25. 4. Demenais F. Regressive logistic models for familial diseases: a formulation assuming an underlying liability model. Am J Hum Genet 1991; 49 : 773–85. 5. Plancoulaine S, Gessain A, Van Beveren M, et al. Evidence for a recessive major gene predisposing to human Herpesvirus (HHV-8). Infection in a population in which HHV-8 is endemic. J Infect Dis 2003; 187 : 1944–50. 6. Clerget-Darpoux F, Bonaiti-Pellie C, Hochez J. Effects of misspecifying genetic parameters in lod score analysis. Biometrics 1986; 42 : 393–9. 7. Altmuller J, Palmer LJ, Fischer G, et al. Genome wide scans of complex human diseases: true linkage is hard to find. Am J Hum Genet 2001; 69 : 936–50 8. Hugot JP, Chamaillard M, Souali H, et al. Association of NOD2 leucine-rich repeat variants with susceptibilitity to Crohn’s diseases. Nature 2001; 411 : 599–603. 9. Alcais A, Abel L. Identification d’un nouveau gène de susceptibilité à la lèpre par clonage positionnel. Med Sci (Paris) 2004; 20 : 729–32. 10. Amiel J, Lyonnet S. Hirschsprung’s disease, associated syndromes and genetics: a review. J Med Genet 2001; 38 : 729–39. 11. Pelet A, De Pontual L, Clement-Ziza M, et al. Homozygosity for a frequent and weakly penetrant predisposing allele at the RET locus in sporadic Hirschsprung.J Med Genet 2005; 42 : e18. 12. Hirschhorn JN, Lohmuller K, Byrne E, Hirschhorn K. A comprehensive review of genetic association studies. Genet Med 2002; 4 : 45–61. 13. Thorton-Wells TA, Moore JH, Haines JL. Genetics, statistics and human disease: analytical relooking for complexity. Trends Genet 2004; 20 : 640–7. 14. Strachan T, Redad AP. Mapping and identifying genes conferring susceptibility to complex diseases. In: Human molecular genetics, 3rded. London: Garland Publishing, 2004; 15: 435–60. 15. Yamasaki K, Takazoe M, Tanaka T, et al. Absence of mutation in the NOD2/CARD15 gene among 483 japonese patients with Crohn’s disease. J Hum Genet 2002; 47 : 469–72. 16. Zhu X, Luke A, Cooper RS, et al. Admixture mapping for hypertension loci with genome-scan markers. Nat Genet 2005; 37 : 177–86. 17. Neel JV. Diabetes mellitus: a «Thifty» genotype rendered detrimental by «progress». Am J Hum Genet 1962; 14 : 353–62. 18. Jeunemaitre X, Inoue I, Williams C, et al. Haplotype of angiotensinogen in essential hypertension. Am J Med Genet 1997; 60 : 1448–60. 19. Altshuler D, Hirschhorn JN, Klannemark M, et al. The common PPAR gamma Pro12 Ala polymorphism is associated with decreased risk of type 2 diabetes. Nat Genet 2000; 26 : 76–80. 20. Feingold J. La génétique prédictive en médecine. Gynecol Obstet 2000; 28 : 391–5. |