| |
| Med Sci (Paris). 2006 November; 22(11): 919–921. Published online 2006 November 15. doi: 10.1051/medsci/20062211919.La mystification et l’ingéniosité de suppresseurs de tumeurs dans les fonctions nucléaires Christelle Cayrou, Yannick Doyon, Anne-Julie Landry, Valérie Côté, and Jacques Côté* Centre de recherche en cancérologie de l’Université Laval, Hôtel-Dieu de Québec (CHUQ), rue McMahon, Québec (Québec) G1R 2J6, Canada MeSH keywords: Noyau de la cellule, ADN, Gènes suppresseurs de tumeur, Protéines à homéodomaine, Humains, Tumeurs, Récepteurs cytoplasmiques et nucléaires, Protéines gène suppresseur tumeur |
La dynamique de la structure chromatinienne est régulée par un jeu de modifications épigénétiques [
1] qui vont dicter le devenir de nombreux processus nucléaires comme la transcription, la réparation d’ADN, la recombinaison et la réplication. Les connaissances actuelles sur les éléments auxquels attribuer ces modifications révèlent le rôle prépondérant de ces facteurs dans le déclenchement et la progression de nombreuses maladies chez l’homme, en particulier certaines formes de cancer. [
2]. Par exemple, la méthylation et la désacétylation de résidus spécifiques des histones sont parmi les mécanismes pouvant aboutir à la répression d’un gène suppresseur de tumeurs [
3]. Les activités modifiant la chromatine fonctionnent sous forme de complexes multiprotéiques [2]. L’identification, la purification et la caractérisation biochimique de ces complexes sont ainsi au cœur de la compréhension moléculaire de l’expression de gènes aussi bien que du maintien et du transfert d’information génétique chez les eucaryotes. Tous les membres de la famille des suppresseurs de tumeurs ING (inhibitor of growth) sont des corégulateurs de la fonction transcriptionnelle de p53, jouant un rôle premier dans la régulation de la croissance et de la prolifération cellulaire, ainsi que dans l’apparition de cancers [
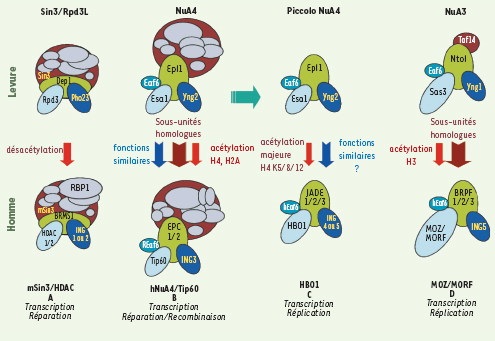
4]. Ces protéines sont très conservées de la levure à l’homme. Chez la levure, chacun des 3 homologues de cette famille se retrouve exclusivement à l’intérieur de complexes histone désacétylase (HDAC ; Pho23 dans Sin3/Rpd3L) et histone acétyltransférase (HAT ; Yng1 dans NuA3 et Yng2 dans NuA4) (Figure 1, partie supérieure) [
5,
6]. Chez l’homme, la famille compte cinq protéines, les ING de 1 à 5. ING1, étant plus apparenté à Pho23 de levure, est une sous-unité du complexe mSin3/HDAC tandis que ING3, similaire à Yng2, se retrouve dans le complexe HAT hNuA4/Tip60 (Figure 1,
partie inférieure, A et B) [
7,
8]. Ces deux associations chez l’homme suggèrent donc que les autres membres de la famille, soit ING2, ING4 et ING5, sont également associées à des complexes HAT/HDAC spécifiques, liant la régulation de la chromatine avec la fonction de p53 et la suppression de tumeurs. Pour tester cette hypothèse, nous avons effectué la caractérisation biochimique complète des complexes humains associés aux protéines ING [
9].  | Figure 1.
Conservation de la composition, de la spécificité enzymatique et des fonctions nucléaires des complexes ING de la levure à l’humain. Les complexes ING1/2 ont été impliqués dans la régulation transcriptionnelle et la réparation de l’ADN. Le complexe ING3 joue des rôles importants durant la transcription, la réparation et la recombinaison. Le complexe ING4 a été impliqué en transcription, de même pour les complexes ING5, sauf que ces derniers sont en plus essentiels pour la réplication de matériel génétique. Les complexes ING sont donc au cœur de l’expression et du maintien du génome des eucaryotes (tiré de [ 9] avec la permission de Elsevier). |
|
Structure et fonctions des complexes ING humains Nous avons d’abord purifié par affinité multiple le complexe associé à ING2. Comme ce dernier est très apparenté à ING1, il ne fut pas trop surprenant de le trouver également associé à un complexe mSin3/HDAC, mais indépendamment de ING1. Nous avons identifié dans ce complexe la protéine BRMS1, qui participe à la suppression des métastases, ainsi que RBP1, qui permet le recrutement du complexe mSin3/HDAC par les protéines de la famille Rb, induisant l’arrêt du cycle cellulaire en réprimant la transcription dépendante de E2F (désacétylation des gènes d’entrée en phase S) [9,
10]. Puisque RBP1 et BRMS1 sont partenaires dans un même complexe HDAC, cela suggère un nouveau mécanisme par lequel ING2 pourrait agir comme suppresseur de tumeurs et de métastases (indépendant de p53). Si ING3 est associée au complexe HAT hNuA4/Tip60 déjà connu (Figure 1, B), ING4, lui, fait partie d’un nouveau complexe HAT de quatre sous-unités (Figure 1C). Outre ING4, régulateur de la croissance et de l’angiogenèse de tumeurs au cerveau [
11], le complexe contient HBO1 (histone acetyltransferase binding to ORC1, précédemment suggérée comme jouant un rôle lors de l’initiation de la réplication, la formation de lymphomes, ainsi que dans la régulation de la transcription par le récepteur des androgènes [
12]) et les paralogues JADE, dont JADE1 qui est un suppresseur de tumeurs du rein [
13]. ING3 et ING4 sont donc associées à deux HAT faisant partie de la famille MYST, qui comprend un grand nombre d’acétyltransférases de structure apparentée et conservées au cours de l’évolution des cellules eucaryotes [12]. Tip60-ING3 (le complexe hNuA4) acétyle préférentiellement les histones H4 et H2A et HBO1-ING4 acétyle l’histone H4 (et H3 à un moindre degré) [9]. Nous avons démontré par interférence par l’ARN que HBO1 contribue à la majorité de l’acétylation de l’histone H4 chez l’homme tandis que Tip60 joue un rôle secondaire. Une différence importante de leurs effets sur la croissance cellulaire a également été constatée : la déplétion de Tip60 provoque un arrêt de croissance par un blocage en phase G2/M, tandis que HBO1 est requise pour le passage à travers les phases S et G2/M [9]. Cette dualité d’effet de HBO1 à deux endroits du cycle cellulaire est essentiellement due à ses associations indépendantes avec deux membres de la famille ING, formant ainsi deux complexes fonctionnellement distincts. En effet, la purification directe de HBO1 a montré son association additionnelle avec ING5 (Figure 1C). Ce complexe partage les trois autres sous-unités déjà identifiées dans le complexe HBO1-ING4 mais est physiquement distinct de ce dernier. Des études d’interférence par l’ARN ont démontré que HBO1-ING4 est important pour le passage à travers la phase G2/M ; pour sa part, HBO1-ING5 est essentiel pour la progression de la phase S [9]. La mesure de la synthèse d’ADN par marquage in vivo a confirmé l’intervention de HBO1, strictement dépendante d’ING5, dans la réplication et, plus précisément, dans la progression de la fourche réplicative. La présence des hélicases MCM dans la purification de ING5, mais non de ING4 soutient cette hypothèse [9]. Finalement, l’effet plus sévère de la déplétion d’ING5 sur la synthèse d’ADN par rapport à celui observé avec HBO1 s’explique par la présence d’un autre complexe HAT contenant ING5 (Figure 1D). En effet, ING5 se trouve dans deux complexes distincts, l’un ciblant l’histone H4 par HBO1 et l’autre ciblant H3 par MOZ/MORF, deux membres très similaires de la famille MYST [9, 12]. Il semble donc que les complexes HBO1-ING5 et MOZ/MORF-ING5 agissent de concert afin de permettre la réplication de l’ADN chez l’homme. |
Nous avons caractérisé les interactions moléculaires stables de chacun des suppresseurs de tumeurs de la famille ING, ce qui a fourni des perspectives mécanistiques sur leur fonction dans la régulation de la prolifération cellulaire. Ces cinq protéines constituent chacune une composante clé de complexes multiprotéiques HAT ou HDAC spécifiques. Elles sont également des régulateurs critiques de l’activité de ces complexes sur la chromatine et sont au cœur de la régulation des niveaux d’acétylation des histones in vivo. Par cette étude, nous avons pu identifier l’activité en charge de la majorité de l’acétylation de l’histone H4 chez l’humain. Nous avons démontré pour la première fois le rôle essentiel de complexes HAT dans la réplication de l’ADN durant toute la durée de la phase S, supposément à la fourche réplicative. Dans l’ensemble, les complexes ING identifiés attestent l’importance cruciale de ces suppresseurs de tumeurs dans l’expression et le maintien du génome, c’est-à-dire durant la transcription, la réparation, la recombinaison et la réplication de l’ADN. Ces fonctions enrôlent chaque complexe ING et leurs composants dans différentes formes de cancer, dans la naissance et la croissance de tumeurs ou la formation de métastases, renforçant ainsi l’idée qu’existe un lien incontournable entre la régulation épigénétique et le cancer.

|
1. Lacoste N, Côté J. The epigenetic code of histones. Med Sci (Paris) 2003; 19 : 955–9. 2. Lund AH, van Lohuizen M. Epigenetics and cancer. Genes Dev 2004; 18 : 2315–35. 3. Fraga MF, Esteller M. Towards the human cancer epigenome: a first draft of histone modifications. Cell Cycle 2005; 4 : 1377–81. 4. Campos EI, Chin MY, Kuo WH, Li G. Biological functions of the ING family tumor suppressors. Cell Mol Life Sci 2004; 61 : 2597–613. 5. Howe L, Kusch T, Muster N, et al. Yng1p modulates the activity of Sas3p as a component of the yeast NuA3 histone acetyltransferase complex. Mol Cell Biol 2002; 22 : 5047–53. 6. Nourani A, Doyon Y, Utley RT, et al. Role of an ING1 growth regulator in transcriptional activation and targeted histone acetylation by the NuA4 complex. Mol Cell Biol 2001; 21 : 7629–40. 7. Kuzmichev A, Zhang Y, Erdjument-Bromage H, et al. Role of the Sin3-histone deacetylase complex in growth regulation by the candidate tumor suppressor p33(ING1). Mol Cell Biol 2002; 22 : 835–48. 8. Doyon Y, Selleck W, Lane WS, et al. Structural and functional conservation of the NuA4 histone acetyltransferase complex from yeast to humans. Mol Cell Biol 2004; 24 : 1884–96 9. Doyon Y, Cayrou C, Ullah M, et al. ING tumor suppressor proteins are critical regulators of chromatin acetylation required for genome expression and perpetuation. Mol Cell 2006; 21 : 51–64. 10. Meehan WJ, Samant RS, Hopper JE, et al. Breast cancer metastasis suppressor 1 (BRMS1) forms complexes with retinoblastoma-binding protein 1 (RBP1) and the mSin3 histone deacetylase complex and represses transcription. J Biol Chem 2004; 279 : 1562–9. 11. Garkavtsev I, Kozin SV, Chernova O, et al. The candidate tumour suppressor protein ING4 regulates brain tumour growth and angiogenesis. Nature 2004; 428 : 328–32. 12. Utley RT, Côté J. The MYST family of histone acetyltransferases. Curr Top Microbiol Immunol 2003; 274 : 203–36. 13. Zhou MI, Foy RL, Chitalia VC, et al. Jade-1, a candidate renal tumour suppressor that promotes apoptosis. Proc Natl Acad Sci USA 2005; 102 : 11035–40. |