Les protéines du groupe Polycomb (PcG) sont des facteurs chromatiniens connus pour maintenir l’état transcriptionnel réprimé de leurs gènes cibles au cours du développement. Ces facteurs agissent au niveau de leurs gènes cibles sous forme de larges complexes multimériques appelés Polycomb Repressive Complex (PRC). Ces complexes agissent sur la transcription de leurs gènes cibles en se fixant sur l’ADN au niveau de séquences régulatrices appelées PcG response elements (PRE). La structure et la fonction des deux complexes identifiés à ce jour (PRC1 et PRC2) sont conservées depuis la drosophile jusqu’à l’Homme. Après recrutement sur un PRE par des facteurs de transcription précoces, le complexe PRC2 est capable d’ajouter trois groupes méthyl à la lysine 27 de l’histone H3 (H3K27me3) des nucléosomes. Cette marque épigénétique permet le recrutement du complexe PRC1 qui maintient la répression des gènes cibles associés au PRE soit par compaction de la chromatine, soit par interaction directe avec la machinerie transcriptionnelle au niveau du promoteur du gène cible [ 1].
Les gènes cibles les plus connus des protéines du PcG sont les gènes homéotiques. Mais seul un très faible pourcentage de gènes cibles avait été identifié à ce jour. Cependant, la caractérisation de certains de ces gènes cibles chez la drosophile — comme les gènes ph, en, hh et ci — suggère que les protéines du PcG régulent beaucoup de gènes impliqués dans le développement et l’embryogenèse. De plus, chez l’homme et la souris, plusieurs types de cancers ont été associés à des défauts d’expression des protéines du PcG, suggérant un rôle plus général dans l’identité cellulaire ainsi que dans la prolifération cellulaire [ 2]. Plusieurs autres fonctions ont également été associées aux protéines du PcG, notamment leur implication dans les cellules souches.
Les cellules souches se définissent par leur capacité à se diviser de manière extensive et à donner naissance à de multiples lignées cellulaires différenciées. Les cellules souches embryonnaires (ES) sont pluripotentes, c’est-à-dire capables d’engendrer un grand nombre de types cellulaires de l’embryon. On soupçonne depuis longtemps que la reprogrammation des cellules souches au cours de la différenciation doit impliquer une altération dynamique de l’état épigénétique de leur programme d’expression génique. Et les protéines du PcG seraient impliquées dans ce programme. En effet, chez l’homme, il est impossible d’établir des lignées de cellules ES dans un contexte mutant pour EZH2 (enhancer of Zeste 2), une protéine du PcG.
Pour comprendre de quelle manière les protéines du PcG maintiennent l’identité des cellules au cours du développement et de la différenciation, sept équipes ont publié très récemment de façon indépendante une cartographie à l’échelle génomique de la distribution des protéines du PcG. Ces travaux reposent en majorité sur l’utilisation de deux techniques. L’immunoprécipitation de chromatine (ChIP), qui permet de récupérer les séquences d’ADN associées à une protéine d’intérêt, et les puces à ADN génomiques, qui permettent désormais de couvrir le génome entier d’un organisme avec une résolution de l’ordre de 100 pb. Bien que les modèles biologiques utilisés varient (cellules souches humaines et murines, cellules de drosophile, cellules cancéreuses ou animaux entiers), il faut souligner la remarquable concordance des résultats obtenus dans ces articles.
Chez la drosophile, B. Tolhuis et al. [ 3] ont cartographié trois membres du PcG (PC, SCE et ESC) sur le chromosome 2L par DamID (DNA adenine methyltransferase identification), une technique alternative au ChIP. N. Nègre et al. [ 4] ont utilisé la technique de ChIP pour étudier les protéines PC et PH, sur de larges portions des chromosomes X et 2L à différents stades du développement de la drosophile. Puis, Y.B. Schwartz et al. [ 5] ont analysé par ChIP la localisation de trois protéines du PcG (PC, E(Z) et PSC) ainsi que la marque H3K27me3 sur l’ensemble du génome. Les résultats publiés dans ces trois articles démontrent que les protéines du PcG définissent de larges domaines de fixation le long des chromosomes pouvant aller de 10 kb à plus de 30 kb (Figure 1). Les profils décrits pour les différents membres du PcG sont fortement corrélés entre eux ainsi qu’avec la marque répressive H3K27me3 (méthylation de l’histone H3 sur la lysine 27). Ainsi, dans les cellules embryonnaires, 80 % des gènes associés aux domaines de fixation du PcG ne sont pas transcrits. Ces gènes fixés par les protéines du PcG sont majoritairement des facteurs de transcription impliqués dans les voies de régulation majeures contrôlant la différenciation, la prolifération cellulaire et la morphogenèse. Toutefois, d’autres catégories de gènes ont été identifiées, encodant des enzymes métaboliques ou des récepteurs stéroïdiens, la plupart du temps impliqués dans le développement. De manière remarquable, ces voies de régulation et leurs gènes associés sont conservés chez les mammifères et un quatrième article a montré qu’elles sont également régulées par les protéines du PcG chez l’homme [ 6].
| Figure 1.
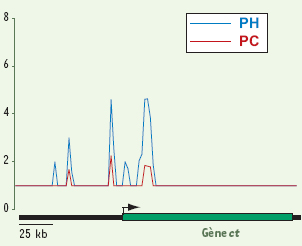
Exemple de cartographie des protéines PC et PH au niveau du gène cut (
ct
) chez la drosophile. Les courbes représentent l’intensité du signal détecté sur puces à ADN après expérience de ChIP et donc la fixation des protéines. Les signaux statistiquement non significatifs ont une valeur de 1. On peut voir sur cette région de 200 kb la fixation de PC et de PH dans des zones régulatrices aux alentours du promoteur du gène ct. |
Trois autres articles ont analysé le profil de fixation des protéines du PcG dans les cellules ES humaines et murines [ 7– 9] et aboutissent aux mêmes conclusions. Cependant, ils ont également pu montrer que les gènes cibles des protéines du PcG sont réprimés dans les cellules ES mais deviennent activés au cours de la différenciation. Cette activation des gènes cibles s’accompagne d’une perte de fixation des protéines du PcG et de la marque H3K27me3 ainsi que de la réoccupation de leur promoteur par l’ARN polymérase II (PolII) et de la marque épigénétique active H3K4me3. Cette observation de la dynamique du PcG au cours de la différenciation est à mettre en relation avec celle observée chez la drosophile [4] où certains gènes, comme le gène gt, requis pour la segmentation embryonnaire, sont fixés par les protéines du PcG dans l’embryon mais plus au stade pupal ou au stade adulte1. Cela pourrait indiquer que la répression des gènes par les protéines du PcG permettrait une certaine plasticité au cours du développement. Cette bivalence des régions régulatrices impliquant la marque répressive H3K27me3 et la marque active H3K4me3 a été mise en évidence dans les cellules ES par deux autres études génomiques [ 10, 11]. Dans ces articles, les auteurs montrent notamment que des domaines répressifs caractérisés par une forte densité de la marque répressive H3K27me3 contiennent de plus petits domaines contenant la marque active H3K4me3. Cette caractéristique chromatinienne permet d’établir un modèle de compétition où un jeu de gènes-clés du développement et de la différenciation serait maintenu réprimé par défaut par les protéines du PcG mais préparés à leur réactivation potentielle (Figure 2).
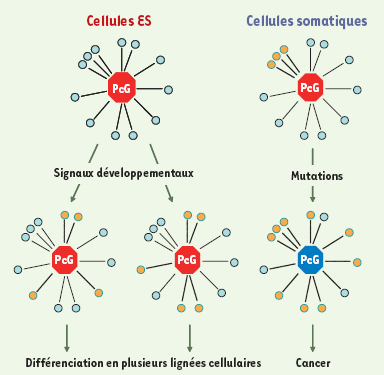
| Figure 2.
Modèle de maintien de l’identité cellulaire par les protéines du PcG. L’ensemble des gènes régulés par les protéines du PcG sont représentés par des cercles bleus. Les gènes des voies de signalisation appropriées s’activent (en orange) au cours de la différenciation cellulaire. Les autres gènes sont maintenus réprimés. Dans les cas de cancer associés aux protéines du PcG, le contrôle de ces voies de signalisation par les protéines du PcG devient erratique et conduit à une perte d’identité cellulaire. |
Ainsi, ces études génomiques de cartographie des protéines du PcG ont posé les fondations essentielles à la compréhension des mécanismes épigénétiques gouvernant la destinée cellulaire.