| |
| Med Sci (Paris). 2006 December; 22(12): 1053–1060. Published online 2006 December 15. doi: 10.1051/medsci/200622121053.Le riborégulateur adénine Un nouveau mode de régulation génétique Jean-François Lemay and Daniel A. Lafontaine* Département de Biologie, Faculté des sciences, Université de Sherbrooke, 2500, boulevard de l’Université, Sherbrooke (Québec), J1K 2R1 Canada |
Un contrôle est exercé chez les organismes à chaque étape de l’expression des gènes, que ce soit lors de la transcription génique, de la maturation des ARN messagers (ARNm), du transport des ARNm ou bien en régulant leur stabilité ou leur traduction par le ribosome. Cette multitude de mécanismes permet à la cellule de maintenir son homéostasie et de réagir aux divers facteurs environnementaux qui l’entourent. L’homéostasie de la cellule est principalement maintenue grâce à la détection des changements dans son environnement par des récepteurs protéiques et par les enzymes qui accomplissent leurs rôles à la suite de ces changements. Jusqu’à tout récemment, on pensait que la régulation de l’homéostasie cellulaire était essentiellement attribuable à l’action de facteurs protéiques. Cependant, la découverte de petits motifs d’ARN, appelés riborégulateurs, a changé cette perception. Aujourd’hui, il est clair que l’ARN est un rouage de la régulation génétique. Il exerce son action par le biais de certains métabolites essentiels propres aux procaryotes et les eucaryotes. |
Découverte des riborégulateurs Afin de comprendre comment les riborégulateurs ont été découverts, il est important de faire un bref survol historique des découvertes scientifiques qui ont pavé la voie au concept de la riborégulation telle qu’on la perçoit aujourd’hui. En effet, depuis le concept du dogme central émis par Francis Crick en 1958 [
1], l’ARN messager était considéré comme une simple navette transportant l’information génétique provenant de l’ADN d’une cellule jusqu’aux ribosomes, afin que ceux-ci décodent cette information pour la fabrication de protéines utiles au bon fonctionnement cellulaire. Au début des années 1980, la découverte des ribozymes a montré que l’ARN pouvait avoir une activité enzymatique [
2,
3], ce qui a grandement stimulé les recherches sur l’ARN au cours des années suivantes. Un des exemples les plus étonnants provient de l’analyse de la structure cristallographique du ribosome, qui suggère que la formation du lien peptidique serait catalysé par l’ARN ribosomique [
4]. Cette complexité structurale des acides nucléiques a été développée notamment grâce à certaines techniques d’évolution artificielle qui ont été mises au point afin de permettre leur évolution accélérée en laboratoire [
5]. Cela a notamment permis de produire une panoplie de ribozymes capables d’effectuer une diversité de réactions enzymatiques rivalisant avec les protéines [5]. De plus, les techniques d’évolution in vitro ont permis de produire des molécules d’acides nucléiques (aptamères) capables de lier de petites molécules manifestant une haute affinité et une grande spécificité [
6,
7]. Une étude en particulier a montré qu’il était possible d’implanter un aptamère dans la région 5’ non-traduite d’un gène rapporteur et de moduler son expression dans des cellules de mammifères à l’aide du colorant Hoechst H33258 [
8]. L’ensemble des travaux entrepris a fortement stimulé la communauté scientifique et certains groupes de chercheurs ont émis l’hypothèse selon laquelle l’ARN devait posséder les capacités structurales et fonctionnelles requises afin d’exploiter les interactions ARN-ligand dans la modulation de l’expression génétique [
9–
14]. En parallèle, un nombre grandissant d’exemples ont montré que la régulation génétique de certains opérons ne pouvait pas être expliquée par les modèles classiques mettant en jeu des facteurs protéiques [
15]. Plus précisément, une série de travaux effectués sur le gène btuB de Escherichia coli et sur l’opéron cob de Salmonella thyphimurium, qui ont la charge du maintien de la concentration cellulaire adéquate du coenzyme B12, ont établi que ce coenzyme participait au mécanisme de rétro-inhibition de l’expression de btuB et cob [
16–
18]. Cependant, plusieurs tentatives destinées à identifier les facteurs protéiques de la régulation génétique ont été vaines [
12,
19]. Aucun facteur protéique n’ayant pu être détecté, il apparaissait plausible qu’une interaction directe entre le coenzyme et l’ARNm devait exister. L’équipe de Ronald Breaker à l’Université de Yale a effectivement démontré que la formation d’un complexe entre le coenzyme B12 et la partie 5’ non-traduite de btuB pouvait induire des changements structuraux dans cet ARN [
20,
21]. Plusieurs autres exemples similaires ont été trouvés notamment pour le gène lysC de Bacillus subtilis (B. subtilis) où l’on observe la réorganisation de la structure de la région 5’ non-traduite de l’ARNm lorsque ce dernier forme un complexe avec la lysine, ayant pour effet l’atténuation du niveau de transcription en l’absence de cofacteur protéique [
22]. |
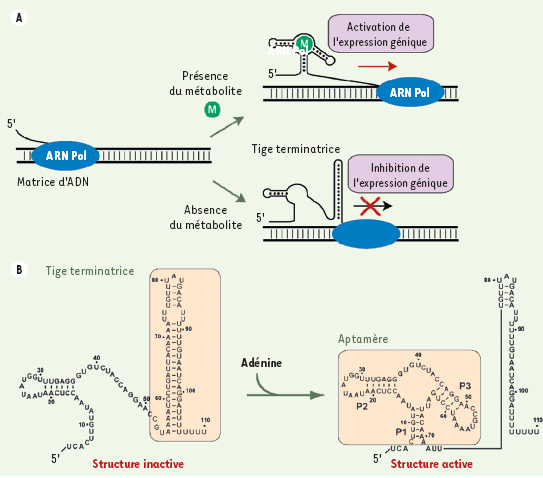
Composition structurale des riborégulateurs On peut donc décrire les riborégulateurs comme des séquences d’ARN situées dans la région non codante de certains ARNm pouvant lier un métabolite de façon spécifique menant à la réorganisation allostérique de sa structure, ce qui permet la régulation du gène situé en aval (Figure 1A). Les riborégulateurs sont composés de deux domaines fonctionnels (Figure 1B) [
23]. La première structure formée lors du processus de la transcription est désignée sous le nom d’« aptamère » et se révèle être un module de reconnaissance du métabolite. Toutes les classes de riborégulateurs possèdent un aptamère qui leur est propre et qui est spécifique du métabolite auquel ils sont rattachés. La deuxième structure est désignée par la dénomination « plate-forme d’expression », structure généralement localisée immédiatement en aval de l’aptamère. Cependant, les deux domaines se chevauchent partiellement, ce qui est très important dans le processus de régulation génétique. La fonction de la plateforme d’expression est de traduire le phénomène de liaison entre le métabolite et l’aptamère en une activité de modulation de l’expression génétique du gène en aval. La plate-forme d’expression est beaucoup moins conservée au cours de l’évolution comparativement à l’aptamère, ce qui reflète la plasticité de l’ARN à effectuer une fonction similaire à partir de structures diverses [
24]. 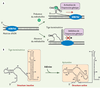 | Figure 1.
Le mécanisme d’action des riborégulateurs. A. Régulation transcriptionnelle d’un riborégulateur. Pendant la transcription, la partie 5’ non-traduite d’un ARNm contenant un riborégulateur forme des structures secondaires spécifiques. Ces structures sont des récepteurs pour des métabolites (M) cellulaires. Si le métabolite est en concentration suffisante, un complexe se forme entre le riborégulateur et le métabolite, ce qui réorganise la structure de l’ARNm et permet la transcription de l’ARNm. En revanche, en l’absence du métabolite, la structure du riborégulateur forme un motif tige-boucle (tige terminatrice), ce qui engendre l’arrêt prématuré de la transcription de l’ARNm et, par le fait même, l’inhibition de l’expression du gène. B. Le riborégulateur adénine chez Bacillus subtilis. La structure associée à l’inhibition de l’expression est caractérisée par une tige terminatrice connue pour son rôle dans l’atténuation de la transcription. La structure active est caractérisée par la présence de l’aptamère qui est organisé autour d’une jonction 3 voies composées des tiges P1, P2 et P3. Le métabolite adénine est reconnu par la région de l’aptamère. En présence de l’adénine, le complexe aptamère-adénine empêche la formation de la tige terminatrice, ce qui permet l’élongation de l’ARNm. |
|
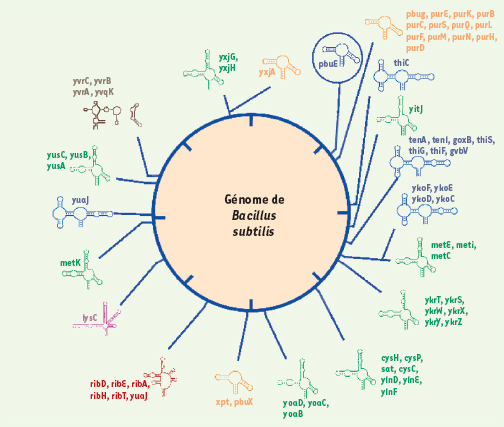
Les différentes familles de riborégulateurs Le champ de recherche sur les riborégulateurs est en pleine expansion ; il faut dire que plusieurs types de riborégulateurs sont maintenant connus (Figure 2). Ceux-ci sont régulés par les métabolites adénine [24], coenzyme B12 [20, 21], flavine mononucléotide [
25,
26], guanine [
27], glucosamine-6-phosphate [
28], glycine [
29], lysine [22,
30], S-adénosylméthionine [
31–
33] et thiamine pyrophosphate [25,
34]. Certains de ces riborégulateurs possèdent des particularités uniques. Par exemple, le riborégulateur glucosamine-6-phosphate compte une activité de coupure autocatalytique qui se manifeste seulement en présence du ligand. L’ARNm est donc sectionné en amont du riborégulateur empêchant ainsi la traduction du gène régulé [28]. Le riborégulateur glycine est, quant à lui, le seul à compter deux aptamères similaires montrant envers le ligand une liaison coopérative qui est importante pour le mécanisme de régulation génétique [29]. Enfin, le riborégulateur thiamine pyrophosphate est le seul jusqu’à ce jour à avoir été identifié chez les eucaryotes. En effet, il a été repéré chez certains champignons tels que Aspergillus nidulans et Aspergillus fumigatus, ainsi que chez d’autres végétaux comme Arabidopsis thaliana [25, 34]. Chez ce dernier, le riborégulateur est localisé dans la section 3’ non-traduite de l’ARNm et laisse donc présager qu’il participerait au processus de stabilisation de l’ARNm.  | Figure 2.
Principaux riborégulateurs contenus dans le génome de Bacillus subtilis. Le riborégulateur adénine est encerclé en bleu. Les autres riborégulateurs sont représentés comme suit : riborégulateurs liant le coenzyme B12 en brun, liant le flavine mononucléotide en rouge, liant les purines en jaune, liant la lysine en rose, liant le S-adénosylméthionine en vert et liant le thiamine pyrophosphate en bleu. Les riborégulateurs liant la glycine et le glucosamine-6-phosphate ne sont pas représentés. |
|
Anatomie du riborégulateur adénine Le riborégulateur adénine est l’un des plus petits riborégulateurs identifiés jusqu’à présent (Figure 1B). Il est très similaire au riborégulateur guanine ; il montre cependant une grande spécificité et une grande affinité envers l’adénine [24]. Contrairement à la plupart des autres, le riborégulateur adénine active l’expression génétique lorsque son ligand est présent en concentration suffisante dans le milieu cellulaire. Jusqu’à présent, trois représentants de ce riborégulateur ont été recensés, soit chez B. subtilis, Clostridium perfringens et Vibrio vulnificus [24]. Le riborégulateur adénine possède deux types de plateforme d’expression pouvant réguler les processus de transcription ou de traduction. La plate-forme d’expression engagée dans la régulation de la transcription est caractérisée par une structure en forme de tige-boucle appelée « terminateur » tandis que celle engagée au niveau de la traduction module l’accessibilité du motif Shine-Dalgarno et le codon de démarrage de la traduction. Que ce soit pour la régulation de la transcription ou de la traduction, le principe qu’emploie le riborégulateur afin de réguler l’expression génétique repose sur la modulation de la structure secondaire de la partie 5’ non-traduite de l’ARNm (Figure 1A). En effet, dans le cas du riborégulateur adénine, une structure « inactive » sera adoptée en l’absence d’adénine tandis qu’une structure « active » sera adoptée en sa présence (Figure 1B). Cette régulation, dite positive, est possible puisque la formation du complexe ligand-aptamère permet l’adoption d’une structure empêchant la présence du terminateur (anti-terminateur). En revanche, pour la grande majorité des riborégulateurs, une régulation négative est observée où la stabilisation du terminateur est favorisée par la présence du ligand. |
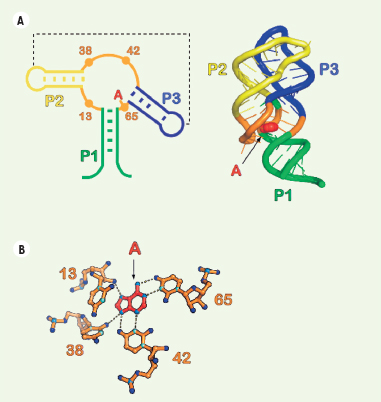
Regard cristallographique sur le complexe aptamère-ligand La région aptamère peut être considérée comme l’antenne du riborégulateur : elle permet de détecter la concentration d’un métabolite donné, ce qui atteste son importance dans le bon fonctionnement du mécanisme de régulation génétique. Afin de comprendre comment l’aptamère réussit à former un complexe de haute affinité et de grande spécificité envers l’adénine, plusieurs groupes de recherche ont produit des cristaux de ces complexes montrant une haute diffraction aux rayons X [
35–
37]. Les résultats obtenus montrent que l’aptamère adopte une structure compacte dans laquelle se manifeste une interaction de type boucle-boucle (Figure 3A) pouvant expliquer la conservation des bases dans les boucles [24, 27]. Des mutations empêchant la création de cette interaction empêchent aussi par conséquent la formation d’un complexe entre le ligand et l’aptamère [35,
41], ce qui suggère fortement que l’élaboration de l’interaction boucle-boucle est essentielle pour la reconnaissance du ligand par l’aptamère. 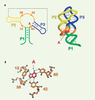 | Figure 3.
Structure cristalline du complexe adénine-aptamère. A. Représentation de la structure secondaire et tertiaire du complexe. La structure secondaire de l’aptamère est représentée à gauche. Les nucléotides importants pour la liaison de l’adénine (A) sont représentés (nucléotides 13, 38, 42 et 65). L’adénine est représentée près du nucléotide 65 puisque ces deux éléments interagissent via une interaction de type Watson-Crick. La ligne pointillée représente les deux régions impliquées dans l’interaction de type boucle-boucle. À droite, la structure tertiaire du complexe montre une structure compacte ainsi que la localisation du ligand au cœur de l’aptamère. Le cœur de la jonction se replie sur la tige P1 pour bien entourer le ligand : 97,8 % de la surface du ligand est inaccessible au solvant. Les tiges P1, P2 et P3 sont en vert, en jaune et en bleu, respectivement. L’adénine est représentée en rouge. B. Interactions entre les bases de l’aptamère et l’adénine. Les bases importantes à la liaison de l’adénine sont représentées ainsi que les ponts hydrogènes effectués avec l’adénine (rouge). La base en position 65 forme une paire de base Watson-Crick avec l’adénine, ce qui lui confère un rôle essentiel pour la liaison du ligand. |
La structure cristalline du complexe révèle que le ligand est presque totalement entouré par l’aptamère puisque 97,8 % de la surface du ligand est inaccessible au solvant [37]. Cela suggère que la reconnaissance spécifique du ligand par l’aptamère provient du fait que ce dernier utilise tous les atomes du ligand dans la formation du complexe. D’une façon étonnante, le résidu en position 65 de l’aptamère effectue un appariement de base de type Watson-Crick avec le ligand (Figure 3B) [37]. Cette interaction est primordiale pour la reconnaissance spécifique du ligand par l’aptamère, et il a été montré que cette interaction constitue le facteur de discrimination principal [24]. D’ailleurs, la conversion en un aptamère spécifique envers la guanine peut être effectuée en échangeant cette base contre une cytosine [24]. De plus, la liaison du ligand est aussi effectuée par trois bases uraciles situées à sa périphérie, le complexant ainsi par des ponts hydrogènes (Figure 3B). |
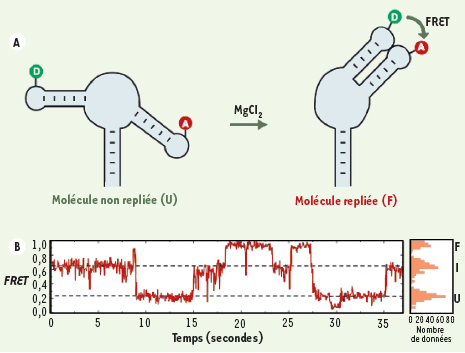
Notre laboratoire a récemment montré à l’aide de techniques biophysiques le rôle de l’adénine et des ions bivalents dans l’interaction de type boucle-boucle [41]. Plus particulièrement, nous avons utilisé la technique FRET (fluorescence resonance energy transfer) pour étudier la formation de cette interaction sous différentes conditions expérimentales. La technique FRET permet d’obtenir de l’information en solution sur la distance séparant deux fluorophores, un donneur et un accepteur (Figure 4A). Au cours de nos études portant sur le riborégulateur adénine, nous avons déterminé que l’interaction de type boucle-boucle se forme indépendamment de la présence de l’adénine. De plus, en utilisant la technique FRET de façon à étudier une unique molécule (single molecule FRET), il nous a été possible d’observer en solution la dynamique de formation de l’interaction boucle-boucle (Figure 4B). Nous avons déterminé qu’un intermédiaire structural caractérise le repliement de la molécule et que l’adénine participe à l’adoption de la structure naissante. Un tel intermédiaire pourrait être caractérisé par l’empilement des tiges P1 et P3 (Figure 1B), comme on l’observe dans la structure cristalline [37].  | Figure 4.
Dynamique de l’aptamère. A. Représentation de la molécule utilisée pour les études de FRET. Les fluorophores, donneur (D) et accepteur (A), sont représentés en vert et en rouge, respectivement. Une molécule ayant une structure non-repliée (U) adoptera une structure repliée (F) sous l’effet d’ions magnésium. La structure repliée est caractérisée par la formation de l’interaction de type boucle-boucle qui est détectable par FRET. B. Exemple d’un résultat obtenu grâce à la technique single molecule FRET. La dynamique de l’interaction de type boucle-boucle peut être étudiée dans le temps. On peut voir que le repliement de l’aptamère se fait de la forme non repliée (U) à la forme repliée (F) via un état intermédiaire (I). Le tableau de droite représente la proportion de chacun des états structuraux. |
|
Mécanisme de régulation du riborégulateur adénine Pour l’instant, peu d’études ont été réalisées sur le mécanisme de régulation du riborégulateur adénine. Par exemple, des études in vivo ont montré que ce riborégulateur modulait positivement l’expression d’un gène rapporteur chez B. subtilis en présence d’une concentration suffisante d’adénine [24]. En revanche, des expériences in vitro ont montré que la séquence complète du riborégulateur ne pouvait lier de façon productive l’adénine [
38, 41]. Ces résultats en apparente contradiction avec le concept général de réversibilité des riborégulateurs ont été publiés dans deux articles récents [38,
39]. Ces études montrent que la vitesse à laquelle la transcription est effectuée joue un rôle prépondérant dans le mécanisme de régulation du riborégulateur. Ce facteur additionnel de régulation provient du fait que la tige-boucle terminatrice est intrinsèquement très stable, empêchant la formation de l’aptamère (Figure 1). Donc, après la formation de cette tige-boucle, l’adénine ne peut plus être liée par le riborégulateur puisque l’aptamère ne peut se former. Le ligand peut donc se lier au riborégulateur uniquement lorsque l’aptamère seul est transcrit ; la liaison de l’adénine par la séquence partiellement transcrite du riborégulateur pourra ainsi donner naissance à un complexe stable. Lorsque l’élongation ultérieure du riborégulateur est effectuée, la structure tige-boucle terminatrice ne pourra être adoptée à cause de la présence du complexe aptamère-ligand. En résumé, ces études suggèrent que le fonctionnement du riborégulateur adénine chez B. subtilis est contrôlé par la cinétique de liaison du ligand et la vitesse de la transcription de l’ARN plutôt que par la simple affinité du ligand. Ce mode de fonctionnement se manifeste aussi avec d’autres riborégulateurs, comme celui modulé par le métabolite flavine mononucléotide (FMN) [26, 39]. Chez ce dernier, il a été démontré qu’il existe deux sites de pause transcriptionnelle, ce qui permettrait au ligand de se lier à l’aptamère au cours d’une durée plus longue. La mutation de ces sites de pause engendre une diminution de la formation du complexe ligand-aptamère [39]. En ce qui concerne le riborégulateur adénine, deux sites de pause ont été prédits, ce qui expliquerait la dépendance de la régulation génétique envers la cinétique de liaison du ligand et la vitesse de transcription [38]. |
Conclusions et perspectives Les études sur le riborégulateur adénine ont permis d’éclaircir la complexité de fonctionnement de cette petite séquence d’ARN dans la régulation de certains gènes. Malgré sa petite taille, l’aptamère adopte une structure tertiaire complexe qui pourrait expliquer sa haute affinité pour son propre ligand et son haut pouvoir de discrimination vis-à-vis des molécules analogues. Le mécanisme de régulation se révèle beaucoup plus complexe et ne repose pas simplement sur l’affinité du ligand envers le riborégulateur. La cinétique de liaison et la vitesse de transcription constituent des facteurs importants pour le fonctionnement du riborégulateur adénine. Ces résultats viennent soutenir l’idée de l’existence de sites de pause de l’ARN polymérase qui favoriseraient la formation de complexes riborégulateur-ligand ; ils offrent donc un nouveau regard sur le phénomène de régulation transcriptionnelle [26, 39]. La plupart des riborégulateurs modulent des gènes engagés dans plusieurs voies métaboliques essentielles [15]. Le recours à des métabolites analogues modulant artificellement l’expression de gènes essentiels à la survie d’agents microbiens pourrait offrir une nouvelle arme face à la résistance accrue des bactéries aux antibiotiques. Récemment, il a été démontré qu’un tel analogue, la pyrithiamine, inhibe le fonctionnement du riborégulateur relié à la production de la vitamine B1 [
40]. Afin de bénéficier du potentiel très considérable qu’offre l’utilisation des riborégulateurs comme moyens thérapeutiques, les études à venir devront approfondir les connaissances sur les interactions entre les riborégulateurs et leurs métabolites naturels. |
Footnotes |
1. Crick FH. On protein synthesis. Symp Soc Exp Biol 1958; 12 : 138–63. 2. Guerrier-Takada C, Gardiner K, Marsh T, et al. The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme. Cell 1983; 35 : 849–57. 3. Kruger K, Grabowski PJ, Zaug AJ, et al. Self-splicing RNA: autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena. Cell 1982; 31 : 147–57. 4. Ban N, Nissen P, Hansen J, et al. The complete atomic structure of the large ribosomal subunit at 2.4 A resolution. Science 2000; 289 : 905–20. 5. Breaker RR. In vitro selection of catalytic polynucleotides. Chem Rev 1997; 97 : 371–90. 6. Uphoff KW, Bell SD, Ellington AD. In vitro selection of aptamers: the dearth of pure reason. Curr Opin Struct Biol 1996; 6 : 281–8. 7. Wallis MG, Schroeder R. The binding of antibiotics to RNA. Prog Biophys Mol Biol 1997; 67 : 141–54. 8. Werstuck G, Green MR. Controlling gene expression in living cells through small molecule-RNA interactions. Science 1998; 282 : 296–8. 9. Gold L, Brown D, He Y, et al. From oligonucleotide shapes to genomic SELEX: novel biological regulatory loops. Proc Natl Acad Sci USA 1997 : 94 ; 59–64. 10. Gold L, Singer B, He YY, et al. SELEX and the evolution of genomes. Curr Opin Genet Dev 1997; 7 : 848–51. 11. Gelfand MS, Mironov AA, Jomantas J, et al. A conserved RNA structure element involved in the regulation of bacterial riboflavin synthesis genes. Trends Genet 1999; 15 : 439–42. 12. Nou X, Kadner RJ. Adenosylcobalamin inhibits ribosome binding to btuB RNA. Proc Natl Acad Sci USA 2000; 97 : 7190–5. 13. Stormo GD, Ji Y. Do mRNAs act as direct sensors of small molecules to control their expression ? Proc Natl Acad Sci USA 2001; 98 : 9465–7. 14. Miranda-Rios J, Navarro M, Soberon M. A conserved RNA structure (thi box) is involved in regulation of thiamin biosynthetic gene expression in bacteria. Proc Natl Acad Sci USA 2001; 98 : 9736–41. 15. Mandal M, Breaker RR. Gene regulation by riboswitches. Nat Rev Mol Cell Biol 2004; 5 : 451–63. 16. Lundrigan MD, Koster W, Kadner RJ. Transcribed sequences of the Escherichia coli btuB gene control its expression and regulation by vitamin B12. Proc Natl Acad Sci USA 1991; 88 : 1479–83. 17. Ravnum S, Andersson DI. Vitamin B12 repression of the btuB gene in Salmonella typhimurium is mediated via a translational control which requires leader and coding sequences. Mol Microbiol 1997; 23 : 35–42. 18. Richter-Dahlfors AA, Ravnum S, Andersson DI. Vitamin B12 repression of the cob operon in Salmonella typhimurium: translational control of the cbiA gene. Mol Microbiol 1994; 13 : 541–53. 19. Ravnum S, Andersson DI. An adenosyl-cobalamin (coenzyme-B12)-repressed translational enhancer in the cob mRNA of Salmonella typhimurium. Mol Microbiol 2001; 39 : 1585–94. 20. Nahvi A, Sudarsan N, Ebert MS, et al. Genetic control by a metabolite binding mRNA. Chem Biol 2002; 9 : 1043. 21. Nahvi A, Barrick JE, Breaker RR. Coenzyme B12 riboswitches are widespread genetic control elements in prokaryotes. Nucleic Acids Res 2004; 32 : 143–50. 22. Sudarsan N, Wickiser JK, Nakamura S, et al. An mRNA structure in bacteria that controls gene expression by binding lysine. Genes Dev 2003; 17 : 2688–97. 23. Winkler WC, Breaker RR. Genetic control by metabolite-binding riboswitches. Chem Biochem 2003; 4 ; 1024–32. 24. Mandal M, Breaker RR. Adenine riboswitches and gene activation by disruption of a transcription terminator. Nat Struct Mol Biol 2004; 11 : 29–35. 25. Mironov AS, Gusarov I, Rafikov R, et al. Sensing small molecules by nascent RNA: a mechanism to control transcription in bacteria. Cell 2002; 111 : 747–56. 26. Winkler WC, Cohen-Chalamish S, Breaker RR. An mRNA structure that controls gene expression by binding FMN. Proc Natl Acad Sci USA 2002; 99 : 15908–13. 27. Mandal M, Boese B, Barrick JE, et al. Riboswitches control fundamental biochemical pathways in Bacillus subtilis and other bacteria. Cell 2003; 113 : 577–86. 28. Winkler WC, Nahvi A, Roth A, et al. Control of gene expression by a natural metabolite-responsive ribozyme. Nature 2004; 428 : 281–6. 29. Mandal M, Lee M, Barrick JE, et al. A glycine-dependent riboswitch that uses cooperative binding to control gene expression. Science 2004; 306 : 275–9. 30. Grundy FJ, Lehman SC, Henkin TM. The L box regulon: lysine sensing by leader RNAs of bacterial lysine biosynthesis genes. Proc Natl Acad Sci USA 2003; 100 : 12057–62. 31. Epshtein V, Mironov AS, Nudler E. The riboswitch-mediated control of sulfur metabolism in bacteria. Proc Natl Acad Sci USA 2003; 100 : 5052–6. 32. McDaniel BA, Grundy FJ, Artsimovitch I, et al. Transcription termination control of the S box system: direct measurement of S-adenosylmethionine by the leader RNA. Proc Natl Acad Sci USA 2003; 100 : 3083–8. 33. Winkler WC, Nahvi A, Sudarsan N, et al. An mRNA structure that controls gene expression by binding S-adenosylmethionine. Nat Struct Biol 2003; 10 : 701–7. 34. Winkler W, Nahvi A, Breaker RR. Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression. Nature 2002; 419 : 952–6. 35. Batey RT, Gilbert SD, Montange RK. Structure of a natural guanine-responsive riboswitch complexed with the metabolite hypoxanthine. Nature 2004; 432 ; 411–5. 36. Gilbert SD, Stoddard CD, Wise SJ, et al. Thermodynamic and kinetic characterization of ligand binding to the purine riboswitch aptamer domain. J Mol Biol 2006; 359 : 754–8. 37. Serganov A, Yuan YR, Pikovskaya O, et al. Structural basis for discriminative regulation of gene expression by adenine- and guanine-sensing mRNAs. Chem Biol 2004; 11 : 1729–41. 38. Wickiser JK, Cheah MT, Breaker RR, et al. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry 2005; 44 : 13404–14. 39. Wickiser JK, Winkler WC, Breaker RR, et al. The speed of RNA transcription and metabolite binding kinetics operate an FMN riboswitch. Mol Cell 2005; 18 : 49–60. 40. Sudarsan N, Cohen-Chalamish S, Nakamura S, et al. Thiamine pyrophosphate riboswitches are targets for the antimicrobial compound pyrithiamine. Chem Biol 2005; 12 : 1325–35. 41. Lemay JF, Penedo JC, Tremblay R, et al. Folding of the adenine riboswitch. Chem Biol 2006; 13 : 857–68. |