Les déplétions de l’ADN mitochondrial (ADNmt) sont caractérisées par une diminution du nombre de molécules d’ADNmt et sont une cause importante des déficits de la chaîne respiratoire. De nombreux gènes nucléaires contrôlent le nombre de copies d’ADNmt et ces anomalies quantitatives sont les conséquences de mutations de certains de ces gènes. Des mutations dans cinq gènes nucléaires ont été rapportées dans des déplétions de l’ADNmt. Ces gènes sont impliqués dans la réplication de l’ADNmt (POLG [ 1] qui code l’ADN polymérase mitochondriale) et dans la voie de récupération des dNTP mitochondriaux (DGUOK [ 2], TK2 [ 3] codant respectivement pour la désoxyguanosine kinase et la thymidine kinase mitochondriales). Le dernier gène identifié, MPV17 [ 4], code pour une protéine localisée dans la mitochondrie mais dont la fonction est inconnue à ce jour. Cependant, pour une majorité des patients avec déplétion de l’ADNmt, le gène muté reste à identifier. La grande hétérogénéité clinique des patients avec déplétions de l’ADNmt suggère une grande hétérogénéité génétique et la découverte de nouveaux gènes de déplétions de l’ADNmt est rendue difficile par la petite taille des familles.
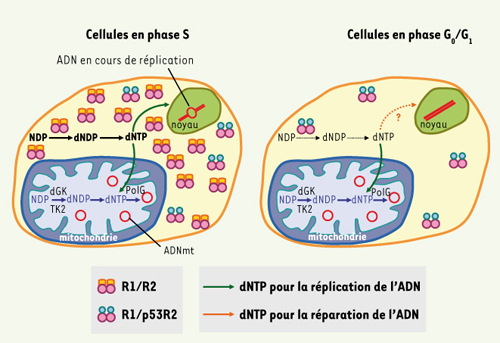
| Figure 1.
Synthèse des désoxyribonucléotides chez les mammifères au cours de la phase S (gauche) et en phase G0/G1. Au cours de la phase S, les dNTP sont synthétisés par le complexe ribonucléotide réductase R1/R2. Dans les cellules non prolifératives, sans protéine R2, les dNTP sont synthétisés par le complexe R1/p53R2. La voie de récupération des dNTP mitochondriaux participe au maintien du pool des dNTP mitochondriaux. |
Une analyse de liaison réalisée sur une famille consanguine présentant une déplétion très sévère de l’ADNmt dans le muscle (1 % de la quantité normale d’ADNmt) nous a permis d’identifier une région sur le chromosome 8 avec un lod score significatif de 3,3. Parmi les quelques 60 gènes de la région, nous avons bien évidemment étudié les gènes codant pour des protéines avec une fonction ou une localisation mitochondriales mais sans succès. Nous nous sommes ensuite intéressés au gène p53R2 qui code pour une petite sous-unité de la ribonucléotide réductase.
La ribonucléotide réductase est une enzyme cytoplasmique qui catalyse la réduction des nucléosides diphosphates (NDP) en dNDP [ 5], étape limitante dans la synthèse de l’ADN. Elle est constituée d’un homodimère de deux grosses sous-unités R1 et d’un homodimère de deux petites sous-unités R2. L’expression de R1 est constante et ubiquitaire alors que R2 est uniquement exprimée au cours de la phase S. La protéine R2 est spécifiquement dégradée en fin de mitose. p53R2 code pour une deuxième petite sous-unité qui a été identifiée en 2000 par deux laboratoires [ 6, 7]. Cet homologue de R2 (80 % d’identité), exprimé de façon constante, est une cible du facteur de transcription p53 et est ainsi appelé p53R2. La synthèse des dNTP dans les cellules en division est très majoritairement assurée par le complexe R1/R2 au cours de la phase S mais le complexe R1/p53R2 est également actif assurant la synthèse de 2 à 3 % des dNTP. Dans les tissus non prolifératifs, la sous-unité R2 est absente et ne permet donc plus la synthèse des dNTP par le complexe R1/R2. Or, dans ces cellules, une synthèse des dNTP est toujours nécessaire pour la réparation de l’ADN après des lésions. La découverte de la sous-unité p53R2 induite par p53 a alors laissé penser que le complexe R1/p53R2 permettait la synthèse de dNTP pour la réparation de l’ADN. Pensant avoir identifié un nouveau gène impliqué dans la tumorigenèse, le groupe de Nakamura a généré une souris p53R2−/− [ 8] afin d’étudier les anomalies de réparation de l’ADN. Malheureusement ces souris ne présentaient aucune tumeur mais développaient rapidement une insuffisance rénale à 4 semaines, un retard de croissance à 6 semaines et mourraient vers l’âge de 14 semaines. Le rôle de p53R2 dans la réparation de l’ADN n’était donc pas aussi évident qu’on le pensait d’autant que l’induction de p53R2 après des lésions de l’ADN n’engendre pas d’augmentation significative des pools de dNTP dans les cellules non prolifératives. De plus, l’induction de p53R2 par p53 est assez lente, avec un maximum 24 h après la lésion de l’ADN alors que la réparation de l’ADN semble complète quelques heures seulement après la lésion.
Les mitochondries contiennent des dNTP indispensables à la réplication de l’ADNmt. Ces dNTP mitochondriaux sont issus soit du transport des dNTP cytoplasmiques dans la mitochondrie soit d’une voie de récupération mitochondriale à partir de l’ADNmt. Cette voie de récupération est essentielle pour la réplication de l’ADNmt puisque des mutations dans des gènes de cette voie (DGUOK et TK2) conduisent à des déplétions sévères de l’ADNmt. Dans des cellules en prolifération le pool de dNTP cytoplasmiques est largement suffisant pour fournir la mitochondrie. En revanche, dans les tissus non prolifératifs, une synthèse continue de dNTP est également nécessaire à la réplication de l’ADNmt qui est indépendante du cycle cellulaire [ 9].
Le séquençage du gène p53R2 chez nos patients a permis d’identifier une mutation non-sens homozygote (Q284X). Nous avons également retrouvé des mutations faux-sens (E194K, W64R, E194G, C236F), de site d’épissage (IVS3-2 A→G) et une petite délétion (∆Glu85) de ce gène chez quatre autres patients issus de trois familles différentes non consanguines [ 10]. Tous les acides aminés mutés sont hautement conservés entre les espèces et ont été identifiés auparavant pour jouer un rôle important dans le fonctionnement de l’enzyme. Ces mutations mettent donc en évidence pour la première fois le lien entre la synthèse de dNTP catalysée par R1/p53R2 et la synthèse de l’ADNmt. Enfin, la découverte de déplétions de l’ADNmt dans le muscle, le rein et le foie de la souris p53R2−/− , prouve que p53R2 joue un rôle majeur dans la réplication de l’ADNmt. Il est aussi important de noter que les parents des enfants atteints, hétérozygotes pour les mutations p53R2, ne développent pas de tumeur.
La dernière surprise a été l’observation d’une induction de p53 dans les cellules (lignées lymphoblastoïdes, fibroblastes et cellules amniocytaires) de patients mutés dans p53R2. L’expression de p53 semble également augmentée dans les tubules du rein des souris p53R2−/− suggérant un mécanisme commun aux cellules humaines et aux tissus murins. On connaît maintenant des systèmes de réponse de gènes nucléaires à un dysfonctionnement mitochondrial tels que la régulation rétrograde chez la levure, l’augmentation du Ca2+ cytosolique ou l’activation de NFκB chez les mammifères [ 11]. Il est donc possible d’imaginer que p53 pourrait également participer à ce contrôle. Il serait intéressant de déterminer si l’induction de p53 est la conséquence d’un état de stress provoqué par les mutations du gène p53R2, par la déplétion de l’ADNmt, ou encore plus généralement par le déficit de la chaîne respiratoire dans ces cellules.