La traduction de l’information des gènes qui est inscrite dans l’ARN messager (ARNm) est catalysée par le ribosome. Cette machinerie moléculaire, universelle et essentielle, est le siège de la biosynthèse des protéines dans tous les organismes vivants. La traduction est aussi une des étapes clés de la régulation de l’expression des gènes. Bien que des défauts de régulation à ce niveau puissent entraîner des pathologies humaines, cet aspect demeure encore peu étudié d’un point de vue médical car les mécanismes moléculaires impliqués restent peu connus [ 1– 3]. Dans les bactéries, le contrôle traductionnel exerce une fonction clé dans tous les processus adaptatifs qui requièrent une réponse rapide, c’est-à-dire l’adaptation de la croissance bactérienne en réponse au stress ou aux conditions de l’environnement [ 4]. Les bactéries pathogènes représentent à l’heure actuelle un problème de santé publique, avec notamment l’apparition de résistances multiples aux antibiotiques. Le ribosome bactérien, en particulier, et les ARN ribosomiques (ARNr), sont la cible de plus de la moitié des antibiotiques [ 5]. Ainsi, mieux comprendre à l’échelle moléculaire le fonctionnement du ribosome et son contrôle devrait à plus long terme favoriser le développement de nouvelles stratégies à visée thérapeutique.
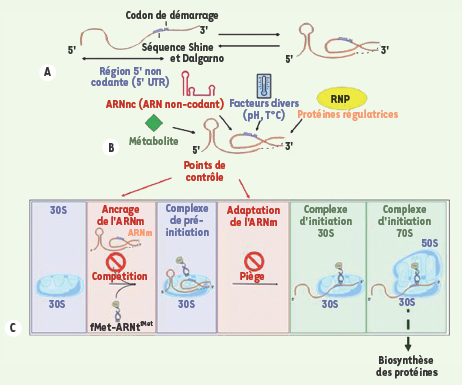
La machinerie du ribosome traverse des phases d’initiation, d’élongation et de terminaison. L’initiation, étape limitante de la traduction, est un processus à plusieurs étapes qui conduit au positionnement précis de l’ARNm pour établir les premières interactions de décodage avec l’ARNt initiateur aminoacylé au niveau du site peptidyl (P) du ribosome, en présence des facteurs d’initiation [ 6– 8]. Dans ce complexe, l’ARNm se trouve localisé dans un sillon au niveau du « cou » de la petite sous-unité ribosomique. Chez les bactéries, la première étape de la formation du complexe d’initiation passe par un complexe de pré-initiation au niveau de la petite sous-unité 30S dans laquelle l’interaction ARNm-ARNt n’a pas encore eu lieu (Figure 1). C’est souvent à cette étape que la régulation a lieu et l’ARNm en est un acteur essentiel. En général, la reconnaissance ARNm-ribosome requiert la présence de signaux spécifiques (séquence Shine et Dalgarno en amont du codon d’initiation) placés dans une région en simple brin de l’ARNm. Cependant, la plupart des régions 5’ non codantes des ARNm présentent des structures secondaires complexes qui peuvent influer sur la cinétique d’assemblage du complexe de pré-initiation. Ces structures peuvent répondre directement à un signal extérieur comme la température, et indirectement par la reconnaissance spécifique de facteurs comme des métabolites, des ARN non-codants (ARNnc) ou des protéines régulatrices (Figure 1). Chez les bactéries, deux mécanismes sont actuellement connus (Figure 1) [ 9] : (1) la compétition pour le site d’initiation de l’ARNm auquel se lie exclusivement le ribosome ou, alternativement, un facteur de régulation (protéine, ARNnc, métabolites) qui masque les signaux de reconnaissance du ribosome ; et (2) le piégeage du ribosome par un ARNm replié, qui, dans certains cas, est stabilisé par une protéine régulatrice. Ce dernier mécanisme offre l’avantage qu’il ne nécessite pas la formation d’un complexe très stable entre l’ARNm et le répresseur. Par ailleurs, le blocage du complexe de pré-initiation peut être levé avant que la dégradation de l’ARNm réprimé ne s’opère. Alors que le mécanisme de compétition a été défini au niveau moléculaire pour plusieurs exemples [4], aucune donnée n’existait jusqu’à présent sur le mécanisme par lequel la structure de l’ARNm piégerait le ribosome.
| Figure 1.
La structure de l’ARNm module l’efficacité de la traduction. La plupart des ARNm bactériens possèdent des motifs structuraux (A) qui sont sensibles à de nombreux facteurs environnementaux comme la température, ou qui interagissent spécifiquement avec un facteur agissant en trans
(B) et qui répondent à un signal donné (métabolites, ARN non codant, protéine). Un changement de structure de l’ARNm peut conduire soit à une activation de la traduction (en rendant accessible le site de fixation du ribosome), soit à une inhibition (en masquant les signaux de reconnaissance par le ribosome dans la région d’initiation de l’ARNm qui contient fréquemment la séquence Shine et Dalgarno spécifique des bactéries). C. L’initiation de la traduction implique plusieurs étapes successives dont la formation d’un complexe de pré-initiation où l’interaction ARNm-ARNt initiateur n’a pas encore eu lieu (en encadré). La plupart des régulateurs agissent à cette étape, soit en empêchant le ribosome d’accéder à son site (compétition), soit en le piégeant dans un complexe de pré-initiation inactif (piège). S’il y a adaptation de l’ARNm, le complexe d’initiation 30S peut se former, pour ensuite s’assembler avec la sous-unité ribosomique 50S et former le complexe d’initiation 70S du ribosome prêt à commencer la synthèse des protéines. |
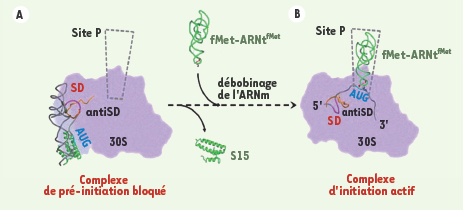
Dans le cadre d’une collaboration étroite entre chercheurs de l’IGBMC et de l’IBMC, associant des études fonctionnelles et structurales, il a été possible de visualiser directement, par microscopie électronique et reconstruction tridimensionnelle, comment la structure de l’ARNm est capable de bloquer le ribosome de façon transitoire (Figure 2A). Chez les entérobactéries, de nombreuses protéines qui reconnaissent l’ARN, comme les protéines ribosomiques, régulent négativement la traduction de leur ARNm en se fixant à des structures particulières, souvent localisées près du site de démarrage de la traduction et qui miment leur substrat naturel [4]. Dans des conditions favorables de croissance, les ribosomes représentent environ 50 % de la masse cellulaire, et une grande partie de l’énergie est donc utilisée par la machinerie de traduction. Par mesure d’économie, la synthèse des constituants du ribosome et leur assemblage sont finement coordonnés en fonction du taux de la croissance bactérienne. Alors que la synthèse des ARNr est régulée au niveau transcriptionnel, la synthèse des protéines ribosomiques (dont les gènes sont organisés en opérons) est contrôlée et coordonnée au niveau post-transcriptionnel [ 10]. Si la synthèse de l’ARNr diminue (en cas de carence en nutriments), la protéine régulatrice libre qui exerce souvent un rôle clé dans l’assemblage du ribosome, réprime la synthèse de son opéron au niveau traductionnel, en reconnaissant une région de l’ARNm qui mime son site de fixation au niveau de l’ARNr. Ainsi, le taux des protéines ribosomiques est constamment ajusté à celui de l’ARNr libre pour adapter rapidement la croissance bactérienne à ses environnements multiples. La protéine ribosomique S15 d’Escherichia coli, qui est requise pour l’assemblage du domaine central de la petite sous-unité du ribosome, empêche le démarrage de la traduction de son propre ARNm en bloquant le ribosome sous la forme d’un complexe de démarrage improductif (mécanisme de piégeage). La structure de ce complexe a révélé que la protéine S15 stabilise un repliement particulier de l’ARNm, appelé « pseudo-nœud », à l’entrée du ribosome qui empêche l’ARNm de se loger dans le sillon de la petite sous-unité ribosomique 30S (Figure 2A). La formation du complexe d’initiation de la traduction est alors bloquée et, par conséquent, la synthèse de la protéine ne peut plus commencer. Une fois que l’ARNm se déplie (par exemple, lorsque la protéine régulatrice S15 se dissocie de l’ARNm en présence d’ARNr libre), le complexe d’initiation peut alors se former et le premier événement de décodage entre l’ARNm et l’ARNt initiateur a lieu à l’intérieur du ribosome (Figure 2B).
| Figure 2.
La structure repliée de l’ARNm peut piéger le ribosome dans un complexe de pré-initiation inactif. La fixation des ARNm structurés au niveau du ribosome implique une reconnaissance initiale (ancrage) suivi d’une accommodation du site de fixation de l’ARNm pour initier la traduction. A. La protéine ribosomique S15 stabilise une structure de l’ARNm en pseudo-nœud et empêche l’étape d’accommodation, piégeant le ribosome dans un complexe de pré-initiation inactif. L’étude de ce complexe de pré-initiation inactif par cryo-microscopie électronique et reconstruction 3D de particules isolées révèle que l’ARNm se trouve stabilisé dans un site bien défini à la surface de la petite sous-unité 30S [
11]. Des analyses de séquences des ARN et protéines constituant ce site suggèrent que ce site est conservé à travers l’évolution. Il pourrait donc être utilisé par tout type de structure d’ARNm lors de la reconnaissance initiale par le ribosome. B. En l’absence de la protéine ribosomique S15, l’ARNm se déplie pour induire l’interaction avec l’ARNt initiateur dans le site P. Le mécanisme de « débobinage » de l’ARNm n’est pas encore bien compris et pourrait impliquer plusieurs composants de la machinerie du ribosome. |
En nous fondant sur la comparaison de plusieurs structures tridimensionnelles de complexes ribosome/ARNm, et sur des analyses de séquences du site du ribosome de différentes espèces, nous proposons que différents types de structures d’ARNm interagissent dans une région précise du ribosome, localisée à la surface de la petite sous-unité (Figure 2A). Il s’agirait d’un site dédié pour la liaison d’ARNm régulateurs et structurés que l’on retrouve aussi bien dans les bactéries, dans certains virus pathogènes (qui contiennent un signal d’entrée interne des ribosomes, IRES, pour détourner le ribosome humain à leur profit) et chez l’homme.
En tout état de cause, nous sommes encore loin d’avoir découvert l’ensemble des réseaux de régulation dépendant de la structure des ARNm aussi bien chez les bactéries que chez les eucaryotes. Cependant, cette étude qui vient d’être publiée dans le journal Cell [11], révèle un mécanisme à caractère général (ribosome en stand-by) qui contrôle la traduction de l’information contenue dans un certain gène, et cela en fonction des besoins cellulaires. Cette étude met aussi en évidence un intermédiaire réactionnel de l’initiation de la traduction, visualisé pour la première fois en 3D, qui représente une étape clé de la reconnaissance ARNm-ribosome. Les aspects dynamiques de la liaison de l’ARNm régulateur au ribosome et du « débobinage » de l’ARNm pourront ainsi être étudiés à l’avenir…