| |
| Med Sci (Paris). 2007 November; 23(11): 997–1001. Published online 2007 November 15. doi: 10.1051/medsci/20072311997.La prévalence et la distribution des maladies génétiques au Québec L’impact du passé sur le présent Anne-Marie Laberge* Service de génétique médicale, Hôpital Sainte-Justine, Montréal, Québec, Canada Institute for Public Health Genetics, University of Washington, Box 357236, Seattle, Washington 98195, États-Unis |
L’avancement des connaissances et le développement de nouvelles technologies dans le domaine de la génétique ont permis de découvrir les gènes et les mutations en cause dans la plupart des maladies génétiques retrouvées au Québec. Bien que la science moderne soit à l’origine de ces découvertes, la distribution des maladies génétiques sur le territoire du Québec est intimement reliée à son passé : l’histoire du peuplement du Québec a influencé la distribution et les caractéristiques des maladies génétiques que l’on y trouve. La fréquence et les particularités cliniques de ces maladies influencent à leur tour les gestes et les décisions des professionnels de la santé. |
Histoire du peuplement du Québec Entre la fondation de la ville de Québec en 1608 et la conquête de la Nouvelle-France par les Anglais en 1759, 25 000 colons sont venus en Nouvelle-France [
1]. Cependant, seulement 8 500 d’entre eux (dont 1 600 femmes) y ont laissé une descendance [1]. La majorité des immigrants en Nouvelle-France venaient de France, mais certaines régions de France sont surreprésentées parmi les immigrants, notamment les régions de la côte atlantique et des environs de Paris [
2]. Avant 1680, l’immigration est la principale source de croissance de la population de Nouvelle-France [
3]. Par la suite, la croissance naturelle dépasse l’immigration. Plus un colon est arrivé tôt en Nouvelle-France, plus il est probable que sa contribution génétique à la population actuelle soit élevée. Les 2 600 colons arrivés avant 1680 représentent environ deux tiers du pool génétique de la population québécoise actuelle [3]. En 1680, ils ont déjà une ou deux générations de descendants, ce qui leur assure une plus grande contribution génétique que les colons arrivés plus tard. Après la conquête anglaise de 1759, la population canadienne française se mélange peu avec les nouveaux arrivants anglophones. À cette époque, la majorité des 60 000 Canadiens français sur le territoire du Québec actuel vivent le long du fleuve St-Laurent [
4]. La migration vers l’intérieur des terres se fait surtout au xixe siècle. De nos jours, le Québec compte plus de 7 millions d’habitants, dont environ 80 % descendent des immigrants français arrivés au temps de la Nouvelle-France. Parmi le reste de la population, on compte principalement les Premières Nations et les immigrants arrivés après la conquête anglaise : anglophones (Anglais, Écossais et Irlandais), Italiens, Juifs, Allemands, Haïtiens, Chinois et Grecs [
5]. Parmi les immigrants anglophones, l’influence irlandaise est particulièrement importante. Entre 1846 et 1851, la famine force nombre d’Irlandais à quitter leur pays. L’immigration irlandaise au Québec est si importante qu’en 1871, 10 à 15 % de la population québécoise est d’origine irlandaise [
6]. De plus, les unions entre Irlandais et Canadiens français sont favorisées par leur religion commune, le catholicisme. La présence francophone en Amérique du Nord ne se limite pas au Québec. Dans les Maritimes, la présence acadienne date du début du XVIIe siècle [4]. La déportation des Acadiens en 1755 par l’armée britannique a disséminé la présence acadienne le long de la côte Atlantique, jusqu’en Louisiane. Certains Acadiens se sont réfugiés au Québec [4]. De nombreux explorateurs, voyageurs et coureurs des bois d’origine canadienne française ont participé à l’exploration de l’Ouest canadien et américain aux xviiie et xixe siècles. Au tournant du xxe siècle, plus d’un demi-million de Canadiens français ont quitté le Québec pour la Nouvelle-Angleterre à la recherche de travail. En 2000, 2,3 millions d’Américains se disent de descendance canadienne française [
7]. |
Concepts de génétique des populations La génétique des populations étudie la structure et la composition génétique des populations. L’équilibre de Hardy-Weinberg permet de prédire la fréquence des allèles et des génotypes dans une population donnée pour un locus autosomique biallélique donné [
8]. Il ne s’applique que si les conditions suivantes sont respectées : population infiniment nombreuse, absence de nouvelles mutations, absence de migration, absence de sélection naturelle et unions aléatoires entre les individus. Tant que ces conditions sont respectées, la fréquence des allèles et des génotypes restera stable d’une génération à l’autre. En l’absence d’une ou de plusieurs de ces conditions, la fréquence des allèles variera d’une génération à l’autre, ce qui peut augmenter ou diminuer la prévalence de maladies génétiques dans la population. Dans la réalité, il est rare que toutes ces conditions soient réunies. Dans le cas de la population canadienne française du Québec, nous discuterons de l’absence de trois de ces conditions. Premièrement, la population de départ est issue d’une migration. Un effet fondateur survient lorsqu’une nouvelle population est créée à partir de la migration d’un nombre restreint d’individus appartenant à une même population mère. Si l’échantillon de population qui a donné naissance à la nouvelle population n’est pas génétiquement représentatif de la population mère, la fréquence de certains allèles dans la nouvelle population peut être différente de celle retrouvée dans la population d’origine [8]. Les colons français venus en Nouvelle-France étaient issus préférentiellement de certaines régions de la France et ne constituaient donc pas un échantillon représentatif de la population de la France en général [1]. Ceci a créé un premier effet fondateur. Au début de la colonie, les colons se sont d’abord installés le long du fleuve St-Laurent, puis ont migré progressivement vers l’intérieur des terres [4]. Ces mouvements migratoires successifs sur le territoire ont créé des effets fondateurs régionaux. L’effet fondateur régional le mieux documenté est celui du Saguenay/Lac-St-Jean, dont la population est issue de migrations successives de la côte de Beaupré, située en aval de la ville de Québec, vers la région de Charlevoix, puis de Charlevoix vers le Saguenay/Lac-St-Jean [
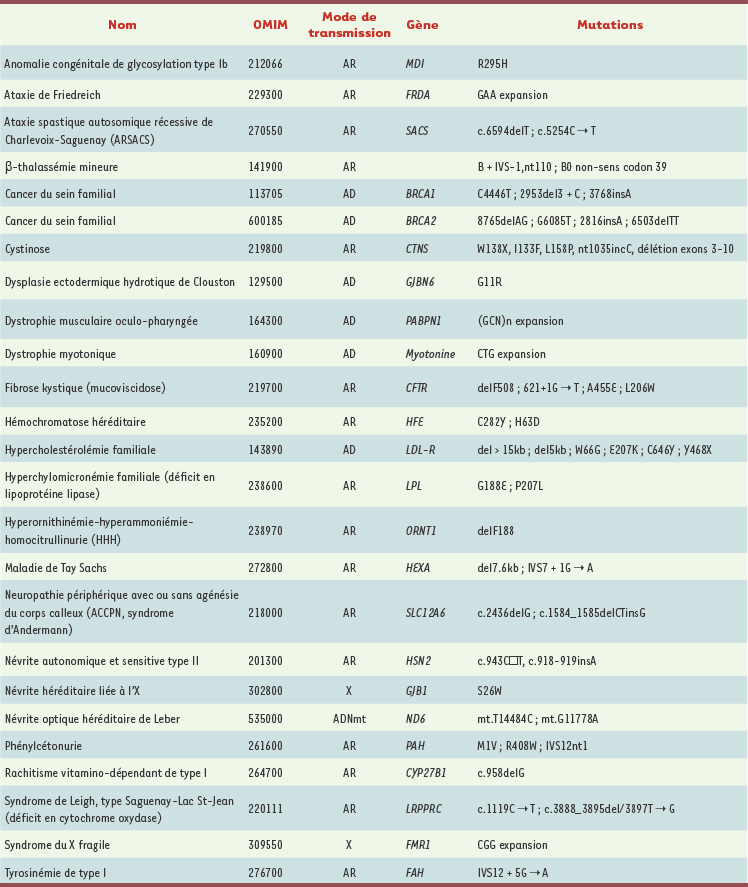
9]. C’est cette succession d’effets fondateurs qui fait que l’on observe de nos jours dans cette région une prévalence élevée de certaines maladies, comme la tyrosinémie, le syndrome de Leigh, la fibrose kystique, l’ARSACS, et l’ACCPN (Tableau I).  | Tableau I.
Maladies génétiques particulières à la population canadienne française du Québec (modifié d’après [
10]). OMIM : online Mendelian inheritance in man ( http://www.ncbi.nlm.nih.gov/sites/entrez ?db=OMIM). AD : autosomique dominant ; AR : autosomique récessif ; ADNmt : mitochondrial ; X : lié à l’X. |
Deuxièmement, la population de la Nouvelle-France était de taille limitée. La dérive génétique est, dans une population, la modification de la fréquence des allèles attribuable au hasard de la transmission [8]. Dans une population infiniment nombreuse, la fréquence des allèles devrait rester stable d’une génération à l’autre (en l’absence de mutations, migrations, sélections, ou d’unions non aléatoires). Plus la population est petite, plus le hasard de la transmission accroît la probabilité de faire varier la fréquence des allèles d’une génération à l’autre. Par analogie, il est beaucoup plus probable d’obtenir une fréquence observée de « piles » de 0,5 si l’on lance une pièce de monnaie un million de fois que si on ne la lance qu’une dizaine de fois. Plus le nombre d’essais est petit, plus la probabilité est forte que le hasard entraîne une fréquence observée différente de la fréquence attendue. Dans le cas de la population canadienne française, la petite taille de la population de départ a pu favoriser la dérive génétique et la modification de la fréquence de certains allèles, à la hausse ou à la baisse. Finalement, les unions n’étaient pas aléatoires. L’endogamie se dit d’unions dont les deux partenaires proviennent de la même population [8]. L’isolement linguistique, religieux et sociopolitique des Canadiens français avant et après la conquête anglaise ont favorisé l’endogamie. Il ne faut pas confondre l’endogamie avec la consanguinité. Une consanguinité étroite (à moins de 5 générations) est rare chez les Canadiens français [9]. Avec les générations, la consanguinité éloignée a augmenté à cause de la taille limitée de la population de départ et de la mobilité réduite des individus sur le territoire, mais cette consanguinité éloignée n’est pas plus fréquente qu’en Europe à la même époque, où la mobilité géographique et sociale était aussi limitée. Le portrait actuel des maladies génétiques au Québec résulte de l’ensemble des contributions de tous ces phénomènes (effets fondateurs, dérive génétique et endogamie). Bien que l’effet fondateur ne se produise qu’au moment de la migration d’un groupe vers un nouveau territoire, son impact sur la fréquence des allèles dans la population d’arrivée est ensuite intensifié ou diminué par le hasard de la transmission d’une génération à l’autre (dérive génétique) et par la présence d’endogamie. Il est donc difficile d’attribuer la prévalence ou la distribution d’une maladie donnée à un phénomène en particulier. |
Particularités québécoises de certaines maladies génétiques Plus d’une vingtaine de maladies héréditaires ont des particularités dans la population québécoise (Tableau I) [10,
11]. Certaines sont pratiquement exclusives à la population canadienne française, comme l’ataxie spastique de Charlevoix-Saguenay ou l’ACCPN [
12,
13]. D’autres sont plus fréquentes au Québec qu’ailleurs dans le monde, comme la tyrosinémie et la dystrophie musculaire oculo-pharyngée [
14,
15]. Finalement, certaines sont attribuables à des mutations particulières à la population canadienne française, dites « mutations fondatrices », sans nécessairement être plus fréquentes au Québec qu’ailleurs, comme la phénylcétonurie ou le cancer du sein héréditaire [
16–
18]. La distribution de ces maladies n’est pas uniforme sur le territoire québécois. Certaines se limitent presque exclusivement à une région, comme le syndrome de Leigh type Saguenay-Lac-St-Jean ou la béta-thalassémie (Portneuf) [
19,
20]. D’autres, comme l’hypercholestérolémie familiale, sont disséminées à peu près partout sur le territoire, leurs fréquences variant selon les régions [
21]. |
Conséquences de ces particularités sur le plan clinique La connaissance de ces particularités québécoises pour certaines maladies génétiques peut influencer différents aspects de la pratique clinique : le diagnostic différentiel, la mise au point d’un test diagnostique, ainsi que le choix du test diagnostique et l’interprétation de son résultat [
22]. Devant un tableau clinique donné, le clinicien doit avoir en tête un diagnostic différentiel approprié. Pour établir ce diagnostic différentiel, il lui est utile de connaître les maladies génétiques les plus courantes dans la population qu’il traite. Cela lui permet d’orienter ses analyses selon la probabilité plus ou moins grande de chacun des diagnostics possibles. Par exemple, si un patient québécois se présente avec une ataxie spastique, le test génétique pour l’ataxie spastique de Charlevoix-Saguenay (ARSACS) devrait figurer en tête des analyses diagnostiques [
23]. Bien que tous les cas d’ataxie spastique ne soient pas des cas d’ARSACS, il constitue un des diagnostics les plus probables et devrait être considéré parmi les premiers. La mise au point de tests diagnostiques par un laboratoire clinique doit tenir compte des particularités de la maladie dans la population locale. Dans certains cas, il est préférable de rechercher un phénotype biochimique plutôt qu’un génotype muté, surtout si le test phénotypique est techniquement plus simple à effectuer, moins coûteux, plus simple à interpréter ou constitue un meilleur indicateur de pronostic. Par exemple, plusieurs mutations canadiennes françaises fondatrices sont connues pour la phénylcétonurie, mais le diagnostic repose sur la mesure du niveau de phénylalanine plasmatique plutôt que sur l’analyse mutationnelle [16]. Le niveau de phénylalanine plasmatique est simple à mesurer et donne une bonne indication de la sévérité du phénotype, tandis que le décompte du nombre de mutations complique le test moléculaire et qu’en outre la sévérité du phénotype est difficile à prédire à partir du génotype. Pour choisir le test diagnostique approprié, il est important de connaître les caractéristiques de la maladie dans la population dont le patient est issu. Si une mutation fondatrice est connue dans cette population, il est important de s’assurer que cette mutation fait partie de celles qui seront recherchées par la technique utilisée. Dans le même ordre d’idée, la recherche exclusive des mutations fondatrices d’une population donnée ne permettra pas de repérer les cas de la maladie attribuables à d’autres mutations moins fréquentes ou affectant des individus issus d’autres descendances. Par exemple, un test moléculaire pour la tyrosinémie qui rechercherait la mutation IVS12 + 5G → A détecterait 95 % des allèles mutés chez les Canadiens français mais serait beaucoup moins sensible s’il était appliqué dans d’autres populations où cette mutation est rare ou inexistante [
24]. La connaissance des caractéristiques génétiques de la maladie est aussi utile pour interpréter les résultats du test. Pour un test moléculaire, la sensibilité et la spécificité du test, ainsi que ses valeurs prédictives – positive et négative –, seront influencées par le répertoire de mutations recherchées et leur fréquence dans la population ciblée, comme dans le cas de la fibrose kystique [
25]. L’utilité clinique du test doit également être prise en considération : si le diagnostic clinique ou phénotypique est établi, le diagnostic moléculaire n’est pas nécessairement utile à la prise en charge du patient. Dans un contexte de ressources limitées, le clinicien doit juger de la pertinence pour un patient donné de passer un test moléculaire souvent complexe et coûteux. Finalement, il est important de souligner que ce qui est vrai pour une population ne vaut pas toujours pour un individu. Un patient canadien français peut être porteur d’une mutation qui n’a pas été précédemment décrite dans cette population. Cela ne remet pas en question son appartenance à la population dont il est issu. Si le diagnostic clinique est clair, il pourra être confirmé par un test phénotypique ou par la recherche d’autres mutations. Par ailleurs, un patient qui n’est pas Canadien français peut être atteint d’une maladie « typiquement » canadienne française, comme dans le cas de l’ARSACS [
26,
27]. Cependant, les mutations en cause seront habituellement différentes. |
Une connaissance de la génétique de la population canadienne française et des particularités des maladies génétiques que l’on y retrouve est utile sur le plan clinique car elle permet de faire un diagnostic différentiel éclairé, de sélectionner les analyses diagnostiques à entreprendre, d’élaborer des tests adaptés à la population visée, d’interpréter les résultats de juste façon et d’utiliser les tests diagnostiques de manière appropriée [22]. Bien que ce texte porte sur la population canadienne française du Québec et ses caractéristiques génétiques, les concepts présentés ici peuvent s’étendre à d’autres populations, particulièrement aux populations soumises à un effet fondateur [22]. De plus, un tel principe s’applique non seulement aux maladies monogéniques que nous avons considérées mais également aux facteurs de prédisposition génétique dont la fréquence varie aussi d’une population à l’autre. |
1. De Braekeleer M, Dao TN. Hereditary disorders in the French Canadian population of Quebec. I. In search of founders. Hum Biol 1994; 66 : 205–23. 2. De Braekeleer M, Dao TN. Hereditary disorders in the French Canadian population of Quebec. II. Contribution of Perche. Hum Biol 1994; 66 : 225–49. 3. Charbonneau H, Desjardins B, Guillemette A, et al.
Naissance d’une population : les Français établis au Canada au xviie siècle. Paris et Montréal : Presses Universitaires de France et Presses de l’Université de Montréal, 1987 : 232 p. 4. Larin R. Brève histoire du peuplement européen en Nouvelle-France. Sillery : Éditions du Septentrion, 2000 : 226 p. 5. Institut de la Statistique du Québec. Recensement de la population 1996-1991-1986. Données comparatives et faits saillants. Cahier 3. Immigration, langue et origine ethnique. Publications du Québec, 1999 : 158 p. 6. McGowan-Jordan J, Stoddard K, Podolsky L, et al. Molecular analysis of cystinosis : probable Irish origin of the most common French Canadian mutation. Eur J Hum Genet 1999; 7 : 671–8. 7. Brittingham A, de la Cruz GP. Ancestry : 2000. Census 2000 brief. US Census Bureau, 2004 : 10. 8. Strachan T, Read AP. Human molecular genetics 3. 2nd ed. New York : Wiley-Liss, 2004; 576 : XXIII. 9. Bouchard G, De Braekeleer M. Histoire d’un génome : population et génétique dans l’est du Québec. Sillery : Presses de l’Université du Québec, 1991 : 607. 10. Laberge AM, Michaud J, Richter A, et al. Population history and its impact on medical genetics in Quebec. Clin Genet 2005; 68 : 287–301. 11. Scriver CR. Human genetics : lessons from Quebec populations. Annu Rev Genomics Hum Genet 2001; 2 : 69–101. 12. Engert JC, Berube P, Mercier J, et al. ARSACS, a spastic ataxia common in northeastern Quebec, is caused by mutations in a new gene encoding an 11.5-kb ORF. Nat Genet 2000; 24 : 120–5. 13. Dupré N, Howard HC, Mathieu J, et al. Hereditary motor and sensory neuropathy with agenesis of the corpus callosum. Ann Neurol 2003; 54 : 9–18. 14. De Braekeleer M, Larochelle J. Genetic epidemiology of hereditary tyrosinemia in Quebec and in Saguenay-Lac-St-Jean. Am J Hum Genet 1990; 47 : 302–7. 15. Brais B, Rouleau GA, Bouchard JP, et al. Oculopharyngeal muscular dystrophy. Semin Neurol 1999; 19 : 59–66. 16. Carter K, Byck S, Waters P, et al. Mutation at the phenylalanine hydroxylase gene (PAH) and its use to document population genetic variation : the Quebec experience. Eur J Hum Genet 1998; 6 : 61–70. 17. Chappuis PO, Hamel N, Paradis AJ, et al. Prevalence of founder BRCA1 and BRCA2 mutations in unselected French Canadian women with breast cancer. Clin Genet 2001; 59 : 418–23. 18. Tonin PN, Perret C, Lambert JA, et al. Founder BRCA1 and BRCA2 mutations in early-onset French Canadian breast cancer cases unselected for family history. Int J Cancer 2001; 95 : 189–93. 19. Merante F, Petrova-Benedict R, MacKay N, et al. A biochemically distinct form of cytochrome oxidase (COX) deficiency in the Saguenay-Lac-Saint-Jean region of Quebec. Am J Hum Genet 1993; 53 : 481–7. 20. Kaplan F, Kokotsis G, DeBraekeleer M, et al. Beta-thalassemia genes in French-Canadians : haplotype and mutation analysis of Portneuf chromosomes. Am J Hum Genet 1990; 46 : 126–32. 21. Vohl MC, Moorjani S, Roy M, et al. Geographic distribution of French-Canadian low-density lipoprotein receptor gene mutations in the Province of Quebec. Clin Genet 1997; 52 : 1–6. 22. Zlotogora J. Knowing the ethnic origin of the patient is important in making a diagnosis. Am J Med Genet 1998; 78 : 393–4. 23. Bouchard JP, Richter A, Melancon SB, et al. Autosomal recessive spastic ataxia (Charlevoix-Saguenay). In : Klockgether T, ed. Handbook of ataxia disorders. New York : Marcel Dekker, 2000 : 311–24. 24. Poudrier J, St-Louis M, Lettre F, et al. Frequency of the IVS12 + 5G ® A splice mutation of the fumarylacetoacetate hydrolase gene in carriers of hereditary tyrosinaemia in the French Canadian population of Saguenay-Lac-St-Jean. Prenat Diagn 1996; 16 : 59–64. 25. Palomaki GE, FitzSimmons SC, Haddow JE. Clinical sensitivity of prenatal screening for cystic fibrosis via CFTR carrier testing in a United States panethnic population. Genet Med 2004; 6 : 405–14. 26. El Euch-Fayache G, Lalani I, Amouri R, et al. Phenotypic features and genetic findings in sacsin-related autosomal recessive ataxia in Tunisia. Arch Neurol 2003; 60 : 982–8. 27. Criscuolo C, Banfi S, Orio M, et al. A novel mutation in SACS gene in a family from southern Italy. Neurology 2004; 62 : 100–2. |