| |
| Med Sci (Paris). 2007 November; 23(11): 1002–1007. Published online 2007 November 15. doi: 10.1051/medsci/200723111002.La cirrhose amérindienne infantile Andrea Richter, Grant A. Mitchell, and Andrée Rasquin* Services de génétique médicale et de gastroentérologie, Hôpital Sainte Justine, Département de pédiatrie, Université de Montréal, 3175, chemin de la Côte Sainte-Catherine, Montréal (Québec) H3T 1C5, Canada |
Bien que le site d’expression et quelques faits concernant les rôles possibles de cirhin soient maintenant connus, nous ne possédons toujours aucune hypothèse physiopathologique liant la protéine cirhin à l’étiologie de la maladie. Quoi qu’il en soit, nous pouvons espérer que la poursuite de la collaboration entre les familles des patients, les cliniciens et les chercheurs, qui a mené à la détection du gène et à la mise au point d’un test de dépistage, pourra conduire à la découverte d’un traitement pour cette maladie qui affecte les autochtones du Nord-Ouest québécois. Nous rappelons ici les étapes de nos travaux sur la cirrhose amérindienne infantile, maladie héréditaire qui n’a été décrite qu’au Québec [
1–
6]. |
Depuis 1970, nous avons suivi 36 enfants dont les seuls traits communs étaient leur origine amérindienne et la cirrhose de type biliaire dont ils seront inéluctablement victimes. La survenue de la maladie chez plusieurs enfants de la même famille, mais jamais chez leurs parents, a orienté notre diagnostic vers une maladie héréditaire de type autosomique récessif. L’enquête familiale a permis de préciser que ces enfants appartenaient à 5 grandes familles et à 3 communautés des Premières Nations habitant au Nord-Ouest du Québec. Ils sont tous d’origine Odjibway-Cree [3, 4]. L’évolution clinique de ces enfants est résumée dans la Figure 1. On compte une prédominance de garçons ; en outre, aucune caractéristique phénotypique n’a été relevée en dehors de la cirrhose. Il s’agit d’une maladie hépatique très sévère dont le taux de survie à l’âge adulte est inférieur à 50 %. La transplantation hépatique (TH) représente actuellement la seule solution thérapeutique. La maladie n’a pas resurgi dans les foies des 12 patients transplantés  | Figure 1.
Évolution clinique des 36 patients atteints de la cirrhose amérindienne.
|
De façon caractéristique, la cholestase néonatale se manifeste, dès les premiers jours de vie, par un ictère. Celui-ci disparaît après quelques semaines pour réapparaître progressivement quand la fonction hépatique se détériore. La complication majeure se manifeste par une hypertension portale très précoce avec hémorragie digestive par rupture de varices œsophagiennes dès l’âge de 8 mois [
3,
4]. |
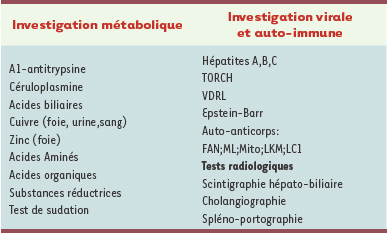
Après l’examen initial, les enfants ont fait l’objet d’une évaluation tous les 3 à 6 mois par un bilan hépatique standard et tous les ans par une échographie couplée à une étude par Doppler du foie. Sur le plan biochimique, on constate une augmentation persistante des phosphatases alcalines (PA, 400 à 900 U/l), des γ-glutamyl transférases (GGT, 150 à 500 U/l) et de la bilirubine directe (> 10 µmole/l) associée à une augmentation du taux des transaminases de 5 à 10 fois supérieur à la normale. Le Tableau I résume les différentes analyses qui ont été effectuées pour éliminer les causes de cirrhose déjà connues [1,
2]. L’investigation clinique a particulièrement porté sur les acides biliaires, puis sur le métabolisme du cuivre et du zinc puisque les familles des personnes atteintes habitent une région minière où sont extraits ces métaux. Toutes ces investigations se sont révélées négatives [3, 4]. Les études radiologiques de l’arbre biliaire n’ont pas montré d’images de sténoses compatibles avec une cholangite sclérosante [4]. L’étude histologique a été effectuée sur les biopsies hépatiques et sur les foies entiers prélevés lors des TH et des autopsies. L’image précoce est hétérogène, montrant, soit les caractéristiques d’une hépatite à cellules géantes, soit celle d’une cholestase obstructive avec prolifération ductulaire, bouchons biliaires et fibrose portale précoce. En revanche, l’évolution de la maladie hépatique aboutit chez tous les enfants à la cirrhose de type biliaire. À la microscopie électronique, on observe une accumulation importante des micro-filaments d’actine dans la région péri-canaliculaire, suggérant la possibilité d’une anomalie de fonctionnement à ce niveau [3]. La comparaison subséquente, en double aveugle, des images hépatiques de différentes pathologies cholestatiques du nouveau-né n’a pas permis de reconnaître la polymérisation des micro-filaments d’actine comme un marqueur distinctif de la maladie.  | Tableau I.
Examen clinique des 36 patients. TORCH : toxoplasmose, rubéole, cytomégalovirus, herpès ; FAN : facteur anti-nucléaire ; ML : muscles lisses ; Mito : mitochondries ; LKM : liver/kidney microsomal ; LC1 : liver cytosol. |
|
La génétique de la cirrhose amérindienne et la recherche fondamentale La localisation du gène L’homogénéité de la présentation clinique des patients, leur regroupement géographique et l’étude des arbres généalogiques suggéraient d’emblée une transmission de type autosomique récessif. Nous avons pu estimer que le taux de porteurs du gène défectueux se situait entre 8 et 12 % dans les populations exposées [ 4]. Ce taux est comparable à celui d’autres maladies des Premières Nations et il est le double de celui des porteurs de fibrose kystique. L’hypothèse selon laquelle nous serions ici en présence d’un effet dit « fondateur » a été formulée d’emblée, car elle est compatible avec l’histoire démographique de ces populations. Dans cette situation, un allèle mutant à haute fréquence se retrouve dans une population fondée par un groupe restreint d’ancêtres parmi lesquels un ou plusieurs individus portaient ce gène mutant [
7–
10]. Nous avons donc choisi la stratégie qui consiste à mettre en commun ( pooling) les ADN pour cartographier le locus. Celui-ci a été détecté dans la région chromosomique 16q22 où un haplotype de 4,9 centiMorgan était partagé de façon homozygote par tous les patients. Ce fait venait donc soutenir notre hypothèse d’un effet fondateur [
5]. Le dépistage de la mutation « cirhin » dans la cirrhose amérindienne La séquence des gènes exprimés dans le foie, qui se situaient dans la région chromosomique partagée par les patients, a été comparée à celle de leurs parents et à celle des sujets témoins. Cette mesure a permis d’identifier le gène responsable ( NAIC/CIRH1A). Ce gène code pour la protéine cirhin. En considérant sa séquence, sa fonction normale n’apparaît pas clairement d’emblée. Tous les patients testés jusqu’à présent sont homozygotes pour une mutation caractérisée par le remplacement de l’arginine 565 par le tryptophane (R565W) (Figure 2A). On peut prédire que cette mutation change la structure secondaire de la protéine. Nous avons mis au point un test de dépistage de la mutation R565W par RFLP, désormais disponible dans notre hôpital pour les familles dont les membres risquent d’être porteurs de la mutation (Figure 2B) [ 6].  | Figure 2.
A. Profils d’électrophorèse des ADN d’un patient atteint de la cirrhose amérindienne, un porteur obligatoire et un individu témoin. Les acides aminés correspondant à la séquence de l’ADN apparaissent au bas de la figure. La mutation induit le remplacement d’une molécule d’arginine par une molécule de tryptophane. B. La détection de la mutation par PCR-RFLP [ 6]. La mutation crée un site de restriction pour l’enzyme de restriction Alu I. L’exon 15 du gène CIRH1A est amplifié par PCR. Après la digestion avec Alu I, les ADN sont séparés par électrophorèse sur un gel d’agarose 3 %. Le fragment amplifié par PCR contient un site de restriction constant Alu I. Celui-ci sert de contrôle positif de la digestion et donne un fragment de 150 pb. Puits 1-3 : échantillons des patients. Puits 4-6 échantillons des hétérozygotes et puits 7-10 échantillons des individus homozygotes normaux (pb : paires de base). |
 | Figure 2.(suite)
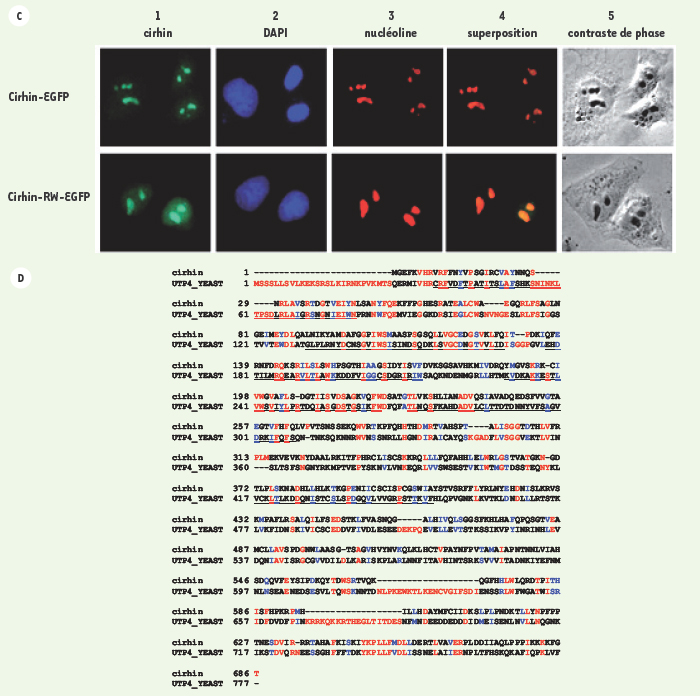
C. La localisation subcellulaire de cirhin-EGFP et R565W cirhin-EGFP dans les cellules HeLa. 1. La fluorescence verte de cirhin marquée avec EGFP. 2. coloration des noyaux avec Diamidino-4’,6-phénylindol-2 dichlorhydrate (DAPI). 3. visualisation des nucléoles par l’anticorps monoclonal de souris contre la nucléoline humaine (rouge). 4. superposition des images EGFP (vert) et nucléole (rouge). 5. image des cellules par microscopie en contraste de phase. D. Alignements des séquences protéiques de cirhin (humain) et UTP4 de (levure). Image produite par ClustalW 1,8 et Boxshade. Les acides amines identiques sont en rouge et les acides aminés similaires sont en bleu. Les domaines WD40 de cirhin sont indiqués au-dessus des résidus et les domaines WD40 de UTP4 sont soulignés. |
Caractéristiques des homologues de cirhin La protéine cirhin est conservée chez les vertébrés. Chez la souris, 88 % des acides aminés (AA) de la protéine homologue de cirhin (Tex292) sont identiques à ceux de l’humain. Le gène tex292 est exprimé dans tous les tissus et en particulier dans le foie et les testicules [
11,
12]. L’hybridation in situ chez l’embryon de souris de 11,5 jours révèle une expression intense du gène qui est prédominante dans le foie. L’expression diminue par la suite suggérant que la maladie puisse provenir d’une perturbation prénatale [ 6]. Chez le poisson zèbre, 53 % des AA de la protéine homologue de cirhin sont identiques à ceux de l’humain. Chez la levure S. cerevisiea, la protéine homologue UTP4, un composant du complexe nucléolaire U3, prend part à la biogenèse de l’ARN ribosomique [
13]. Une proportion de 23 % de la protéine UTP4 est semblable à celle de la protéine humaine. Le rôle de cirhin ? Une protéine qui réside dans le nucléole Les recherches en bioinformatique prédisent que cirhin contient des successions de WD40 et inclut de multiples signaux cibles qui pourraient la diriger vers différents organites cellulaires. Les protéines WD40 forment une grande famille engagée dans des fonctions diverses telles que le traitement de l’ARN, la transduction des signaux, le trafic vésiculaire, l’assemblage du cytosquelette et le contrôle du cycle cellulaire [
14]. La transfection transitoire de cellules HeLa (lignée cellulaire humaine) avec une protéine de cirhin marquée, grâce à l’EGPF (Enhanced Green Fluorescent protein), a montré que la protéine se retrouvait dans le nucléole. La mutation R565W n’altère pas cette localisation (Figure 2C). Deux séquences protéiques sont requises pour que la protéine de cirhin soit retrouvée dans le nucléole : un domaine C-terminal agissant comme signal fonctionnel de localisation nucléaire et un signal spécifique (120 AA entre les résidus M315 et K432 de cirhin), jamais décrit, pour la cible nucléolaire [
15]. Les bases de données actuelles sur le nucléole contiennent plus de 700 protéines incluant cirhin [
16,
17]. Parmi ses fonctions de base, le nucléole joue un rôle essentiel dans la biogenèse de l’ARN des ribosomes [
18,
19]. Chez les eucaryotes, la séquestration des protéines dans le nucléole représente un mécanisme assez commun de régulation du cycle cellulaire [
20,
21]. Cirhin est en interaction avec une protéine nucléaire Malgré sa localisation nucléolaire, nous avons formulé l’hypothèse que cirhin n’est pas en relation directe avec les acides nucléiques car elle ne contient pas de motifs de liaison avec l’ARN ou l’ADN. Son rôle pourrait favoriser l’interaction directe, protéine à protéine, et constituerait un rouage important d’un mécanisme moléculaire de base. Nous avons employé le système de double hybride chez la levure pour identifier les partenaires d’interaction de cirhin [21]. En utilisant cirhin comme appât, nous n’avons trouvé dans la bibliothèque des ADNc du foie humain qu’un seul partenaire qui réagissait fortement. Il codait pour la portion C-terminale d’une protéine connue, le HIVEP1 [
23]. Nous avons appelé cette partie interactive : cirip (Cirhin interaction protein). Pour déterminer si l’interaction entre cirhin et cirip est affectée par la mutation R565W, nous avons créé une souche de levure R565W Cirhin/Cirip. Nous avons montré que la souche mutante R565W Cirhin réagit avec cirip mais moins fortement qu’avec la protéine cirhin normale. L’étude in vitro sur des cellules humaines a révélé que cirhin et cirip agissent de concert comme régulateurs de transcription des sites NF-kB [
24,
25]. L’interaction de cirhin, protéine nucléolaire, avec HIVEP1, protéine nucléaire, dans la pathogénie de la maladie reste néanmoins indéterminée. Les études en cours et les directions futures de la recherche Pour déterminer si cirhin et son homologue chez la levure (UTP4) ont les mêmes fonctions, une expérience de complémentarité a été effectuée. Si on « complémente » avec cirhin la levure qui a une délétion dans UTP4 [
26], la viabilité de la souche n’est pas restituée. Cette découverte indique que les 2 protéines nucléolaires ne sont pas fonctionnellement identiques malgré la similarité de leurs séquences. De fait, la similarité de 23 % des séquences entre les 2 protéines peut être largement attribuée à la présence des motifs WD40 (Figure 2D) (Richter, Bussey, résultats non publiés). En collaboration avec le Dr M. Pack (Philadelphie), nous employons actuellement le poisson zèbre comme modèle de vertébré pour modéliser la cirrhose amérindienne. L’orthologue de cirhin sera invalidé avec de l’ARNi : cette technique est utilisée pour rendre silencieuse l’expression du gène ciblé. Si nous obtenons un phénotype viable, nous essayerons de faire des expériences de sauvetage avec cirhin et cirhin R565W. Il existe, chez la souris, une lignée de cellules souches embryonnaires, XH230, où la séquence tex292 a été inactivée. Nous avons obtenu des chimères qui ont produit les hétérozygotes souhaités (tex292+/-). Nous avons obtenu des portées de 5 à 6 souriceaux en moyenne et le génotypage n’a révélé aucun homozygote (tex292−/−
), qui conduirait à une mortalité prénatale. Notre hypothèse actuelle stipule que l’inactivation complète de tex292 est probablement létale au stade prénatal ([
27] et Yu et al., en préparation). Dans le but de produire un modèle animal qui se rapproche de la maladie chez l’humain, nous nous efforçons de créer une souris qui porte la mutation équivalente à R565W. |
L’observation clinique attentive de la cirrhose amérindienne infantile a mis les investigateurs sur le chemin de la recherche clinique puis fondamentale. Nous avons pu déterminer que les patients atteints de la cirrhose amérindienne étaient homozygotes pour la mutation R565W dans la protéine cirhin. Cette découverte a permis d’établir un test de dépistage pour les porteurs du gène défectueux. Les études expérimentales en cours devraient nous aider à mieux comprendre le rôle exact de cirhin et de proposer des hypothèses physiopathologiques, et, à terme, de trouver un traitement. Ces recherches constituent un exemple de collaboration étroite entre les patients, leurs familles, les communautés autochtones, ainsi que les cliniciens et les chercheurs de l’hôpital Sainte-Justine pour élucider la pathogénèse de maladies dépistées au Québec. |
Les auteurs remercient les Instituts de recherche en santé du Canada pour leur soutien financier.
|
1. Balistreri WF, Bezerra JA. Whatever happened to « neonatal hepatitis » ? Clin Liver Dis 2006; 10 : 27–53. 2. Suchy FJ. Neonatal cholestasis. Pediatr Rev 2004; 25 : 388–96. 3. Weber AM, Tuchweber B, Yousef I, et al. Severe familial cholestasis in North American Indian children : a clinical model of microfilament dysfunction ? Gastroenterology 1981; 81 : 653–62. 4. Drouin E, Russo P, Tuchweber B, et al. North American Indian cirrhosis in children : a review of 30 cases. J Pediatr Gastroenterol Nutr 2000; 31 : 395–404. 5. Betard C, Rasquin-Weber A, Brewer C, et al. Localization of a recessive gene for North American Indian childhood cirrhosis to chromosome region 16q22 and identification of a shared haplotype. Am J Hum Genet 2000; 67 : 222–8. 6. Chagnon P, Michaud J, Mitchell GA, et al. A Missense mutation (R565W) in cirhin (FLJ14728) in North American Indian childhood cirrhosis. Am J Hum Genet 2002; 71 : 1443–9. 7. Haworth JC, Booth FA, Chudley AE, et al. Phenotypic variability in glutaric aciduria type I : Report of fourteen cases in five Canadian Indian kindreds. J Pediatr 1991; 118 : 52–8. 8. Greenberg CR, Reimer D, Singal R, et al. A G-to-T transversion at the +5 position of intron 1 in the glutaryl CoA dehydrogenase gene is associated with the Island Lake variant of glutaric acidemia type I. Hum Mol Genet 1995; 4 : 493–5. 9. Robinson BH, Oei J, Sherwood WG, et al. The molecular basis for the two different clinical presentations of classical pyruvate carboxylase deficiency. Am J Hum Genet 1984; 36 : 283–94. 10. Weiler T, Greenberg CR, Nylen E, et al. Limb-girdle muscular dystrophy and Miyoshi myopathy in an aboriginal Canadian kindred map to LGMD2B and segregate with the same haplotype. Am J Hum Genet 1996; 59 : 872–8. 11. Lopez-Fernandez LA, del Mazo J. Characterization of genes expressed early in mouse spermatogenesis, isolated from a subtractive cDNA library. Mamm Genome 1996; 7 : 698–700. 12. Nagase T, Kikuno R, Ohara O. Prediction of the coding sequences of unidentified human genes. XXII. The complete sequences of 50 new cDNA clones which code for large proteins. DNA Res 2001; 8 : 319–27. 13. Dragon F, Gallagher JE, Compagnone-Post PA, et al. A large nucleolar U3 ribonucleoprotein required for 18S ribosomal RNA biogenesis. Nature 2002; 417 : 967–70. 14. Li D, Roberts R. WD-repeat proteins : structure characteristics, biological function, and their involvement in human diseases. Cell Mol Life Sci 2001; 58 : 2085–97. 15. Yu B, Mitchell GA, Richter A. Nucleolar localization of cirhin, the protein mutated in North American Indian childhood cirrhosis. Exp Cell Res 2005; 311 : 218–28. 16. Scherl A, Coute Y, Deon C, et al. Functional proteomic analysis of human nucleolus. Mol Biol Cell 2002; 13 : 4100–9. 17. Leung AK, Andersen JS, Mann M, Lamond AI. Bioinformatic analysis of the nucleolus. Biochem J 2003; 376 : 553–69. 18. Andersen JS, Lam YW, Leung AK, et al. Nucleolar proteome dynamics. Nature 2005; 433 : 77–83. 19. Olson MO, Dundr M, Szebeni A. The nucleolus : an old factory with unexpected capabilities. Trends Cell Biol 2000; 10 : 189–96. 20. Carmo-Fonseca M, Mendes-Soares L, Campos I. To be or not to be in the nucleolus. Nat Cell Biol 2000; 2 : E107–12. 21. Carmo-Fonseca M. The contribution of nuclear compartmentalization to gene regulation. Cell 2002; 108 : 513–21. 22. Toby GG, Golemis EA. Using the yeast interaction trap and other two-hybrid-based approaches to study protein-protein interactions. Methods 2001; 24 : 201–17. 23. Hicar MD, Liu Y, Allen CE, Wu LC. Structure of the human zinc finger protein HIVEP3 : molecular cloning, expression, exon-intron structure, and comparison with paralogous genes HIVEP1 and HIVEP2. Genomics 2001; 71 : 89–100. 24. Xiao W. Advances in NF-kappaB signaling transduction and transcription. Cell Mol Immunol 2004; 1 : 425–35. 25. Yu B, Mitchell GA, Richter A. Cirhin, the protein mutated in NAIC is a transcriptional cofactor of HIVEP1 DNA binding protein. 54th American Society of Human Genetics meeting, 2004 : 458 (abstract). 26. Winzeler EA, Shoemaker DD, Astromoff A, et al Functional characterization of the S. cerevisiae genome by gene deletion and parallel analysis. Science 1999; 285 : 901–6. 27. Yu B, Mitchell GA, Richter A. Embryonic lethality in mice deficient in tex292, ortholog of the North American Indian childhood cirrhosis (NAIC) gene CIRH1A. Salt Lake City : American Society of Human Genetics 55th Annual Meeting, octobre 2005 (abstract). |