| |
| Med Sci (Paris). 2007 December; 23(12): 1173–1176. Published online 2007 December 15. doi: 10.1051/medsci/200723121173.
Brainbow ou le cerveau en couleurs Jean Livet* Department of Molecular and Cellular Biology and Center for Brain Science, Harvard University, Cambridge, Massachusetts 02138, États-Unis MeSH keywords: Animaux, Encéphale, Protéines luminescentes, Souris, Protéines tissu nerveux, Neurones |
Notre conception actuelle du système nerveux a pris forme grâce à l’extraordinaire travail de Santiago Ramón y Cajal (1852-1934). Cajal a largement fondé son travail sur la technique de coloration de Golgi, qui marque aléatoirement un petit nombre de neurones avec un précipité noir, et permet de distinguer leurs prolongements (axones et dendrites), autrement invisibles dans l’enchevêtrement du tissu nerveux. Grâce à ce marquage, il put décrire les interconnexions entre les différents types de neurones et mettre en évidence les principes d’organisation des circuits neuronaux. La panoplie des techniques de marquage neuronal s’est aujourd’hui considérablement élargie. En particulier, l’avènement de la GFP (green fluorescent protein) a insufflé un formidable renouveau technique en permettant de créer un marquage vital endogène. Une étape décisive a été la génération de souris transgéniques exprimant la GFP à un fort niveau sous le contrôle d’un promoteur neuronal [
1]. Dans certaines lignées de souris, l’expression restreinte à un petit nombre de neurones fournit l’inappréciable équivalent d’une « coloration de Golgi vitale ». |
Une palette de protéines fluorescentes La biologie moléculaire offre maintenant aux scientifiques un large éventail de protéines fluorescentes de différentes couleurs : cyan (CFP), jaune (YFP), rouge (RFP)… [
2]. En croisant deux lignées de souris exprimant des « XFP » différentes dans des fractions de neurones, on peut obtenir des souris « bicolores » permettant d’étudier les interactions entre deux neurones [
3]. D’autres stratégies permettent de combiner deux XFP dans un même animal [
4] ; cependant, deux couleurs sont insuffisantes pour visualiser en les distinguant les multiples neurones composant un circuit. En principe, des combinaisons de trois ou quatre XFP exprimées à différents niveaux pourraient aboutir à la production d’un grand nombre de teintes différentes, de la même manière que l’écran d’un téléviseur combine les trois couleurs primaires rouge, vert et bleu pour former l’espace des couleurs. Ces combinaisons de couleurs pourraient servir à distinguer des neurones adjacents dans un circuit donné. Mais comment exprimer différentiellement plusieurs XFP dans des neurones adjacents qui appartiennent au même sous-type et ne présentent donc a priori pas de différences moléculaires ? |
Un tirage au sort moléculaire Dans un premier temps, nous avons cherché à créer une sorte de « dé moléculaire » permettant à chaque cellule d’effectuer indépendamment un choix aléatoire d’expression entre plusieurs XFP. Pour créer une telle expression stochastique, nous avons tiré parti d’un outil moléculaire très employé : le système de recombinaison Cre/lox [
5]. La recombinase Cre reconnaît spécifiquement une séquence de 34 paires de bases appelée loxP. Cre catalyse l’excision d’un segment d’ADN limité par deux séquences loxP de même orientation, ou son inversion lorsque ces séquences loxP sont dans des orientations opposées1
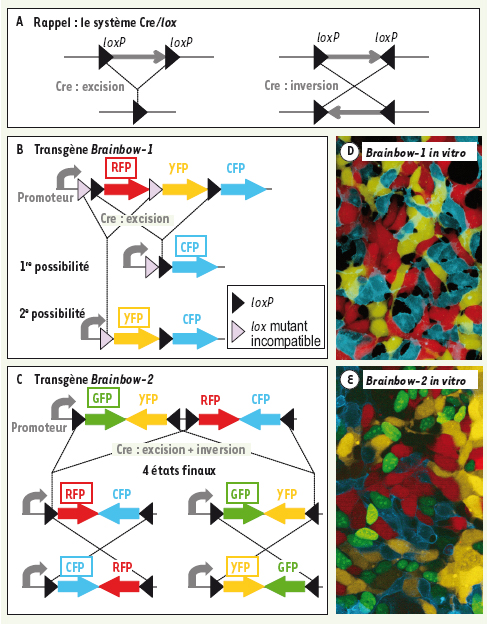
(Figure 1A).  | Figure 1.
Les stratégies d’expression aléatoire « Brainbow ». A. Rappel sur le système de recombinaison Cre/ lox : Cre catalyse la recombinaison entre deux sites loxP. En fonction de l’orientation des sites loxP, on obtient l’excision ou l’inversion du segment d’ADN situé entre ces derniers [ 5]. B. Dans le transgène Brainbow-1, deux paires de sites lox incompatibles entre eux sont alternées de manière à créer deux possibilités de recombinaison. Avant recombinaison, le transgène exprime RFP (gène encadré). Deux recombinaisons mutuellement exclusives sont possibles (entre sites lox canoniques ou entre sites lox mutants, mais pas entre un site lox canonique et un site lox mutant) et conduisent respectivement à l’expression de CFP ou YFP. C. Dans le transgène Brainbow-2, deux segments capables d’inversion, limités par des sites loxP d’orientation opposée, sont placés en tandem, créant de multiples possibilités de recombinaison. Les événement d’excision réduisent la construction à une unité inversible simple, qui conduit à deux possibilités d’expression selon son orientation finale. D, E. Expression des transgènes Brainbow dans des cellules HEK-293 en culture. La stratégie Brainbow-1 permet l’expression stochastique « mosaïque » de trois protéines fluorescentes. La recombinaison conduit à l’expression de CFP ou YFP, tandis que RFP reste exprimée dans les cellules dont le transgène n’a subi aucune recombinaison. La stratégie Brainbow-2 permet d’obtenir l’expression aléatoire de quatre XFP. |
Nous avons conçu plusieurs configurations de transgènes que nous appelons « Brainbow », utilisant le système Cre/lox pour créer un choix stochastique d’expression [
6]. Dans chacun de ces transgènes, un choix initial entre plusieurs possibilités de recombinaisons conduit à différentes configurations finales (les différentes facettes du dé moléculaire) dont chacune entraîne l’expression d’un gène distinct. Un premier type de transgène (Brainbow-1) utilise des sites lox variants, qui peuvent recombiner entre eux mais portent une mutation les rendant incompatibles vis-à-vis des sites lox « canoniques » (sites loxP) [5]. Nous avons placé ces sites lox canoniques et variants en configuration alternée pour créer deux possibilités d’excision mutuellement exclusives (Figure 1B). Dans une deuxième configuration de transgène (Brainbow-2), nous avons utilisé l’inversion de segments d’ADN limités par des sites loxP d’orientations opposées. L’inversion d’un tel segment persiste tant que la recombinase Cre est présente, mais s’arrête dans une orientation aléatoire lorsque Cre cesse d’agir, offrant deux possibilités d’expression [
7]. Nous avons réalisé qu’en positionnant en tandem deux de ces segments susceptibles d’inversion, le nombre d’alternatives serait encore accru : plusieurs possibilités d’inversion et excisions apparaissent dans cette configuration ; lorsqu’un événement d’excision se produit, le transgène est réduit à une unité simple ayant deux possibilités d’orientation, ce qui, on l’a vu, crée deux possibilités d’expression. La construction Brainbow-2 contenant deux sites capables d’inversion, l’on obtient au final quatre possibilités d’expression (Figure 1C).
Nous avons construit des transgènes Brainbow utilisant les deux stratégies décrites ci-dessus pour exprimer plusieurs XFP. En culture cellulaire, ces transgènes fonctionnent comme prévu : sous l’action de la recombinase Cre, chaque cellule effectue indépendamment un choix aléatoire de recombinaison, conduisant à l’expression d’une des XFP du transgène (Figure 1D, E). |
Expression combinatoire des XFP : des cerveaux multicolores Nous avons ensuite produit des souris transgéniques exprimant les constructions Brainbow sous le contrôle du promoteur neuronal Thy1 [1]. Pour déclencher la recombinaison, nous avons croisé les souris Brainbow avec des animaux exprimant la recombinase inductible CreER, activable par le ligand Tamoxifène [
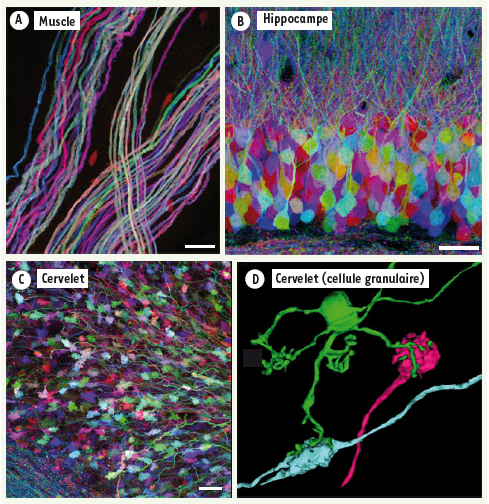
8]. Nous avons obtenu des souris présentant un marquage neuronal multicolore « mosaïque », indiquant que la recombinaison s’effectue de manière indépendante dans chaque cellule. Qui plus est, dans de nombreuses lignées de souris Brainbow, nous avons observé que les neurones n’expriment pas une XFP unique, mais des combinaisons de protéines fluorescentes (Figure 2A-C). Cette expression combinatoire était attendue : comme nous avons pu le montrer, elle résulte de l’intégration de plusieurs copies du transgène Brainbow dans le génome murin, couramment obtenue lors du processus de transgenèse. La présence de ces copies multiples conduit à une répétition du choix aléatoire d’expression dans une même cellule (le dé est lancé plusieurs fois). Plusieurs protéines fluorescentes sont ainsi co-exprimées à des niveaux divers par les neurones. La superposition des signaux issus de ces XFP crée de multiples couleurs, près d’une centaine dans les cerveaux examinés. Les différences de couleurs entre neurones permettent de les distinguer les uns des autres. Nous avons ainsi pu suivre et tracer plus de 300 axones dans une seule série d’images confocales du cervelet et visualiser certaines de leurs interactions synaptiques. Les couleurs permettent notamment de distinguer les neurones qui convergent sur une même cellule post-synaptique (Figure 2D).  | Figure 2.
Expression combinatoire des protéines fluorescentes dans les souris Brainbow. Plusieurs lignées de souris Brainbow présentent une expression combinatoire de protéines fluorescentes après l’intégration de multiples copies du transgène. Dans le système nerveux central et périphérique, le marquage multicolore obtenu permet de distinguer les neurones les uns des autres. A. Nerf moteur à la surface d’un muscle squelettique (Image confocale : Ryan W. Draft). B. Hippocampe, neurones du gyrus denté. C. Axones (« fibres moussues ») et synapses (élargissements appelés « rosettes ») dans la couche granulaire du cervelet. D. Reconstruction tridimensionnelle d’un neurone granulaire du cervelet à partir d’images confocales. Le marquage multicolore confère une identité aux axones (fibres moussues) qui contactent ce neurone granulaire : étant de couleurs distinctes, ces axones proviennent de neurones présynaptiques différents (reconstruction : Tamily A. Weissman) (barres d’échelle : 20 µm). |
|
Perspectives : vers le déchiffrage du « connectome » Aujourd’hui émerge l’idée ambitieuse de cartographier les circuits neuronaux de manière exhaustive, en reconstruisant un grand nombre, voire la totalité de leurs composants cellulaires, comme cela a été fait avec le nématode C. elegans [
9,
10]. Le déchiffrage du « connectome » représente potentiellement pour les neurosciences une révolution similaire à celle qu’a apporté la génomique en biologie moléculaire. Dans les approches actuellement développées, l’anatomie des neurones est suivie sur des séries d’images (coupes successives) obtenues en microscopie électronique [9], la moindre erreur survenant au cours de ce traçage itératif pouvant avoir des répercussions sur l’ensemble de la reconstruction. L’emploi du marquage multicolore Brainbow apparaît comme une alternative plus sûre, la couleur particulière de chaque neurone permettant de vérifier son identité à tout endroit du tracé. La limite de résolution imposée par la diffraction de la lumière restreint pour l’instant le champ d’application de cette approche. Les techniques émergentes de microscopie hyper-résolutive, ou la corrélation de l’imagerie optique et électronique pourraient permettre de s’affranchir de ces contraintes. Par ailleurs, l’outil Brainbow devrait être utile pour de multiples autres applications : bien que conçue pour l’observation des neurones, cette technique pourrait aisément être transposée à d’autres types cellulaires pour des applications telles que le traçage de lignages cellulaires, la visualisation des interactions entre cellules, ou pour créer des perturbations génétiques « mosaïques ». |
Ce travail a été effectué dans le laboratoire de Jeff W. Lichtman à Harvard University, en collaboration avec Joshua R. Sanes. Tamily A. Weissman, Hyuno Kang, Ryan Draft, Ju Lu et Robyn Bennis ont aussi contribué à cette étude financée par the James S. McDonnell foundation et NIH/NINDS. Merci à Laure Coulombel et Isabelle Livet pour leur relecture constructive de cet article.
|
Footnotes |
1. Feng G, Mellor RH, Bernstein M, et al. Imaging neuronal subsets in transgenic mice expressing multiple spectral variants of GFP. Neuron 2000; 28 : 41–51. 2. Shaner NC, Steinbach PA, Tsien RY. A guide to choosing fluorescent proteins. Nat Methods 2005; 2 : 905–9. 3. Kasthuri N, Lichtman JW. The role of neuronal identity in synaptic competition. Nature 2003; 424 : 426–30. 4. Zong H, Espinosa JS, Su HH, et al. Mosaic analysis with double markers in mice. Cell 2005; 121 : 479–92. 5. Branda CS, Dymecki SM. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell 2004; 6 : 7–28. 6. Livet J, Weissman TA, Kang H et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature 2007; 450 : 56–62. 7. Lam KP, Rajewsky K. Rapid elimination of mature autoreactive B cells demonstrated by Cre-induced change in B cell antigen receptor specificity in vivo. Proc Natl Acad Sci USA 1998; 95 : 13171–5. 8. Metzger D, Clifford J, Chiba H, Chambon P. Conditional site-specific recombination in mammalian cells using a ligand-dependent chimeric Cre recombinase. Proc Natl Acad Sci USA 1995; 92 : 6991–5. 9. Briggman KL, Denk W. Towards neural circuit reconstruction with volume electron microscopy techniques. Curr Opin Neurobiol 2006; 16 : 562–70. 10. Kasthuri N, Lichtman JW. The rise of the « projectome ». Nat Methods 2007; 4 : 307–8. |