| |
| Med Sci (Paris). 2007 March; 23(3): 273–278. Published online 2007 March 15. doi: 10.1051/medsci/2007233273.Des agents thérapeutiques ciblant des interactions protéine-protéine Mythe ou réalité ? Béatrice Laudet, Renaud Prudent, Odile Filhol, and Claude Cochet* Inserm U873, Institut de Recherches en Technologies et Sciences pour le Vivant,CEA Grenoble,17, rue des Martyrs,38054 Grenoble Cedex 9, France |
Dans un organisme vivant, les cellules sont régulièrement exposées à une variété de signaux à partir de leur micro-environnement. Nombre de ces signaux sont détectés par des récepteurs présents à la surface des cellules, puis amplifiés et « transduits » par des réseaux complexes de signalisation intracellulaire. L’organisation spatiale et l’activation de ces réseaux dépendent de façon cruciale d’interactions spécifiques entre les différentes protéines qui les composent. Les processus d’apoptose, notamment, mettent en jeu des réseaux complexes d’interaction protéine-protéine qui dictent la survie ou la mort cellulaire ; les interactions protéine-protéine sont donc fondamentales pour la régulation des points de contrôle de l’homéostasie cellulaire. Elles constituent un réseau dynamique et adaptable, l’« interactome », capable d’intégrer des signaux intra- comme extracellulaires et de modifier le comportement cellulaire. |
Propriétés des interactions protéine-protéine Les interactions protéine-protéine mises en jeu dans les complexes enzyme-substrat ne seront pas décrites dans cet article, consacré aux propriétés des interactions « non catalytiques » qui conduisent à des homo- ou hétéro-oligomérisations de protéines. Différentes approches combinées de biologie structurale permettent de comprendre comment fonctionnent les interfaces protéine-protéine au sein de ces complexes moléculaires. Bien que les principes structuraux et fonctionnels qui gouvernent ces interactions soient d’une grande diversité, les interfaces protéine-protéine sont, dans de nombreux cas, larges (typiquement 1 300 à 3 000 Å2), et leurs surfaces compactes, hydrophobes et relativement planes [
1,
2]. La combinaison des analyses cristallographiques et de mutagenèse dirigée a révélé la présence, au niveau de ces interfaces, d’acides aminés essentiels pour l’interaction (hot spots). La résolution de la structure tridimensionnelle de différents complexes protéiques a permis de mettre en évidence l’existence de régions centralisées et accessibles, constituées d’acides aminés cruciaux pour l’interaction des protéines composant ces complexes [
3]. Ces hot spots sont présents sur les deux côtés de l’interface protéine-protéine. Les structures cristallographiques montrent que ces régions sont hautement complémentaires l’une de l’autre, avec des résidus hydrophobes d’une surface s’engageant précisément dans des « puits » présents sur la face opposée. Il est remarquable de constater que dans le cas du complexe p53-Mdm2 (double-minute protein 2), la mutation de seulement 3 des 393 acides aminés de p53 suffit à empêcher la formation du complexe [
4]. De nombreuses études suggèrent que la présence de ces hot spots confère à ces régions les propriétés nécessaires à la liaison de ligands [
5]. L’apparente complémentarité entre les deux surfaces créée par ces hot spots se traduit par un certain degré de flexibilité et d’adaptabilité, qui permet la fixation séquentielle, sur une même protéine, de multiples partenaires [
6]. Les interactions protéine-protéine peuvent être définies sur la base de la durée de vie des complexes qui sont soit très stables et permanents, soit transitoires, s’associant ou se dissociant constamment in vivo. Il existe également un type d’associations fortes, mais néanmoins transitoires, qui requièrent un signal moléculaire pour altérer l’affinité mutuelle des composants du complexe. La protéine kinase AMPc-dépendante (PKA) est un exemple de ce type de complexe, où la fixation de l’AMP cyclique sur la sous-unité régulatrice (R) de la kinase rompt son interaction avec la sous-unité catalytique (C) [
7]. Il faut souligner ici que la plupart des complexes transitoires ont des rôles régulateurs importants [6], le corollaire en étant que les mécanismes contrôlant leur formation sont vitaux pour les fonctions biologiques de ces complexes. |
Inhiber ou rompre des interactions protéine-protéine Le dysfonctionnement d’interactions protéine-protéine hautement spécifiques, lié notamment à des mutations, est à l’origine de différentes pathologies, dont le cancer. La perturbation de ces complexes protéiques a également des effets physiologiques majeurs. La possibilité d’interférer avec les interactions responsables de la formation de ces complexes devrait donc permettre d’exercer un contrôle unique sur des événements cellulaires majeurs, à l’origine de diverses pathologies, et permettre de développer de nouveaux agents thérapeutiques. Les inhibiteurs d’interactions protéine-protéine sont susceptibles de présenter plusieurs avantages par rapport à d’autres types d’inhibiteurs. D’une part, l’extrême complémentarité des interfaces protéine-protéine offre la possibilité de développer des inhibiteurs hautement spécifiques, évitant ainsi l’inhibition de cibles cellulaires non spécifiques (off-target). D’autre part, les complexes protéiques ciblés par ces inhibiteurs devraient être moins sujets à des mutations leur conférant une résistance, contrairement aux protéines qui fixent des inhibiteurs au niveau de leur site actif : de fait, une simple mutation sur une sous-unité au niveau de l’interface nécessite une mutation complémentaire sur l’autre sous-unité pour restaurer l’interaction ; comme une double mutation simultanée sur des sous-unités différentes n’est guère possible, il est hautement improbable que la protéine ciblée acquière une résistance à l’inhibiteur. Toutefois, il faut admettre que ces avantages ont été pendant longtemps tempérés par le fait que, contrairement à la démarche classique qui repose sur l’inhibition d’une activité enzymatique, le ciblage de ce type d’interaction par des composés chimiques conventionnels semblait particulièrement difficile. Aujourd’hui, les développements spectaculaires de la génomique et de la protéomique, ainsi que l’émergence de nouvelles cibles thérapeutiques impliquant des interactions protéine-protéine, ont récemment stimulé, avec succès, une recherche orientée vers le ciblage de ce type d’interaction. |
Les propriétés intrinsèques des interfaces protéiques, flexibilité et capacité à adopter des conformations multiples, sont mises à contribution pour le développement de petites molécules pharmacologiques capables d’interagir avec ces interfaces protéiques. Ce développement requiert au préalable une connaissance approfondie du réseau dans lequel elles s’inscrivent, afin de s’assurer de la pertinence de l’interaction ciblée : il s’agit notamment de s’assurer de l’absence de voie alternative, qui rend cette interaction cruciale pour la transmission du signal. Dans le cas des tumeurs surexprimant Mdm2, par exemple, on choisira une interaction nécessaire pour la survie de cellules cancéreuses, mais facultative pour les cellules normales [
8]. Diverses méthodes [
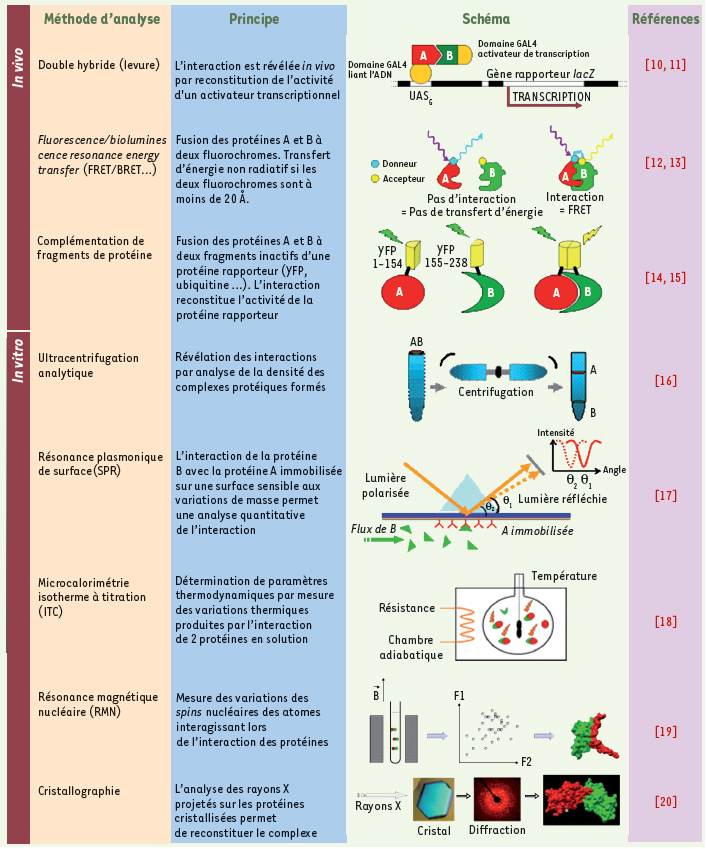
9] peuvent être mises en œuvre pour l’analyse des interactions protéine-protéine (Figure 1). La co-cristallisation d’un complexe protéique ou, à défaut, une étude de complémentarité in silico permettent de caractériser l’interface ciblée et le type d’interaction mis en jeu. Ainsi, une surface d’interaction inférieure à 900 Å2, signature probable d’un complexe transitoire, est une configuration favorable pour le développement d’inhibiteurs. Une étude détaillée de la structure permet également d’identifier les acides aminés contribuant de manière prépondérante à l’interaction ; la mutation (ala-scanning) de ces acides aminés d’intérêt, puis l’analyse des mutants par des approches structurales, thermodynamiques ou biophysiques telles que celles qui sont décrites dans la Figure 1 valident ces hot spots potentiels.  | Figure 1.
Principales méthodes utilisées pour l’identification et l’analyse des interactions protéine-protéine. YFP : yellow fluorescent protein. |
Une fois l’interface caractérisée, la recherche de molécules inhibitrices peut s’effectuer par trois approches : le criblage réel, le criblage virtuel ou la conception de novo. Criblage réel Il consiste à tester systématiquement des banques de molécules, ou chimiothèques, à l’aide d’un test d’interaction simple, rapide, robuste, reproductible, miniaturisé et automatisé. In vitro, ce test fait appel à des techniques de type ELISA ou à la polarisation de fluorescence ; le criblage peut également reposer sur un test cellulaire, dans lequel l’interaction protéine-protéine peut notamment être évaluée par la technique de double hybride ou le FRET (Figure 1). Il est important de noter que l’utilisation de banques de composés fonctionnellement très diverses (petites molécules chimiques, peptides, voire peptidomimétiques) semble essentielle pour la découverte de molécules ayant ce type de propriétés uniques. Une technique récente d’identification d’inhibiteurs des interactions protéine-protéine repose sur l’utilisation de mélanges de très petites molécules (fragments) : si ces molécules possèdent isolément une faible affinité pour une cible, leur association en présence de la protéine augmente considérablement leurs propriétés inhibitrices [
21]. Criblage virtuel La deuxième stratégie s’appuie sur les développements récents de la bio-informatique. Elle consiste à tester in silico la complémentarité de forme et l’interaction probable entre une molécule et la structure cristallographique de la protéine, ce que l’on appelle le docking. On peut ainsi tester plusieurs millions de molécules référencées dans diverses chimiothèques en quelques heures. L’objectif initial est souvent de créer une sous-chimiothèque enrichie en « touches » potentiellement positives, afin d’accroître le taux de réussite lors des tests réels. Conception de novo
Une approche encore plus audacieuse est la conception de novo, qui consiste à construire rationnellement une molécule à partir des seules données structurales du complexe. L’enjeu, particulièrement difficile, est de synthétiser ensuite cette molécule « idéale » inhibitrice de la formation du complexe. Cependant, l’enthousiasme initial suscité par cette dernière approche doit être tempéré [
22] : en effet, les algorithmes actuels ne prennent, pour l’instant, pas ou peu en compte des phénomènes primordiaux tels que la flexibilité conformationnelle des protéines ou l’influence du solvant et des ions dans l’interaction protéine-ligand. Évaluation des composés Dans tous les cas, les molécules pré-sélectionnées après criblage doivent être validées en culture cellulaire, et le phénotype attendu résultant de l’exposition à ces molécules doit être le même que celui observé avec des mutants d’interaction. Dans ce contexte, les inhibiteurs sont soumis à diverses contraintes cellulaires. Ils doivent notamment être capables d’accéder aux compartiments cellulaires où se trouvent leurs cibles. Par ailleurs, l’influence des modifications post-traductionnelles des protéines sur leurs interactions est une propriété dont on peut tirer parti. Il est ainsi possible de développer des inhibiteurs d’interaction de protéines de surface, fondés sur des polysaccharides agissant comme leurres glycosidiques : l’interaction protéine-protéine est dans ce cas rompue par compétition avec la partie glycosylée [
23]. L’ubiquitinylation est un autre type de modification post-traductionnelle exploitable ; à titre d’exemple, des composés nommés ubistatines [
24] sont capables, en se fixant sur les polymères d’ubiquitine, de rompre l’interaction ubiquitine-protéasome. Enfin, dans le cadre d’essais précliniques, les propriétés pharmacologiques et thérapeutiques des composés retenus sont évaluées dans des modèles animaux appropriés à la pathologie considérée. |
Des résultats significatifs ont d’ores et déjà été obtenus par cette orientation thérapeutique, dans des domaines aussi variés que l’inflammation (Jak/Stat) [
25], la prolifération cellulaire (anti-IGFR) [
26] et la virologie (protéase du VIH) [
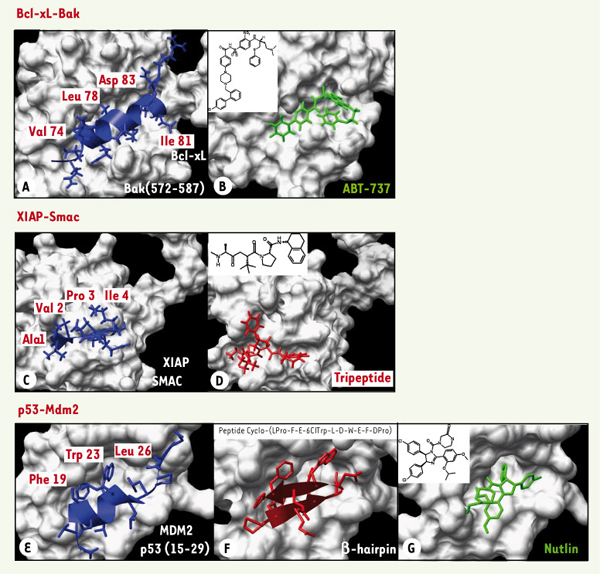
27]. Toutefois, cette revue se limitera à une brève description de nouveaux agents anticancéreux ciblant des interactions protéine-protéine opérant dans les processus d’apoptose (Figure 2).  | Figure 2.
Cibler des interactions protéine-protéine : le mythe devient réalité. A, B. Interaction Bcl-xL-Bak. A. Quatre résidus de Bak représentent des hot spots pour son interaction avec Bcl-xL (pdb 1BXL, [
29]). B. Une molécule chimique (ABT-737) inhibe l’interaction Bcl-xL-Bak (IC 50 = 1 nM, pdb 1YSI, [
31]) en mimant ces résidus critiques pour l’interaction. C, D. Interaction XIAP-Smac. C. La protéine Smac interagit avec XIAP via 4 résidus essentiels aminoterminaux, Ala1, Val2, Pro3, Ile4 (pdb 1G3F, [
35]). D. Un tripeptide non naturel optimisé inhibe l’interaction avec une IC 50 de 5 nM (pdb 1TFT, [
39]). E, F, G. Interaction p53-Mdm2. E. La liaison du domaine de transactivation de p53 à la protéine Mdm2 (pdb 1YCR, [ 4]) fait intervenir principalement 3 résidus hot spots (Phe19, Trp23, Leu26). F. Un peptide mimétique contraint occupe la même poche de Mdm2 (pdb 2AXI, [
40]) et inhibe l’interaction avec une IC 50 de 140 nM. G. Des petites molécules chimiques, les nutlins, miment les hot spots de p53 et inhibent l’interaction avec une IC 50 de 100 nM (pdb 1RVI [ 8]). Les images graphiques ont été produites grâce au logiciel UCSF Chimera package [
41]. |
Interaction Bcl2-Bax La découverte de molécules capables de sensibiliser des cellules résistantes à l’apoptose est une stratégie attractive pour le développement de nouveaux agents anticancéreux [
28]. Certains membres de la famille des protéines anti-apoptotiques telles que Bcl-x L et Bcl-2 interagissent avec des protéines pro-apoptotiques appartenant à la même famille, telles que Bak ou Bax. Des études en RMN ont révélé que ces interactions reposent sur un domaine BH3 ( Bcl-2 homology domain 3) présent dans Bax. Un peptide de 16 acides aminés ( BH3 peptide) dérivé du domaine BH3 de Bax forme une hélice α qui interagit avec une poche hydrophobe de Bcl-x L [ 29] et inhibe l’interaction Bcl-x L-Bax ( Figure 2A). Le criblage d’une banque de 16 320 petites molécules chimiques a, par la suite, permis d’identifier deux composés capables d’inhiber la liaison du peptide BH3 à Bcl-2. L’un de ces composés, ABT-737, bloque l’hétérodimérisation entre Bax et Bcl-2 (Figure 2B) ; il induit l’apoptose de cellules transformées et sensibilise les cellules tumorales surexprimant Bcl-2 aux chimiothérapies [
30]. Par ailleurs, d’autres composés chimiques ciblant cette interaction provoquent la régression de tumeurs du poumon dans un modèle de xénogreffe chez la souris [ 31]. Interaction Smac-XIAP Les protéines de la famille des IAP ( inhibitors of apoptosis proteins) sont des inhibiteurs endogènes des caspases ; elles sont surexprimées dans de nombreux cancers, où elles contribuent aux mécanismes de résistance à l’apoptose observée dans les cellules cancéreuses [
32]. Les IAP possèdent un domaine à doigt de zinc, appelé Bir ( Baculovirus inhibitory repeat), qui est le site d’interaction avec Smac ( second mitochondria-derived activator of caspases), une protéine mitochondriale activatrice des caspases au cours de l’apoptose [
33]. Un tripeptide non naturel dérivé de Smac inhibe son interaction avec XIAP ( X-linked mammalian inhibitor of apoptosis protein) (Figure 2D). Ce type de peptides sensibilisent des cellules tumorales à l’apoptose induite par le facteur apoptotique TRAIL ( TNF-related apoptosis-inducing ligand) et stimulent l’activité antitumorale de ce facteur dans un modèle de xénogreffe intracraniale de gliome [
34]. Des études cristallographiques ont montré que seuls quatre acides aminés de Smac se fixent sur le domaine Bir de XIAP (Figure 2C) et sont essentiels pour bloquer ses effets anti-apoptotiques [ 35]. Là encore, la conformation de ces quatre acides aminés au sein du complexe IAP-Smac a servi de support pour la synthèse rationnelle d’une molécule chimique capable, en se fixant avec une haute affinité à IAP, de promouvoir l’activation des caspases, en synergie avec le TNFα et TRAIL, et d’induire l’apoptose de cellules cancéreuses humaines. Enfin, en raison de son interférence avec la signalisation TNFα-NFκβ, ce « mime chimique » de Smac pourrait conduire au développement d’agents thérapeutiques dans le cadre des pathologies de l’inflammation [
36]. Interaction p53-Mdm2 La protéine p53 est une cible thérapeutique attractive en oncologie en raison de sa capacité à induire la mort cellulaire par apoptose, et donc de permettre l’éradication des cellules cancéreuses. Toutefois, l’activité de p53 est réprimée par un régulateur négatif, la protéine Mdm2, dont l’amplification dans de nombreux cancers favorise une prolifération incontrôlée. L’inhibition de l’interaction p53-Mdm2 dans ces cancers représente donc une stratégie intéressante pour activer une apoptose p53-dépendante dans les tumeurs surexprimant Mdm2 [
37]. Intervenir sur le complexe p53-Mdm2 est apparu possible lorsqu’il a été montré que de petits peptides correspondant à l’extrémité aminoterminale de p53, en se fixant sur l’extrémité aminoterminale de Mdm2, bloquent l’interaction p53-Mdm2 dans des cellules tumorales et induisent leur apoptose [ 37]. La structure de Mdm2 complexée à ce peptide (Figure 2F) a révélé que les chaînes latérales de trois acides aminés de p53 s’engagent dans une cavité hydrophobe de la molécule Mdm2 (Figure 2E), ce qui est suffisant pour former le complexe p53-Mdm2 [ 4]. Ces observations ont rapidement stimulé la recherche de composés chimiques de faible poids moléculaire, capables de bloquer cette interaction. Le criblage d’une banque de molécules chimiques a alors permis d’isoler une classe de composés appelés Nutlins (Figure 2G), qui déplacent p53 de son complexe avec Mdm2 avec des valeurs de IC 50 de l’ordre de 100 nM à 300 nM [ 8]. Comme attendu, les Nutlins provoquent l’activation de p53 et l’apoptose de cellules cancéreuses. Plus spectaculaire encore, l’administration orale de Nutlin pendant 3 semaines à des souris nude porteuses de xénogreffes de cellules d’ostéosarcome conduit à un arrêt de la croissance des tumeurs [ 8]. |
Conclusions et perspectives En conclusion, l’identification de petites molécules capables de moduler des interactions protéine-protéine est restée pendant longtemps un domaine largement sous-exploité par la recherche pharmacologique. Cependant, le développement de différentes technologies innovantes et la validation récente de ce type d’inhibiteurs dans des essais précliniques, comme c’est le cas de la colchicine, inhibiteur de l’interaction tubuline α-tubuline β [
38], démontrent que les interactions protéiques intracellulaires constituent une nouvelle source de cibles thérapeutiques importantes. Les molécules modulatrices de ces interactions représentent une nouvelle classe d’outils originale et prometteuse, tant en recherche fondamentale qu’en thérapeutique. Elles peuvent aider à différencier les multiples fonctions portées par une même protéine, à replacer la protéine dans une cascade de réactions, ainsi qu’à disséquer et reconstituer des réseaux de signalisations protéiques. Enfin, et surtout, on peut prédire sans risque que cette stratégie a de grandes chances de faire émerger de nouvelles familles d’agents pharmacologiques actifs dans diverses pathologies. |
Les travaux du groupe Structure et fonctions de la CK2 de l’unité U873 bénéficient du soutien financier de l’Inserm, du CEA et de la Ligue nationale contre le cancer (équipe labellisée EL2004.LNCC/CC2).
|
Footnotes |
1. Lo Conte L, Chothia C, Janin J. The atomic structure of protein-protein recognition sites. J Mol Biol 1999; 285 : 2177–98. 2. Nooren IMA, Thornton JM. Structural characterization and functional significance of transient protein-protein interactions. J Mol Biol 2003; 325 : 991–1018. 3. Bogan AA, Thorn KS. Anatomy of hot spots in protein interfaces. J Mol Biol 1998; 280 : 1–9. 4. Kussie PH, Gorina S, Marechal V, et al. Structure of the MDM2 oncoprotein bound to the p53 tumor suppressor transactivation domain. Science 1996; 274 : 948–53. 5. Teague SJ. Implication of protein flexibility for drug discovery. Nat Rev Drug Discov 2003; 2 : 527–41. 6. DeLano WL, Ultsch MH, de Vos AM, Wells JA. Convergent solutions to binding at a protein-protein interface. Science 2000; 287 : 1279–83. 7. Kim c, Xuong NH, Taylor SS. Crystal structure of a complex between the catalytic and regulatory (RIalpha) subunits of PKA. Science 2005; 307 : 690–6. 8. Vassilev LT, Vu BT, Graves B, et al.
In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science 2004; 303 : 844–8. 9. Piehler J. New methodologies for measuring protein interactions in vivo and in vitro. Curr Opin Struct Biol 2005; 15 : 4–14. 10. Toby GG, Golemis EA. Using the yeast interaction trap and other two-hybrid-based approaches to study protein-protein interactions. Methods 2001; 24 : 201–17. 11. Serebriiskii IG, Kotova E. Analysis of protein-protein interactions utilizing dual bait yeast two-hybrid system. Meth Mol Biol 2004; 261 : 263–96. 12. Trugnan G, Fontanges P, Delautier D, Ait-Slimane T. FRAP, FLIP, FRET, BRET, FLIM, PRIM… De nouvelles techniques pour voir la vie en couleur ! Med Sci (Paris) 2004; 20 : 1027–34. 13. Pfleger KD, Eidne KA. Illuminating insights into protein-protein interactions using bioluminescence resonance energy transfer (BRET). Nat Methods 2006; 3 : 165–74. 14. Thaminy S, Miller J, Stagljar I. The split-ubiquitin membrane-based yeast two-hybrid system. Meth Mol Biol 2004; 261 : 297–312. 15. Hu CD, Kerpolla TK. Simultaneous visualization of multiple protein interactions in living cells using multicolour fluorescence complementation analysis. Nat Biotechnol 2003; 21 : 539–45. 16. Lebowitz J, Lewis MS, Schuck P. Modern analytical ultracentrifugation in protein science: a tutorial review. Protein Sci 2002; 11 : 2067–79. 17. Karlsson R, Falt A. Experimental design for kinetic analysis of protein-protein interactions with surface plasmon resonance biosensors. J Immunol Methods 1997; 200 : 121–33. 18. Pierce MM, Raman CS, Nall BT. Isothermal titration calorimetry of protein-protein interactions. Methods 1999; 19 : 213–21. 19. Kay LE. NMR studies of protein structure and dynamics. J Magn Resonance 2005; 173 : 193–207. 20. Dauter Z. Current state and prospects of macromolecular crystallography. Acta Crystallographica D Biol Crystallogr 2006; 62 : 1–11. 21. Erlanson DA, McDowell RS, O’Brien T. Fragment-based drug discovery. J Med Chem 2004; 47 : 3463–82. 22. Wermuth CG. Les grandes méthodes de découverte sont-elles appropriées ? Biofutur 2003; 239 : 23–7. 23. Kitov PI, Sadowska JM, Mulvey G, et al. Shiga-like toxins are neutralized by tailored multivalent ligands. Nature 2000; 403 : 669–72. 24. Verma R, Peters NR, D’Onofrio M, et al. Ubistatins inhibit proteasome-dependent degradation by binding the ubiquitin chain. Science 2004; 306 : 117–20. 25. Luo C, Laaja P. Inhibitors of JAKs/STATs and the kinases: a possible new cluster of drugs. Drug Discov Today 2004; 9 : 268–75. 26. Garcia-Echeverria C, Pearson MA, Marti A, et al. In vivo antitumor activity of NVP-AEW541. A novel, potent, and selective inhibitor of the IGF-IR kinase. Cancer Cell 2004; 5 : 231–8. 27. Song M, Rajesh S, Hayashi Y, Kiso Y. Design and synthesis of new inhibitors of HIV-1 protease dimerization with conformationally constrained templates. Bioorg Med Chem Lett 2001; 11 : 2465–8. 28. Huang Z. Bcl-2 family proteins as targets for anticancer drug design. Oncogene 2000; 19 : 6627–31. 29. Sattler M, Liang H, Nettesheim D, et al. Structure of Bcl-xL-Bak peptide complex: recognition between regulators of apoptosis. Science 1997; 275 : 983–6. 30. Degterev A, Lugovskoy A, Cardone M, et al. Identification of small-molecule inhibitors of interaction between the BH3 domain and Bcl-xL. Nat Cell Biol 2001; 3 : 173–82. 31. Oltersdorf T, Elmore SW, Shoemaker AR, et al. An inhibitor of Bcl-2 family proteins induces regression of solid tumours. Nature 2005; 435 : 677–81. 32. Bilim V, Kasahara T, Hara N, et al. Role of XIAP in the malignant phenotype of transitional cell cancer (TCC) and therapeutic activity of XIAP antisense oligonucleotides against multidrug-resistant TCC in vitro. Int J Cancer 2003; 103 : 29–37. 33. Du C, Fang M, Li Y, et al. Smac, a mitochondrial protein that promotes cytochrome c-dependent caspase activation by eliminating IAP inhibition. Cell 2000; 102 : 33–42. 34. Fulda S, Wick W, Weller M, Debatin KM. Smac agonists sensitize for Apo2L/TRAIL- or anticancer drug-induced apoptosis and induce regression of malignant glioma in vivo. Nat Med 2002; 8 : 808–15. 35. Liu Z, Sun C, Olejniczak ET, et al. Structural basis for binding of Smac/Diablo to the XIAP BIR3 domain. Nature 2000; 408 : 1004–8. 36. Li L, Thomas RM, Suzuki H, et al. A small molecule Smac mimic potentiates TRAIL- and TNFalpha-mediated cell death. Science 2004; 305 : 1471–4. 37. Bottger A, Bottger V, Sparks A, et al. Design of a synthetic Mdm2-binding mini protein that activates the p53 response in vivo. Curr Biol 1997; 7 : 860–9. 38. Mukherjee AK, Basu S, Sarkar N, Ghosh AC. Advances in cancer therapy with plant based natural products. Curr Med Chem 2001; 8 : 1467–86. 39. Oost TK, Sun C, Armstrong RC, et al. Discovery of potent antagonists of the antiapoptotic protein XIAP for the treatment of cancer. J Med Chem 2004; 47 : 4417–26. 40. Fasan R, Dias RL, Moehle K, et al. Structure-activity studies in a family of beta-hairpin protein epitope mimetic inhibitors of the p53-HDM2 protein-protein interaction. Chembiochem 2006; 7 : 515–26. 41. Pettersen EF, Goddard TD, Huang CC, et al. UCSF Chimera. A visualization system for exploratory research and analysis. J Comput Chem 2004; 25 : 1605–12. |