| |
| Med Sci (Paris). 2007 May; 23(5): 519–525. Published online 2007 May 15. doi: 10.1051/medsci/2007235519.Adresser du matériel allogène dans le compartiment mitochondrial Un défi pour comprendre la physiologie mitochondriale et une perspective pour la thérapie Pierre Rustin,1* Howard T. Jacobs,2 André Dietrich,3 Robert N. Lightowlers,4 Ivan Tarassov,5 and Marisol Corral-Debrinski6 1Inserm U676, Hôpital Robert Debré et Université Paris 7, Faculté de Médecine Denis Diderot, IFR02, 75019 Paris, France 2Institute of Medical Technology and Tampere University Hospital, University of Tampere, FI-33014, Tampere, Finland 3Institut de Biologie Moléculaire des Plantes du CNRS, Université Louis Pasteur, 12, rue du Général Zimmer, 67084 Strasbourg Cedex, France 4Mitochondrial Research Group, School of Neurology, Neurology and Psychiatry, Medical School, University of Newcastle upon Tyne, Framlington Place, Newcastle upon Tyne, NH2 4HH, Royaume-Unis 5UMR 7156 « Génétique moléculaire, Génomique, Microbiologie », CNRS et Université Louis Pasteur, 21, rue René Descartes, 67084 Strasbourg, Cedex, France 6Laboratoire de Physiopathologie Cellulaire et Moléculaire de la Rétine, Inserm U592 et Université Pierre et Marie Curie (UPMC-Paris 6), Hôpital Saint Antoine, 75571 Paris Cedex 12, France |
Les maladies mitochondriales forment un groupe croissant d’affections multiformes aux causes génétiques multiples [
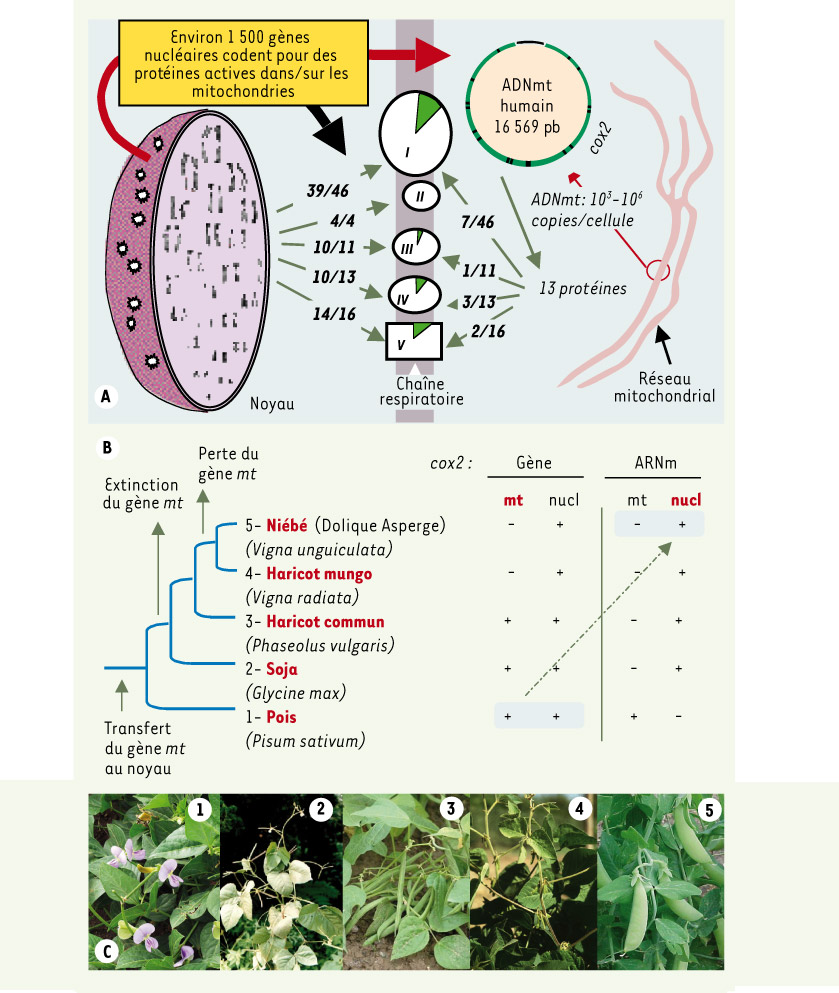
1]. Cela ne constitue pas véritablement une surprise, si l’on considère qu’outre les 37 gènes portés par le génome mitochondrial, plus de 1500gènes nucléaires sont nécessaires au bon fonctionnement mitochondrial (Figure 1A). Il est aventureux de chiffrer l’impact de ces maladies. De plus, il semble que le vieillissement des organismes aérobies (et les maladies qui chez l’homme lui sont associées) résulte en grande partie de perturbations des fonctions mitochondriales. On assiste de fait à la naissance d’une véritable « médecine mitochondriale » avec comme socle commun les perturbations de la synthèse d’ATP, de la manipulation de l’oxygène et des intermédiaires carbonés [1]. 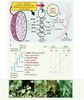 | Figure 1.
Les gènes codant pour les sous-unités des complexes de la chaîne respiratoire. A. La double origine nucléaire et mitochondriale de la centaine de protéines constituant la chaîne respiratoire participe à la complexité génétique des maladies mitochondriales. Un nombre considérable de protéines additionnelles, plus de 1500, sont en réalité nécessaires au bon fonctionnement des mitochondries. Les composants de la chaîne respiratoire synthétisés à partir de l’ADN mitochondrial sont indiqués en vert. B, C. Au cours de l’évolution, une bonne partie des gènes codant pour les constituants de la chaîne respiratoire a migré depuis l’ADN mitochondrial vers l’ADN nucléaire. C’est le cas du gène Cox2 qui dans la famille des Papilionacées est présent à l’état actif dans l’un ou l’autre des génomes. mt: mitochondrial ; nucl : nucléaire. |
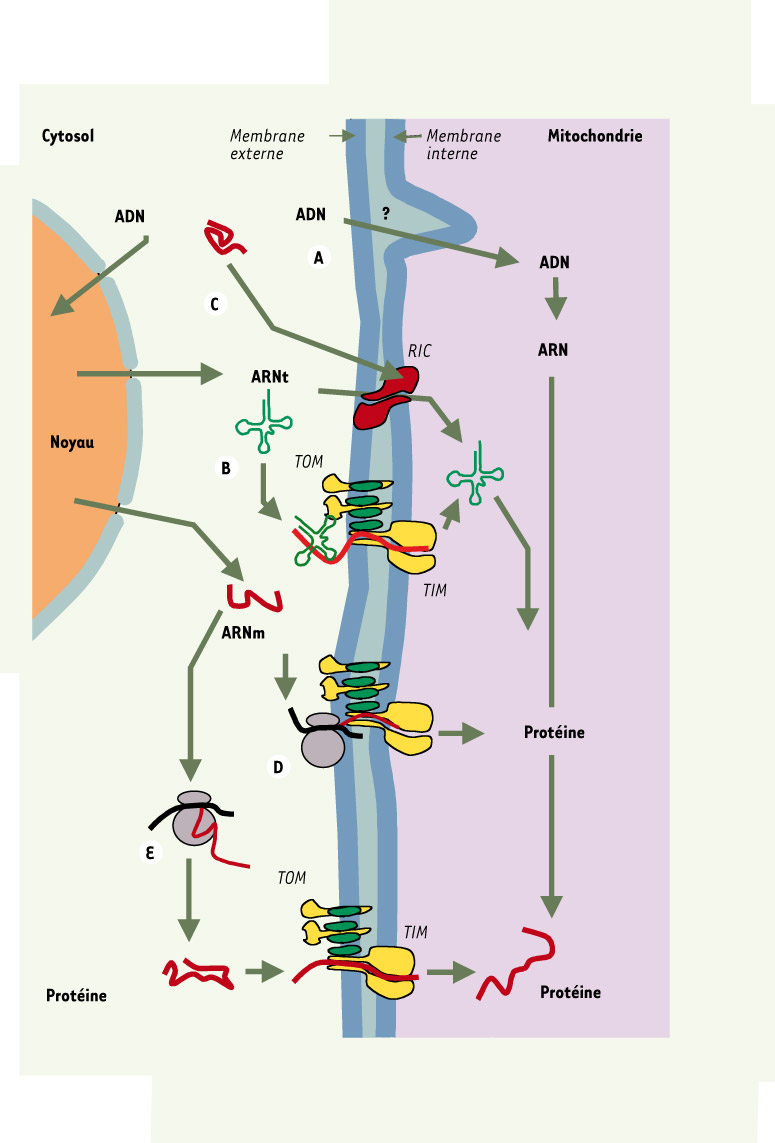
Face à ces maladies, les capacités d’interventions thérapeutiques restent quasi inexistantes. En effet, à quelques rares exceptions près, l’identification chez une partie des patients des gènes porteurs de mutations n’a malheureusement pas réellement fourni les clés espérées pour combattre ces maladies. Cette situation a conduit de nombreux groupes à travers le monde, en particulier en France, à imaginer des stratégies nouvelles pour tenter d’enrayer ces atteintes. La nouveauté des stratégies retenues implique une part de risque évidente et les obstacles avant d’envisager leur mise en œuvre chez l’homme sont encore nombreux. Parmi ces approches nouvelles figure l’idée d’importer dans les mitochondries du matériel exogène, ADN, ARN ou protéines ayant une activité curative (Figure 2). Comme on peut bien l’imaginer, importer chacun de ces types de molécules impose des contraintes propres. Des machineries très élaborées et maintenant bien caractérisées permettent le transport des protéines à travers les membranes mitochondriales (Figure 3) [
2]. En revanche, l’import de certains ARN dans les mitochondries humaines et sa signification physiologique ont été longtemps un sujet de controverse et les relations entre cet import et celui des protéines restent incertaines et probablement différentes d’une espèce à l’autre. Finalement, les mécanismes intervenant dans l’import d’ADN dans les mitochondries sont pour l’instant bien mystérieux. 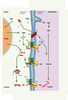 | Figure 2.
Les différents types de molécules allotopiques possiblement importées dans les mitochondries. De l’ADN nu mis au contact des mitochondries est susceptible d’y être importé par un mécanisme encore inconnu, puis d’y être utilisé pour synthétiser de l’ARN (A). Des ARN de transfert cytosoliques peuvent être importés dans les mitochondries, soit par l’intermédiaire du complexe TOM/TIM normalement responsable de l’import des protéines (B), soit par un système d’import spécifique RIC (RNA import complex) provenant d’un protozoaire (C). Un gène normalement porté par l’ADNmt peut-être recodé selon le code universel et l’ARNm correspondant traduit à la surface des mitochondries pour assurer un import efficace de la protéine synthétisée (D). Une protéine d’intérêt, normalement absente chez l’homme, peut être exprimée dans le but de complémenter fonctionnellement un déficit mitochondrial (E). TOM: translocase of the outer membrane ; TIM: translocase of the inner membrane.
|
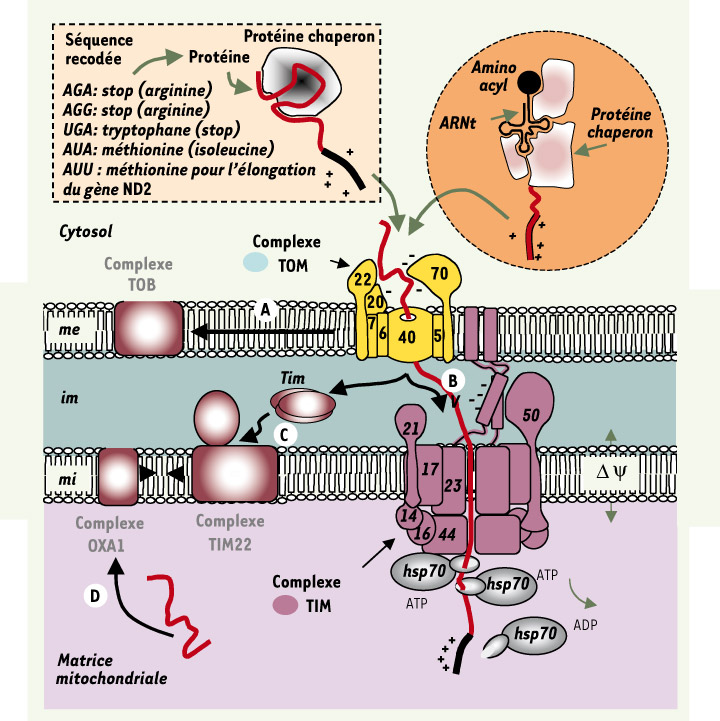
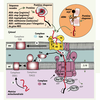 | Figure 3.
Les complexes membranaires permettant l’import et la mise en place des protéines dans les mitochondries. On distinguera les deux membranes mitochondriales, externe (me) et interne (mi). La première porte le système TOM (translocase of the outer membrane) qui reconnaît les séquences d’adressage des protéines destinées à être importées dans la matrice mitochondriale. Les protéines dites β-barrel sont distribuées dans la membrane externe par le système TOB (topogenesis of mitochondrial outer-membrane β-barrel proteins) (A). À cheval sur les deux membranes grâce à sa sous-unité 23, le complexe TIM (translocase of the inner membrane) assure, sous la dépendance du potentiel de membrane (∆Ψ) et en liaison avec les protéines HSP70 agissant elles grâce à l’hydrolyse de l’ATP, l’import des polypeptides depuis l’espace inter-membranaire (im) jusqu’à l’espace matriciel (B). Spécifiquement impliqué dans la mise en place de protéines du type du transporteur des adénylates, TIM22 (C) apparaît distinct du complexe TIM. Finalement, le complexe OXA1 permet la redistribution à la membrane interne de protéines synthétisées dans la matrice mitochondriale (D). Le système TOM/TIM permet l’import de protéines allotopiques synthétisées dans le noyau à partir de gènes mitochondriaux recodés pour tenir compte des 5 différences existant entre le code génétique mitochondrial et le code universel (en haut à gauche ; indiqué entre parenthèses). Le même système est impliqué dans l’import d’ARN de transfert cytosolique grâce à une reconnaissance de protéines chaperons liant l’ARNt (en haut à droite). |
|
Importer de l’ADN dans les mitochondries
À priori l’idée d’importer de l’ADN dans les mitochondries semble folle, un tel phénomène n’ayant jamais été observé à ce jour in vivo. Il existe cependant un échange directionnel reconnu de matériel génétique depuis l’ADN mitochondrial (ADNmt) vers le noyau. Durant l’évolution, une grande partie des gènes initialement portés par l’ADNmt se sont retrouvés dans l’ADN nucléaire, ce transfert s’accompagnant de nombreuses insertions de pseudo-gènes. Ces gènes peuvent, selon les espèces, être ou non présents dans les deux génomes, mitochondrial et nucléaire. Une illustration de ce transfert de certains gènes entre mitochondries et noyau nous est donnée chez les plantes (Figure 1B,C) [
3]. Ainsi, dans la famille des Papilionacées1, le gène cox2 codant pour une sous-unité du complexe IV (cytochrome oxydase ; Figure 1A) de la chaîne respiratoire (CR) va se trouver porté par l’ADNmt et avoir une copie non fonctionnelle dans le génome nucléaire. Chez une espèce de plante très voisine, la copie nucléaire est devenue active, alors que dans une troisième espèce, la copie mitochondriale sera absente. La séquence des copies retrouvées dans le noyau montre cependant que selon toute probabilité, chez ces plantes, ce n’est pas de l’ADN qui a quitté les mitochondries mais plutôt de l’ARN édité (modification post-transcriptionnelle de certains C en U fréquente dans les mitochondries végétales). L’insertion de l’ARN ainsi exporté des mitochondries dans le génome nucléaire résulterait d’une activité transcriptase inverse. Chez l’homme, un phénomène voisin implique des fragments d’ADNmt appelés NUMT (nuclear DNA sequences from mitochondrial origin), mais cette fois, comme dans la levure Saccharomyces cerevisiae, il semble bien qu’il s’agisse réellement d’ADN inséré lors de la réparation de cassures double brin de l’ADN nucléaire [
4]. Une telle translocation d’ADN du cytoplasme vers le noyau pourrait intervenir lors de la désorganisation de la membrane nucléaire intervenant durant les divisions cellulaires ou grâce à un transport actif via les complexes formant les pores nucléaires. L’insertion de ces fragments de gènes mitochondriaux perturberait l’expression de gènes normalement portés par le génome nucléaire. Elle semble se produire en effet de façon préférentielle dans des régions codantes ou régulatrices et pourrait rendre compte de certaines pathologies. Encore chez les plantes et plus spectaculaire, le séquençage du génome d’une crucifère (Arabidopsis thaliana) a révélé une insertion de la quasi-totalité du génome mitochondrial (367 kb) dans le chromosome 2 [
5]. Inversement, les génomes mitochondriaux des végétaux, dont la taille approche 600 kb, ont acquis des séquences nucléaires, chloroplastiques ou virales, auxquelles s’ajoute un grand nombre de séquences d’origine et de fonction inconnues [
6]. Des plasmides mitochondriaux autonomes sont également présents dans beaucoup d’espèces végétales [
7]. Le mécanisme à la base de tous ces échanges est inconnu, cependant leur existence laisse à penser qu’un transfert d’ADN à partir de, et vers, les mitochondries est possible. Une expérience de mise en contact d’ADN nu avec des mitochondries isolées a ainsi montré que des mitochondries intactes extraites de végétaux peuvent importer de l’ADN nu par des voies naturelles [
8]. De façon similaire, des mitochondries isolées de foie de rat ou de cellules humaines se sont révélé être compétentes dans l’import d’ADN [
9]. L’ADN ainsi importé peut servir de matrice pour re-synthétiser de l’ADN ou pour permettre une transcription en ARN, résultant en un ARN polycistronique pouvant ensuite subir les étapes de maturation. Bien que le mécanisme impliqué dans ce transport d’ADN nu n’ait pas pu être élucidé à ce jour, il rappelle la compétence naturelle de certaines bactéries, dont des protéobactéries, à réaliser un tel transport. Dans la mesure où les mitochondries sont considérées comme ayant évolué à partir de telles protéobactéries, il est tentant d’imaginer que ce transport représente la persistance d’une voie ancestrale d’origine bactérienne. Confortant cette idée, il a été montré récemment que de l’ADN peut être transféré par conjugaison entre des bactéries Escherichia coli et des mitochondries de mammifères [
10]. |
Importer de l’ARN dans les mitochondries Cette fois, même s’il subsiste de nombreuses incertitudes, les données ne manquent pas quant aux mécanismes qui permettent d’importer de l’ARN dans les mitochondries, puisque ces systèmes existent, en particulier pour les ARN de transfert (ARNt), chez plusieurs protozoaires, plantes et levures [
11]. Les mitochondries de mammifères n’importent pas naturellement d’ARNt, mais pourraient importer certains petits ARN, en particulier l’ARN ribosomique (ARNr)5S [
12]. Un mécanisme de transport mitochondrial des ARNt serait néanmoins présent de manière cryptique dans la cellule humaine. Ce système requiert plusieurs protéines cytosoliques permettant de cibler les ARNt vers les mitochondries, étape suivie par une internalisation nécessitant l’intégrité du système d’import des protéines (Figures 2 et3) [
13]. Le processus d’import d’un ARNtLys a été particulièrement bien étudié chez la levure et ses facteurs essentiels ont été identifiés [
14]. Il est également possible d’introduire une ou plusieurs mutations dans un ARNt, jusqu’à changer son identité (le faire charger par un autre acide aminé), et garder sa capacité à être importé dans les mitochondries. Ces connaissances ont été exploitées pour mettre au point le premier modèle de complémentation d’une mutation pathogénique dans un ARNt normalement codé par l’ADNmt. Pour cela, deux versions d’ARNtLys ont été exprimées dans des cellules humaines dont l’ADNmt était muté. Il a été démontré que l’expression des ARNtLys importables de levure dans les cellules humaines mutées dans le gène d’ARNtLys mitochondrial (A8344G, cause reconnue du syndrome myoclonic epilepsy with ragged red fibers ou MERRF2
) permettait une récupération partielle des fonctions mitochondriales affectées par la mutation, comme l’activité des complexes respiratoires, le potentiel de membrane, la synthèse des protéines [
15]. Ainsi, l’existence de systèmes cryptiques d’import d’ARNt présents chez l’homme permettent l’import d’ARNt provenant de levure et ainsi la complémentation fonctionnelle de déficits liés à des mutations d’ARNt mitochondriaux. Un autre groupe d’organismes protozoaires, les trypanosomatidés, possède un appareil d’import des ARNt différent de celui de la levure. Indépendant de l’appareil d’import des protéines, il est constitué d’un complexe multi-protéique RIC (RNA import complex) ancré dans la membrane interne des mitochondries. Il a été démontré que le système d’import RIC pouvait être incorporé dans les mitochondries isolées humaines et promouvoir un import très efficace d’ARNt (Figure 2) [
16]. Plus récemment, en se fondant sur ces résultats in vitro, il a été montré que l’on pouvait forcer le complexe RIC à entrer dans des cellules humaines par une voie contrôlée par la cavéoline. Une fois dans les cellules, le complexe RIC s’associe aux mitochondries et permet l’import d’ARNt. Ainsi il est possible de complémenter fonctionnellement des fibroblastes humains porteurs de mutations dans l’ARNtLys mitochondrial associées aux syndromes MERFF et KSS (Kearns-Sayre syndrome, une maladie systémique où s’observent souvent une ophtalmoplégie, une rétinite pigmentaire, une dysphagie, une faiblesse musculaire proximale, une surdité, une ataxie cérébelleuse et des défauts de conduction cardiaque) [
17]. À côté de ces manipulations permettant l’adressage artificiel d’ARNt, les mitochondries humaines, comme noté plus haut, ont la compétence d’importer, de façon naturelle cette fois, l’ARNr 5S [12]. Cet import requiert de l’énergie sous forme d’ATP et la présence de protéines additionnelles, suggérant que, comme celui des ARNt, ce transport s’effectue par le biais du système d’import des protéines. Il serait tentant d’exploiter cette voie pour développer un autre système de complémentation, en adressant dans les organites des ARNr 5S recombinants ayant une activité thérapeutique. |
Importer des protéines dans les mitochondries Deux stratégies ont été développées afin d’importer cette fois des protéines dans les mitochondries en utilisant les systèmes d’import naturellement présents (Figure 3) [
18,
19]. Dans le but de complémenter le dysfonctionnement d’une protéine normalement codée par un gène porté par l’ADNmt, on a tenté - après avoir changé le code du gène pour le rendre compatible avec le système de traduction cytosolique - d’exprimer ce gène dans le noyau des cellules [18]. Lorsqu’elle est pourvue d’une séquence d’adressage mitochondriale, la protéine nucléaire produite peut être reconnue par le système TOM (translocator of the outer membrane) puis TIM (translocator of the inner membrane) et être importée dans les mitochondries. L’efficacité est néanmoins relativement faible, car les protéines codées par le génome mitochondrial humain sont connues pour leur forte hydrophobicité [19]. La nature hydrophobe de ces protéines s’oppose à un trafic intracellulaire efficace permettant leur distribution aux mitochondries. Pour résoudre ce délicat problème, certains ont proposé l’addition de séquences permettant d’adresser l’ARN messager (ARNm) aux ribosomes associés aux mitochondries ce qui favoriserait la synthèse des protéines à la surface de ces organites [
20]. Ainsi, les protéines synthétisées ne transitent plus dans la phase aqueuse cytosolique, mais sont prises en charge par le système TIM/TOM, permettant une complémentation efficace des déficits de la chaîne respiratoire. Autre approche, l’import de protéines normalement absentes chez l’homme et dont la fonction peut permettre de court-circuiter des segments non fonctionnels de la chaîne respiratoire (CR) (Figure 4). Ainsi, il existe chez les plantes et chez certains micro-organismes, en particulier la levure S. cerevisiae, une protéine qui, à elle seule, est susceptible de transférer les électrons du NADH mitochondrial vers le pool des quinones [
21]. Importée dans la mitochondrie, cette protéine, nommée Ndi1 chez la levure, peut permettre de contourner le complexe I [
22]. Ndi1 peut ainsi complémenter fonctionnellement des cellules humaines déficitaires en complexe I [
23]. Les plantes et certains animaux possèdent aussi une oxydase alternative (AOX), susceptible de transférer les électrons depuis le pool de quinones jusqu’à l’oxygène pour former de l’eau [
24]. Cette voie permet de court-circuiter tout le segment cytochromique terminal de la CR. Les conditions d’activation de cette voie sont particulièrement intéressantes puisqu’elles impliquent une forte réduction du pool des quinones [
25] et la présence d’acides organiques tels que le pyruvate [
26]: deux conséquences reconnues des déficits touchant la CR. Il a été démontré récemment que l’AOX pouvait être exprimée sans effets néfastes détectables dans des cellules humaines en culture, cellules dont la respiration devient alors insensible au cyanure (inhibiteur de la cytochrome oxydase) [
27] et dont les mitochondries produisent moins de superoxydes en cas de blocage de la CR. Les travaux visent désormais à complémenter des cellules déficitaires en cytochrome oxydase par la protéine AOX, alors que plusieurs équipes unissent leurs forces pour créer différents modèles animaux (souris et mouches) porteurs de cette protéine. Finalement, il a été montré dans des cellules en culture que l’import dans les mitochondries d’une enzyme de restriction, en l’occurrence SmaI, coupant spécifiquement l’ADNmt muté dans le gène ATPase 6 (T8993G), permettait de détruire l’ADN muté [
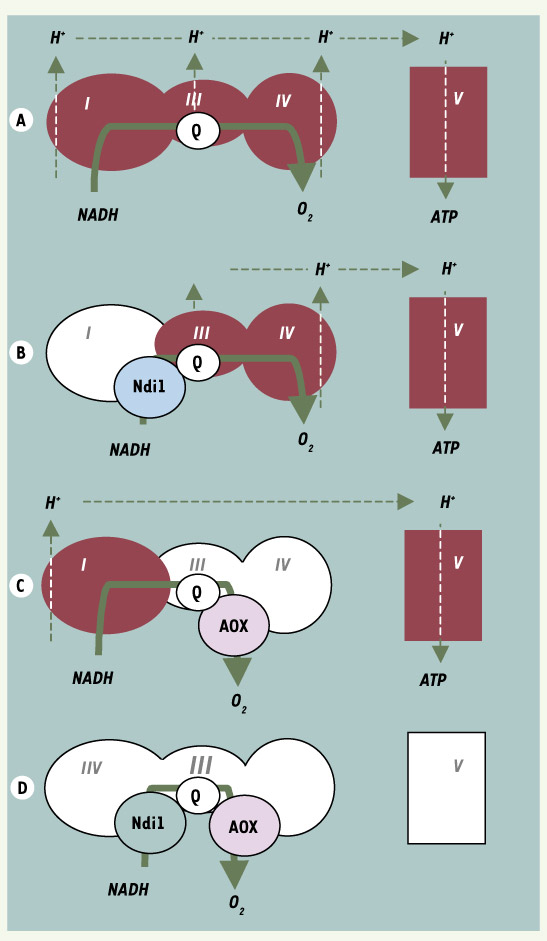
28].  | Figure 4.
Complémentation fonctionnelle de déficits de la chaîne respiratoire par des protéines allotopiques. A. La chaîne respiratoire des mammifères permet au cours du transfert des électrons d’établir un gradient électro-chimique à travers la membrane interne des mitochondries. L’ATPase (complexe V) utilise le gradient de protons ainsi établi pour synthétiser l’ATP à partir d’ADP et de phosphate inorganique. B. L’expression de la protéine Ndi1 de la levure Saccharomyces cerevisiae permet à elle seule de court-circuiter les 46 sous-unités du complexeI de la chaîne respiratoire. C. De façon similaire l’expression de l’oxydase alternative (AOX) permet de court-circuiter la partie cytochromique de la chaîne respiratoire. D. L’expression simultanée des protéines Ndi1 et AOX permet d’envisager la mise en route d’une voie alternative de transfert d’électrons telle qu’elle existe chez les plantes, totalement indépendante de la synthèse d’ATP. |
|
Il apparaît qu’importer du matériel exogène dans les mitochondries se révèle dès maintenant une stratégie productive et prometteuse pour l’avenir. Dans un premier temps, elle révèle et exploite des propriétés des mitochondries encore largement inconnues. La place de ces dernières dans nombre de pathologies confère à ces informations nouvelles toute leur valeur. Mais outre ces aspects fondamentaux, elle ouvre une voie nouvelle pour mieux comprendre ces pathologies, la complémentation fonctionnelle de tout ou partie des déficits permettant en particulier de mieux en apprécier les différents composants. Ainsi, si l’expression des protéines Ndi1 et AOX va permettre le rétablissement du flux d’électrons vers l’oxygène, elle ne restituera que partiellement le gradient de protons nécessaire à la synthèse d’ATP (Figure 4).
Bien sûr, il n’est pas question de prétendre ici que ces approches vont déboucher demain sur des thérapies. Les obstacles restent nombreux, en particulier dans le cas de maladies pluri-systémiques comme peuvent l’être parfois les maladies mitochondriales. Pourtant, ces études ouvrent une nouvelle approche vers la thérapie de ces maladies qui nous laissent très démunis. Bien entendu, elles vont rencontrer en outre les problèmes inhérents à toute thérapie génique - ciblage des cellules atteintes, rejet par l’organisme, difficulté de production des vecteurs, contrôle de l’innocuité des manipulations effectuées, etc.-mais elles préparent les outils thérapeutiques de demain pour vaincre les maladies mitochondriales. |
Footnotes |
1. Wallace DC. A mitochondrial paradigm of metabolic and degenerative diseases, aging, and cancer: a dawn for evolutionary medicine. Annu Rev Genet 2005; 39 : 359–407. 2. Mokranjac D, Neupert W. Protein import into mitochondria. Biochem Soc Trans 2005; 33 : 1019–23. 3. Nugent JM, Palmer JD. RNA-mediated transfer of the gene coxII from the mitochondrion to the nucleus during flowering plant evolution. Cell 1991; 66 : 473–81. 4. Ricchetti M, Tekaia F, Dujon B. Continued colonization of the human genome by mitochondrial DNA. PLoS Biol 2004; 2 : E273. 5. Stupar RM, Lilly JW, Town CD, et al. Complex mtDNA constitutes an approximate 620-kb insertion on Arabidopsis thaliana chromosome 2: implication of potential sequencing errors caused by large-unit repeats. Proc Natl Acad Sci USA 2001; 98 : 5099–103. 6. Knoop V. The mitochondrial DNA of land plants: peculiarities in phylogenetic perspective. Curr Genet 2004; 46 : 123–39. 7. Brown GG, Zhang M. Mitochondrial plasmids: DNA and RNA. In: Levings III, C S, Vasil I K, eds. The molecular biology of plant mitochondria. Dordrecht: Kluwer Academic Publishers, 1995. 8. Koulintchenko M, Konstantinov Y, Dietrich A. Plant mitochondria actively import DNA via the permeability transition pore complex. EMBO J 2003; 22 : 1245–54. 9. Koulintchenko M, Temperley R J, Mason PA, et al. Natural competence of mammalian mitochondria allows the molecular investigation of mitochondrial gene expression. Hum Mol Genet 2006; 15 : 143–54. 10. Yoon YG, Koob MD. Transformation of isolated mammalian mitochondria by bacterial conjugation. Nucleic Acids Res 2005; 33 : e139. 11. Entelis NS, Kolesnikova OA, Martin RP, Tarassov IA. RNA delivery into mitochondria. Adv Drug Deliv Rev 2001; 49 : 199–215. 12. Entelis NS, Kolesnikova OA, Doga S, et al. 5 S rRNA and tRNA import into human mitochondria. Comparison of in vitro requirements. J Biol Chem 2001; 276 : 45642–53. 13. Kolesnikova OA, Entelis NS, Mireau H, et al. Suppression of mutations in mitochondrial DNA by tRNAs imported from the cytoplasm. Science 2000; 289 : 1931–3. 14. Entelis N, Brandina I, Kamenski P, et al.A glycolytic enzyme, enolase, is recruited as a cofactor of tRNA targeting toward mitochondria in Saccharomyces cerevisiae.
Genes Dev 2006; 20 : 1609–20. 15. Kolesnikova OA, Entelis NS, Jacquin-Becker C, et al. Nuclear DNA-encoded tRNAs targeted into mitochondria can rescue a mitochondrial DNA mutation associated with the MERRF syndrome in cultured human cells. Hum Mol Genet 2004; 13 : 2519–34. 16. Mahata B, Bhattacharyya SN, Mukherjee S, Adhya S. Correction of translational defects in patient-derived mutant mitochondria by complex-mediated import of a cytoplasmic tRNA. J Biol Chem 2005; 280 : 5141–4. 17. Mahata B, Mukherjee S, Mishra S, et al. Functional delivery of a cytosolic tRNA into mutant mitochondria of human cells. Science 2006; 314 : 471–4. 18. Manfredi G, Fu J, Ojaimi J, et al. Rescue of a deficiency in ATP synthesis by transfer of MTATP6, a mitochondrial DNA-encoded gene, to the nucleus.Nat Genet 2002; 30 : 394–99. 19. Bokori-Brown M, Holt IJ. Expression of algal nuclear ATP synthase subunit 6 in human cells results in protein targeting to mitochondria but no assembly into ATP synthase. Rejuvenation Res 2006; 9 : 455–69. 20. Kaltimbacher V, Bonnet C, Lecoeuvre G, et al. mRNA localization to the mitochondrial surface allows the efficient translocation inside the organelle of a nuclear recoded ATP6 protein. RNA 2006; 12 : 1408–17. 21. Bénit P, Lebon S, Chol M, et al. Mitochondrial NADH oxidation deficiency in humans. Curr Genomics 2004; 5 : 137–46. 22. Yagi T, Seo BB, Nakamaru-Ogiso E, et al. Can a single subunit yeast NADH dehydrogenase (Ndi1) remedy diseases caused by respiratory complex I defects ? Rejuvenation Res 2006; 9 : 191–7. 23. Bai Y, Hu P, Park JS, et al. Genetic and functional analysis of mitochondrial DNA-encoded complex I genes. Ann NY Acad Sci 2004; 1011 : 272–83. 24. McDonald A, Vanlerberghe G. Branched mitochondrial electron transport in the animalia: presence of alternative oxidase in several animal phyla.IUBMB Life 2004; 56 : 333–41. 25. Affourtit C, Krab K, Moore AL. Control of plant mitochondrial respiration. Biochim Biophys Acta 2001; 1504 : 58–69. 26. Umbach AL, Ng VS, Siedow JN. Regulation of plant alternative oxidase activity: a tale of two cysteines. Biochim Biophys Acta 2006; 1757 : 135–42 27. Hakkaart GA, Dassa EP, Jacobs HT, Rustin P. Allotopic expression of a mitochondrial alternative oxidase confers cyanide resistance to human cell respiration. EMBO Rep 2005; 7 : 341–5. 28. Tanaka M, Borgeld HJ, Zhang J, et al. Gene therapy for mitochondrial disease by delivering restriction endonuclease SmaI into mitochondria. J Biomed Sci 2002; 9 : 534–41. |