| |
| Med Sci (Paris). 2007 June; 23(6-7): 626–632. Published online 2007 June 15. doi: 10.1051/medsci/20072367626.Les cancers du sein Vers un modèle cellulaire et moléculaire intégré Emmanuelle Charafe-Jauffret, Max Chaffanet, François Bertucci, Christophe Ginestier, Jocelyne Jacquemier, Odile deLapeyrière, and Daniel Birnbaum* UMR 599 Inserm Institut Paoli-Calmettes Laboratoire d’Oncologie Moléculaire, Centre de Recherche en Cancérologie de Marseille Université de la Méditerranée, 27, boulevard Leï Roure, 13009 Marseille, France |
Le cancer du sein demeure une maladie mal connue. Afin d’appliquer le traitement le mieux adapté à chaque patiente, en fonction du type et de la gravité de sa maladie, il est nécessaire d’aborder le cancer du sein dans sa complexité. Pour atteindre ce but, de nombreuses questions restent à résoudre : quelles sont les différentes classes de cancers du sein ? Quels sont les gènes altérés à l’origine de l’oncogenèse mammaire ? Quelles sont les cellules touchées par ces altérations ? Des éléments de réponse à ces différentes questions sont apportés de manière continue par les résultats des recherches actuelles dont nous présentons ici un bref état des lieux. |
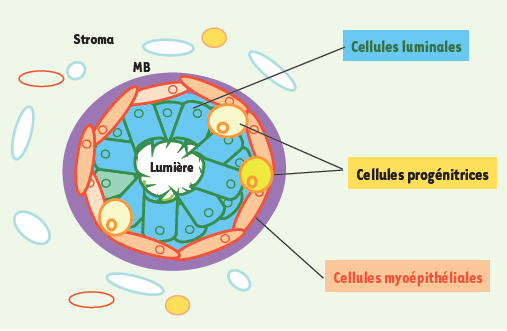
L’épithélium mammaire : une diversité cellulaire provenant de cellules souches Pour comprendre l’oncogenèse mammaire et la classification actuelle des cancers du sein, il faut connaître l’histologie de l’épithélium mammaire normal. Ce dernier est constitué de deux types de cellules différenciées, les cellules luminales et les cellules myoépithéliales (Figure 1). ll faut y ajouter des cellules souches et des progéniteurs plus ou moins engagés dans une voie de différenciation. Les cellules luminales bordent la lumière des canaux et des lobules. Les cellules myoépithéliales entourent les cellules luminales et sont en contact avec la lame basale et le stroma environnant. Les cellules souches, plus rares, sont situées en position basale ou supra-basale, probablement au niveau de niches spécialisées.  | Figure 1.
Coupe transversale d’un canal de la glande mammaire. Le schéma montre les différentes cellules épithéliales entourées de stroma. On distingue trois types de cellules : les cellules luminales, en contact avec la lumière, les cellules myoépithéliales, en contact avec la membrane basale (MB) et les cellules progénitrices (cellules souches et précurseurs immatures) en position basale ou supra-basale. |
Les cellules luminales expriment des marqueurs associés aux récepteurs hormonaux (récepteurs des œstrogènes et de la progestérone), certaines cytokératines (CK8, CK18) ainsi que des facteurs de transcription spécifiques comme GATA3 et FOXA1. Les cellules myoépithéliales expriment d’autres cytokératines (CK14) et, à l’état très différencié, des marqueurs du muscle lisse (actine du muscle lisse). L’orientation d’un progéniteur épithélial bipotent vers le lignage luminal ou myoépithélial pourrait être sous le contrôle de gènes « maîtres », par exemple GATA3 pour le lignage luminal. Chez la souris, la perte de GATA3 provoquée par une invalidation génique conditionnelle conduit à une expansion des cellules progénitrices luminales. GATA3 pourrait jouer le rôle de régulateur important de la différenciation luminale en maintenant l’état quiescent des cellules progénitrices et l’état différencié des cellules luminales [
1–
3]. Pour démontrer le caractère « maître » de GATA3, ce facteur a été exprimé dans des cellules souches et les résultats montrent qu’il induirait bien une différenciation luminale. L’ensemble de ces données récentes suggère une implication de GATA3 dans les cancers du sein. De fait, une expression élevée de GATA3 est une signature des cancers luminaux peu envahissants [
4,
5]. À l’inverse, il reste à prouver que la perte d’expression de GATA3 est associée à des tumeurs peu différenciées et envahissantes et à déterminer le rôle exact joué par GATA3 dans la progression des tumeurs du sein. |
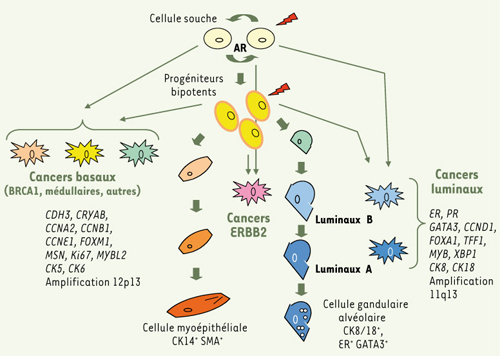
Peut-on réduire l’hétérogénéité de la maladie à des classes homogènes et proposer une classification pertinente ? Jusqu’à la fin des années 1990, seules les données histologiques et cliniques permettaient de classer les différents types de cancer du sein. Plus récemment, l’utilisation de techniques génomiques à haut débit comme les puces à ADN a permis d’y ajouter une caractérisation moléculaire. Les premières études d’expression génique dans les cancers du sein ont montré l’hétérogénéité moléculaire de la maladie, déjà suspectée au vu des données cliniques et histologiques. Cinq sous-types majeurs ont été identifiés sur la base de l’expression transcriptionnelle d’un demi-millier de gènes [
6] : luminal A, luminal B, basal, ERBB2 et normal. Cette classification a ensuite été validée par plusieurs plates-formes d’analyse transcriptionnelle, pour différentes populations de patientes et différentes formes anatomocliniques de la maladie. Elle permet de dégager des groupes de pronostic différent. Combinée aux classifications cliniques et histologiques classiques [
7,
8], elle représente une base nosologique, en même temps qu’un des succès de l’analyse transcriptionnelle, si souvent décriée à tort ou à raison. Les sous-types luminal A et basal sont les plus affirmés et sont très différents l’un de l’autre (environ 5 000 gènes différentiellement exprimés). Le sous-type luminal A correspond à des cancers généralement de bas grade, exprimant des récepteurs hormonaux et d’évolution plutôt favorable. Leur traitement inclut une hormonothérapie dans la majorité des cas. Les gènes exprimés dans ces cancers sont associés au récepteur des œstrogènes (ERα) et à la différenciation luminale (GATA3) (Figure 2). Les cancers basaux n’expriment ni les récepteurs hormonaux ni ERBB2. Contrairement aux cancers luminaux, ils sont de haut grade et de mauvais pronostic. Très prolifératifs, ils expriment les gènes codant pour des régulateurs du cycle cellulaire (cyclines, CDK, etc.) et présentent fréquemment des mutations de TP53. Ce sous-type, traité classiquement par chimiothérapie, ne dispose pas encore de thérapie spécifique, mais plusieurs cibles potentielles figurent dans la liste des gènes surexprimés (kinases, gènes du cycle…). Dans les lignées cellulaires dérivées de cancer du sein, il est possible de subdiviser le sous-type basal en A et B [
9]. Le sous-type basal B pourrait être identifié par l’expression de marqueurs du mésenchyme (vimentine). Il serait similaire au sous-type de lignées décrit comme « mésenchymateux » [
10]. La définition formelle des sous-types repose sur les profils d’expression mais, pour des raisons pratiques, beaucoup assimilent le sous-type basal aux cancers du sein « triple négatifs » (n’exprimant pas les récepteurs hormonaux et ne surexprimant pas ERBB2). Le sous-type ERBB2 se caractérise par une forte expression de ERBB2 et des gènes de l’unité d’amplification 17q12 autour de ERBB2 [
11]. Ce sous-type peut être traité par un anticorps qui inhibe la voie ERBB2 comme le trastuzumab (HerceptineTM) ou un inhibiteur de tyrosine kinase comme le lapatinib [
12]). Le sous-type luminal B regrouperait des cas luminaux moins différenciés que les luminaux A et plus prolifératifs, et le sous-type normal reste assez mal défini. De nombreux cas de cancers du sein ne peuvent être inclus dans ces sous-types et demandent à être mieux caractérisés.  | Figure 2.
Différenciation épithéliale mammaire normale et tumorale. À partir de cellules souches et leurs descendants immédiats (progéniteurs bipotents prolifératifs), deux lignages cellulaires se développent, aboutissant à des cellules matures différenciées. Le lignage luminal comprend des cellules capables de produire le lait pendant la lactation. Le lignage myoépithélial entoure le précédent et comprend des cellules différenciées exprimant des marqueurs du muscle lisse. Des marqueurs spécifiques de lignage et de stade de différenciation sont indiqués. CK : cytokératine ; ER : récepteur des œstrogènes ; SMA : actine du muscle lisse. Les altérations génétiques touchent les cellules souches ou les progéniteurs (flèches rouges) produisant une tumeur faite de cellules immatures si les capacités de différenciation sont limitées (tumeurs basales) ou de cellules progressant le long du lignage luminal (tumeurs luminales). Le sous-type ERBB2 est représenté de façon spéculative car on ne connaît pas la cellule d’origine. Il est impossible de représenter le sous-type « normal » dont on sait trop peu de choses. Des gènes et altérations spécifiques de ces deux types de cancer sont indiqués. AR : auto-renouvellement. |
Ces sous-types peuvent être également distingués au niveau protéique. Des études d’expression par immunohistochimie sur tissue microarray ont permis d’identifier les sous-types luminaux et basaux ainsi que des marqueurs protéiques de sous-types, comme la cadhérine P, la moésine et CD44 pour les cancers basaux [
13–
16]. La différence entre les cancers basaux et luminaux est profonde. Il s’agit probablement de deux maladies différentes. Cela exige leur reconnaissance en routine et un traitement approprié. Différentes formes cliniques et histologiques de cancer du sein ont été étudiées. Les cinq sous-types sont observés dans les cancers du sein inflammatoires [
17], avec une prédominance de cancers basaux et ERBB2 [
18]. Les cancers du sein médullaires sont exclusivement basaux [
19], de même que les cancers héréditaires associés à une mutation de BRCA1 [
20]. Les cancers du sein lobulaires seraient de deux types, luminal et normal [
21,
22]. La possibilité d’intégrer ainsi progressivement la classification histoclinique et la classification moléculaire [8] devrait permettre une meilleure approche des traitements. |
Quels sont les gènes altérés dans les cancers du sein ? L’identification de nouveaux gènes cibles altérés est nécessaire pour améliorer et diversifier les traitements. Les gènes dont l’implication dans l’oncogenèse mammaire est prouvée sont encore peu nombreux, mais leur effectif pourrait croître rapidement. Certains de ces gènes sont activés par amplification de leur région chromosomique, comme CCND1 (11q13) et ERBB2 (17q12). D’autres gènes sont altérés par délétion, mutation ou perte d’expression, comme CDH1 (cadhérine E) dans les cancers lobulaires, TP53, et BRCA1 et BRCA2 dans les cancers héréditaires. La sous-unité catalytique P110 de la phosphatidyl-inositol-3 kinase est fréquemment mutée [
23]. De nombreuses amplifications et délétions récurrentes ciblant des régions chromosomiques précises ont été identifiées par hybridation génomique comparative. Cette approche, réalisée sur des cibles chromosomiques dans sa forme classique, et plus récemment sur puces à ADN (CGH-array, comparative genomic hybridization), montre deux types d’altérations. Le premier type est représenté par des gains ou pertes de grandes portions de matériel génétique. L’étendue de ces altérations, qui peuvent impliquer un bras entier de chromosome, rend difficile l’identification des éléments importants pour l’oncogenèse. Le deuxième type est fait d’altérations plus régionales, voire très localisées. C’est le cas notamment de l’amplification du gène ESR1 (ERα) qui serait retrouvée dans environ 20 % des cancers du sein. Cette amplification très localisée sur le bras long du chromosome 6 serait prédictive de la réponse au tamoxifène (thérapeutique anti-œstrogène très utilisée dans les cancers du sein) [
24]. Les gènes concernés par les altérations localisées sont plus faciles à identifier, mais des études de validation sont généralement nécessaires. Dans le cas de l’amplification 8p12 par exemple, le rôle de FGFR1, qui code pour un récepteur aux facteurs de croissance des fibroblastes, n’est toujours pas bien établi [
25–
28]. Certaines amplifications ont été associées à des impacts cliniques variables [
29]. Depuis peu, la très haute densité en oligonucléotides des puces à ADN commerciales (exemple : Agilent 244A®, Affymetrix GeneChip human mapping 500K set® ; Illumina®, Nimblegen 385K®) permet, par CGH-array, une détection exhaustive et résolutive des déséquilibres génomiques [
30]. Combinée à l’étude d’expression, voire à d’autres analyses telles que la FISH (fluorescence in situ hybridization) et la Q-PCR (quantitative polymerase chain reaction), l’approche CGH-array à haute résolution se révèle très puissante. Cela devrait permettre d’identifier des régions et des gènes altérés ayant échappé jusqu’à maintenant aux analyses plus grossières, et de comparer plus finement les génomes des sous-types moléculaires de cancer du sein (pour revue, voir [
31,
32]). Dans le même temps, des études de séquençage massif des cancers [
33–
35] ont été entreprises. Les premiers résultats font état de mutations dans de nombreux gènes [33–35]. Grâce à l’ensemble de ces approches, un répertoire des altérations présentes dans les cancers du sein sera bientôt disponible. Chacune de ces altérations devra faire l’objet d’une validation fonctionnelle prouvant son implication dans l’oncogenèse mammaire. Cela pourra se faire par différentes méthodes, par exemple modulation de l’expression (surexpression, inhibition d’expression par siARN) et utilisation de modèles animaux. Un exemple de ce type de démarche est décrit dans une étude récente sur le gène CRYAB, surexprimé dans les cancers du sein de sous-type basal [
36]. Une liste de cibles validées sera alors disponible pour développer de nouveaux traitements. |
Quelles sont les cellules à l’origine des cancers du sein ? L’efficacité d’un traitement sera maximale s’il touche la bonne cellule. Un nombre croissant d’arguments permettent de penser que le cancer du sein se développe à partir des cellules souches mammaires (ou de leurs descendants immédiats, les progéniteurs précoces) après une série d’altérations génétiques. Ces cellules, définies ainsi comme cellules souches cancéreuses (CSC) et dotées de la capacité à s’auto-renouveler, seraient les seules capables de pérenniser la croissance de la tumeur. L’auto-renouvellement et la différenciation seraient régulés par la division asymétrique, processus typique de la cellule souche normale ou tumorale par lequel elle donne naissance à une cellule fille identique et une cellule qui va s’engager dans la voie de la différenciation. Les CSC pourraient également être à l’origine des métastases. Un traitement ne pourrait donc être réellement efficace que s’il cible et détruit ces CSC ou agit sur leur environnement (la « niche »). Le problème majeur est d’isoler et de caractériser les CSC. Plusieurs techniques ont été décrites afin d’isoler des cellules souches de la glande mammaire normale ou cancéreuse. Certaines utilisent les propriétés intrinsèques de ces cellules à exclure les colorants vitaux comme le Hœchst (identifiant une side population, SP [36,
37]), d’autres leur capacité à survivre et à proliférer dans un milieu sans sérum et en conditions non-adhérentes (formation d’agrégats, mammosphères ou « tumorosphères », par analogie avec le mode de prolifération des cellules souches neurales sous forme de neurosphères, décrites initialement [
38,
39]), d’autres utilisent des marqueurs de surface pour trier les cellules en cytométrie de flux. Dans cette dernière catégorie, un phénotype particulier, CD44+/CD24-/lin- (lin, lineage, désignant une combinaison d’antigènes spécifiques de cellules différenciées), a été associé aux CSC mammaires humaines [
40]. Ces cellules ont été étudiées par puces à ADN et une signature d’expression génique a été établie ; elle est associée à une valeur pronostique [40,
41]. Nous avons démontré récemment que cette signature permettait de différencier les cancers du sein de sous-type luminal versus basal, avec une surexpression dans le sous-type basal des gènes surexprimés dans les CSC potentielles. À côté de ces marqueurs de surface, un nouveau marqueur témoignant de la fonction d’une enzyme impliquée dans la différenciation, apparaît très prometteur. Il s’agit de l’activité enzymatique de l’aldéhyde déshydrogénase (ALDH), qui intervient dans le métabolisme oxydatif de l’acide rétinoïque et qui est mesurable par fluorescence grâce à un kit commercial approprié, Aldefluor (StemCell technologies, Vancouver, Canada). Les cellules issues de mammoplastie de réduction exprimant l’activité ALDH sont capables de reconstituer les différents lignages cellulaires de l’épithélium mammaire normal in vitro et in vivo. Des cellules issues de tumeur exprimant l’activité ALDH sont jusqu’à 1 000 fois plus tumorigènes que les cellules Aldefluor-négatives, et peuvent reconstituer l’hétérogénéité de la tumeur initiale, ce qui reflète le caractère « souche » de ces cellules. De tels marqueurs permettront d’isoler les CSC et de les étudier. Sur ces populations enrichies et leurs dérivés xénotransplantés, il sera possible de pratiquer nombre d’études in vitro et in vivo (expression génique, protéomique, tests de tumorigénicité, de sensibilité aux drogues, obtention de lignées, étude du rôle des microARN, etc.) qui augmenteront notre compréhension de la maladie. |
Une synthèse très schématique et très provisoire Quels enseignements peut-on tirer de ces différentes études ? La meilleure caractérisation des cellules souches et des deux lignages mammaires ainsi que la définition des sous-types moléculaires permettent de dégager de grandes lignes cohérentes et un schéma provisoire (Figure 2). La dichotomie luminale/myoépithéliale cadre bien avec la différence luminale/basale des cancers du sein. Les cancers du sein auraient leur origine au niveau des cellules souches ou progénitrices (Figure 2). Selon le type d’altérations génétiques, et probablement aussi selon l’environnement dans lequel elles évoluent, les cellules issues des CSC progresseraient de façon plus ou moins importante vers un stade différencié. Les cancers de sous-type luminal proviendraient de CSC ou de progéniteurs déjà engagés donnant une descendance capable de se différencier en cellules luminales de façon plus ou moins complète (luminaux A et luminaux B, respectivement). Les cancers de sous-type basal auraient au contraire pour origine des cellules donnant une descendance capable de s’engager dans la différenciation myoépithéliale ou dans les deux lignages. Certains cancers du sein seraient ainsi constitués de cellules relativement bien différenciées (cancers luminaux A) et d’autres, au pronostic plus sévère (cancers basaux, cancers luminaux B), de cellules plus immatures (blocage précoce de la différenciation) et proliférantes. La différenciation des cellules tumorales ne va cependant pas jusqu’aux cellules à différenciation terminale, comme les cellules glandulaires produisant du lait ou les cellules myoépithéliales les plus matures. Des exceptions possibles sont les rarissimes cancers sécrétoires, caractérisés sur le plan moléculaire par le gène de fusion ETV6-NTRK3 [
42], et par les non moins rarissimes myoépithéliomes. Les cancers ERBB2 pourraient dériver de CSC ayant la capacité de s’orienter soit vers un lignage, soit vers l’autre, soit vers les deux. Le sous-type ERBB2 ne correspondrait pas à un lignage mais à une anomalie génétique capable de conférer à la tumeur des propriétés d’agressivité et un transcriptome particulier. Comme pour les leucémies le point de départ cellulaire du cancer du sein pourrait être double [
43]. Le cancer peut trouver son origine dans la cellule souche elle-même. Celle-ci étant dotée de capacité d’auto-renouvellement et d’une longue durée de vie, les altérations génétiques sont principalement à l’origine de la prolifération continue. Le cancer pourrait au contraire débuter dans les progéniteurs prolifératifs précoces. Les cancers basaux, très prolifératifs, pourraient dériver de tels progéniteurs. Les altérations génétiques modifient alors le programme moléculaire de façon à ce que la cellule acquière auto-renouvellement et survie prolongée. Les cancers issus de ces deux origines pourraient avoir des propriétés différentes. Il est possible de placer sur ce schéma des listes de gènes exprimés de manière spécifique dans tel ou tel lignage, et bientôt des gènes spécifiques des cellules souches mammaires. Les avancées moléculaires et cellulaires, conjointes et cohérentes, font progresser notre connaissance de l’oncogenèse mammaire. Cela devrait encore s’accélérer. L’application de méthodes de criblage type ChIP on chip permettra d’établir des réseaux de régulation de la différenciation et de définir une hiérarchie dans leurs interactions [3,
44]. Les études génomiques permettront de définir des altérations spécifiques de chaque sous-type de cancer [
45], comme par exemple l’amplification de la région 12p13 dans les cancers du sein basaux [29,
46]. De ces études émergeront marqueurs et cibles permettant le développement de l’arsenal thérapeutique et son application précise à chaque cas de cancer. La caractérisation des CSC permettra de les cibler par ces traitements adaptés.

|
Au total, on regroupe sans doute sous le terme de sous-type moléculaire plusieurs catégories de différences, associées aux lignages épithéliaux, aux stades de différenciation cellulaires et aux influences des anomalies génétiques. Une meilleure définition des sous-types moléculaires est donc nécessaire mais un modèle est né. Bien entendu, comme tous les modèles, celui-ci est destiné à être amélioré, modifié, contesté, remplacé, jusqu’à ce qu’un modèle consensuel, plus juste, plus complet et plus précis, soit utilisé en recherche comme en clinique. L’intérêt de ce premier modèle est néanmoins de montrer un début de cohérence et de convergence issu d’études diverses, moléculaires et cellulaires, et de poser les bases de réflexion pour les études et les prises en charge futures de la maladie. |
Nous remercions la Ligue Nationale Contre le Cancer pour son soutien.
|
1. Asselin-Labat ML, Sutherland KD, Barker H, et al. Gata-3 is an essential regulator of mammary-gland morphogenesis and luminal-cell differentiation. Nat Cell Biol 2006; 9 : 201–9. 2. Asselin-Labat ML, Shackleton M, Stingl J, et al. Steroid hormone receptor status of mouse mammary stem cells. J Natl Cancer Inst 2006; 98 : 1011–4. 3. Kouros-Mehr H, Slorach EM, Sternlicht MD, Werb Z. GATA-3 maintains the differentiation of the luminal cell fate in the mammary gland. Cell 2006; 127 : 1041–55. 4. Bertucci F, Houlgatte R, Benziane A, et al. Gene expression profiling of primary breast carcinomas using arrays of candidate genes. Hum Mol Genet 2000; 9 : 2981–991. 5. Jenssen TK, Kuo WP, Stokke T, Hovig E. Associations between gene expressions in breast cancer and patient survival. Hum Genet 2002; 111 : 411–20. 6. Sorlie T, Tibshirani R, Parker J, et al. Repeated observation of breast tumor subtypes in independent gene expression data sets. Proc Natl Acad Sci USA 2003; 100 : 8418–23. 7. Birnbaum D, Bertucci F, Ginestier C, Tagett R, et al. Basal and luminal breast cancers : basic or luminous ? Int J Oncol 2004; 25 : 249–58. 8. Charafe-Jauffret E, Ginestier C, Monville F, et al. How to best classify breast cancer : conventional and novel classifications. Int J Oncol 2005; 27 : 1307–13. 9. Neve RM, Chin K, Fridlyand J, et al. A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes. Cancer Cell 2006; 10 : 515–27. 10. Charafe-Jauffret E, Ginestier C, Monville F, et al. Gene expression profiling of breast cell lines identifies potential new basal markers. Oncogene 2006; 25 : 2273–84. 11. Bertucci F, Borie N, Ginestier C, et al. Identification and validation of an ERBB2 gene expression signature in breast cancers. Oncogene 2004; 23 : 2564–75. 12. Konecny GE, Pegram MD, Venkatesan N, et al. Activity of the dual kinase inhibitor lapatinib (GW572016) against HER-2-overexpressing and trastuzumab-treated breast cancer cells. Cancer Res 2006; 66 : 1630–9. 13. Abd El-Rehim DM, Ball G, Pinder SE, et al. High-throughput protein expression analysis using tissue microarray technology of a large well-characterised series identifies biologically distinct classes of breast cancer confirming recent cDNA expression analyses. Int J Cancer 2005; 116 : 340–50. 14. Dolled-Filhart M, Ryden L, Cregger M, et al. Classification of breast cancer using genetic algorithms and tissue microarrays. Clin Cancer Res 2006; 12 : 6459–68. 15. Jacquemier J, Ginestier C, Rougemont J, et al. Protein expression profiling identifies subclasses of breast cancer and predicts prognosis. Cancer Res 2005; 65 : 767–79. 16. Nielsen TO, Hsu FD, Jensen K, et al. Immunohistochemical and clinical characterization of the basal-like subtype of invasive breast carcinoma. Clin Cancer Res 2004; 10 : 5367–74. 17. Bertucci F, Finetti P, Rougemont J, et al. Gene expression profiling identifies molecular subtypes of inflammatory breast cancer. Cancer Res 2005; 65 : 2170–8. 18. Van Laere SJ, Van den Eynden GG, Van der Auwera I, et al. Identification of cell-of-origin breast tumor subtypes in inflammatory breast cancer by gene expression profiling. Breast Cancer Res Treat 2006; 95 : 243–55. 19. Bertucci F, Finetti P, Cervera N, et al. Gene expression profiling shows medullary breast cancer is a subgroup of basal breast cancers. Cancer Res 2006; 66 : 4636–44. 20. Turner NC, Reis-Filho JS. Basal-like breast cancer and the BRCA1 phenotype. Oncogene 2006; 25 : 5846–53. 21. Korkola JE, DeVries S, Fridlyand J, et al. Differentiation of lobular versus ductal breast carcinomas by expression microarray analysis. Cancer Res 2003; 63 : 7167–75. 22. Zhao H, Langerod A, Ji Y, et al. Different gene expression patterns in invasive lobular and ductal carcinomas of the breast. Mol Biol Cell 2004; 15 : 2523–36. 23. Buttitta F, Felicioni L, Barassi F, et al. PIK3CA mutation and histological type in breast carcinoma : high frequency of mutations in lobular carcinoma. J Pathol 2006; 208 : 350–5. 24. Holst F, Stahl PR, Ruiz C, et al. Estrogen receptor alpha (ESR1) gene amplification is frequent in breast cancer. Nat Genet 2007; 39 : 655–60. 25. Chin K, DeVries S, Fridlyand J, et al. Genomic and transcriptional aberrations linked to breast cancer pathophysiologies. Cancer Cell 2006; 10 : 529–41. 26. Gelsi-Boyer V, Orsetti B, Cervera N, et al. Comprehensive profiling of 8p11-12 amplification in breast cancer. Mol Cancer Res 2005; 3 : 655–67. 27. Ray ME, Yang ZQ, Albertson D, et al. Genomic and expression analysis of the 8p11-12 amplicon in human breast cancer cell lines. Cancer Res 2004; 64 : 40–7. 28. Reis-Filho JS, Simpson PT, Turner NC, et al. FGFR1 emerges as a potential therapeutic target for lobular breast carcinomas. Clin Cancer Res 2006; 12 : 6652–62. 29. Letessier A, Sircoulomb F, Ginestier C, et al. Frequency, prognostic impact, and subtype association of 8p12, 8q24, 11q13, 12p13, 17q12, and 20q13 amplifications in breast cancers. BMC Cancer 2006; 6 : 245. 30. Coe BP, Ylstra B, Carvalho B, et al. Resolving the resolution of array CGH. Genomics 2007; 89 : 647–53. 31. Edgren H, Kallioniemi O. Integrated breast cancer genomics. Cancer Cell 2006; 10 : 453–4. 32. Sorlie T. Molecular classification of breast tumors : toward improved diagnostics and treatments. Meth Mol Biol 2007; 360 : 91–114. 33. Benvenuti S, Arena S, Bardelli A. Identification of cancer genes by mutational profiling of tumor genomes. FEBS Lett 2005; 579 : 1884–90. 34. Chanock SM, Burdett LP, Yeager MP, et al. Somatic sequence alterations in twenty-one genes selected by expression profile analysis of breast carcinomas. Breast Cancer Res 2007; 9 : R5. 35. Sjoblom T, Jones S, Wood LD, et al. The consensus coding sequences of human breast and colorectal cancers. Science 2006; 314 : 268–74. 36. Moyano JV, Evans JR, Chen F, et al. AlphaB-crystallin is a novel oncoprotein that predicts poor clinical outcome in breast cancer. J Clin Invest 2006; 116 : 261–70. 37. Kondo T, Setoguchi T, Taga T. Persistence of a small subpopulation of cancer stem-like cells in the C6 glioma cell line. Proc Natl Acad Sci USA 2004; 101 : 781–6. 38. Dontu G, Abdallah WM, Foley JM, et al.
In vitro propagation and transcriptional profiling of human mammary stem/progenitor cells. Genes Dev 2003; 17 : 1253–70. 39. Ponti D, Costa A, Zaffaroni N, et al. Isolation and in vitro propagation of tumorigenic breast cancer cells with stem/progenitor cell properties. Cancer Res 2005; 65 : 5506–11. 40. Al Hajj M, Wicha MS, Benito-Hernandez A, et al. Prospective identification of tumorigenic breast cancer cells. Proc Natl Acad Sci USA 2003; 100 : 3983–8. 41. Liu R, Wang X, Chen GY, et al. The prognostic role of a gene signature from tumorigenic breast-cancer cells. N Engl J Med 2007; 356 : 217–26. 42. Tognon C, Knezevich SR, Huntsman D, et al. Expression of the ETV6-NTRK3 gene fusion as a primary event in human secretory breast carcinoma. Cancer Cell 2002; 2 : 367–76. 43. Wicha MS, Liu S, Dontu G. Cancer stem cells : an old idea : a paradigm shift. Cancer Res 2006; 66 : 1883–90. 44. Carroll JS, Brown M. Estrogen receptor target gene : an evolving concept. Mol Endocrinol 2006; 20 : 1707–14. 45. Bergamaschi A, Kim YH, Wang P, et al. Distinct patterns of DNA copy number alteration are associated with different clinicopathological features and gene-expression subtypes of breast cancer. Genes Chrom Cancer 2006; 45 : 1033–40. 46. Yao J, Weremowicz S, Feng B, et al. Combined cDNA array comparative genomic hybridization and serial analysis of gene expression analysis of breast tumor progression. Cancer Res 2006; 66 : 4065–78. |