| |
| Med Sci (Paris). 2008 March; 24(3): 248–251. Published online 2008 March 15. doi: 10.1051/medsci/2008243248.Origine cellulaire des tumeurs d’Ewing Un coin de voile est levé Franck Tirode, Karine Laud-Duval, and Olivier Delattre* Inserm U830, Institut Curie, 26, rue d’Ulm, 75248 Paris, France MeSH keywords: Adipocytes, Adolescent, Adulte, Tumeurs osseuses, Différenciation cellulaire, Transformation cellulaire néoplasique, Enfant, Chondrocytes, Régulation de l'expression des gènes tumoraux, Humains, Mésoderme, Modèles biologiques, Cellules souches tumorales, Protéines de fusion oncogénique, Ostéocytes, Protéine du proto-oncogène c-fli-1, Protéine EWS de liaison à l'ARN, Sarcome d'Ewing, Cellules stromales, Facteurs de transcription, Cellules cancéreuses en culture |
La tumeur d’Ewing, seconde tumeur de l’os par ordre de fréquence chez les adolescents et les jeunes adultes, présente une translocation chromosomique caractéristique fusionnant la partie 5’ du gène EWS à la partie 3’ d’un membre de la famille ETS. La fusion la plus commune, EWS-FLI-1, résulte d’une translocation entre les chromosomes 11 et 22 [
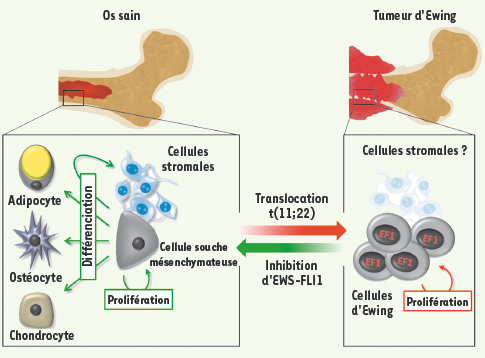
1]. Le rôle causal de cette fusion dans le développement tumoral a été clairement documenté. La compréhension des mécanismes intimes de cette transformation cellulaire repose très schématiquement d’une part sur l’élucidation du rôle biologique de la protéine EWS-FLI-1, et en particulier sur l’identification des principaux gènes cibles de ce facteur de transcription aberrant, et d’autre par sur la caractérisation précise du contexte cellulaire au sein duquel s’exerce cet effet.  | Figure 1.
Mécanismes de la transformation par EWS-FLI d’une cellule souche mésenchymateuse. Dans un os sain, les cellules souches mésenchymateuses sont capables à la fois d’autorenouvellement et de différenciation dans divers tissus. Suite à la survenue d’une translocation t(11 ; 22) et à l’expression d’une protéine EWS-FLI-1, cette cellule souche mésenchymateuse, devenue cellule d’Ewing, est bloquée dans sa différenciation terminale et prolifère rapidement. En inhibant l’expression d’EWS-FLI-1 dans les cellules d’Ewing, on restaure le phénotype mésenchymateux, ces cellules devenant à nouveau capables de se différencier. |
|
Nature de la cellule cible à l’origine des tumeurs d’Ewing : les multiples hypothèses D’un point de vue histologique, ces tumeurs sont caractérisées par de petites cellules rondes indifférenciées, possédant un petit cytoplasme, intriquées dans un stroma intercellulaire épars. Ces caractéristiques phénotypiques sont peu informatives sur la cellule à l’origine des tumeurs. Il y a 86 ans, James Ewing, auteur de la première description de cette tumeur [
2], proposait une origine endothéliale. Depuis lors, plusieurs hypothèses alternatives ont été proposées. L’hypothèse neurale est fondée sur l’observation que la fusion EWS-FLI-1 caractérise certes des tumeurs très indifférenciées mais aussi des tumeurs neuroectodermiques primitives qui expriment des marqueurs spécifiques du tissu neural. De plus, des cellules d’Ewing cultivées in vitro en présence d’une combinaison de facteurs de différenciation spécifique, forment des structures semblables à des neurites et expriment certains marqueurs caractéristiques de la lignée neurale [
3,
4]. Une autre hypothèse, fondée sur l’expression par les cellules d’Ewing de certaines protéines des jonctions serrées (comme ZO-1 ou la claudine) évoque une origine cellulaire épithéliale [
5]. Enfin, des études récentes menées chez la souris ont soulevé l’hypothèse mésenchymateuse. L’équipe de Suzan Backer a ainsi pu mettre en évidence que l’expression d’EWS-FLI-1 dans des cellules stromales osseuses bloquait leur différenciation terminale dans les voies adipocytaire et ostéocytaire [
6]. Puis les équipes de Robert Ilaria et d’Ivan Stamenkovic ont montré que des cellules stromales osseuses exprimant EWS-FLI-1 de façon stable induisent chez la souris des tumeurs très semblables aux tumeurs d’Ewing humaines [
7,
8]. Si ces études ne démontrent pas l’origine mésenchymateuse des tumeurs d’Ewing, elles n’en indiquent pas moins que ce contexte cellulaire est permissif à la transformation par le gène de fusion. |
Une stratégie : inhiber le transcrit de fusion pour démasquer la cellule d’origine De notre côté, nous avons fait l’hypothèse qu’une cellule d’Ewing dans laquelle EWS-FLI-1 est inhibée pourrait retrouver les caractéristiques de son type cellulaire d’origine et récupérer une capacité de réponse à certains programmes de différenciation [
9]. Afin d’inhiber EWS-FLI-1 nous avons développé un modèle de cellules d’Ewing exprimant un shARN spécifique du transcrit de fusion et inductible par la doxycycline. Ce système permet une extinction à long terme de l’expression d’EWS-FLI-1. Le profil d’expression génétique de ces cellules, exprimant ou non EWS-FLI-1, ainsi que celui de tumeurs d’Ewing ont été comparés aux profils de nombreux tissus normaux, dérivés en particulier du mésenchyme. Il apparaît qu’EWS-FLI-1 module l’expression de nombreux gènes associés à la lignée mésenchymateuse et en particulier aux cellules souches mésenchymateuses (CSM). Les cellules souches mésenchymateuses sont encore relativement peu caractérisées mais sont généralement isolées par leur capacité à adhérer au plastique et surtout par leur capacité de différenciation en adipocytes, chondrocytes, ostéocytes et cellules stromales médullaires1,. Nous avons donc cherché si des cellules d’Ewing, dans lesquelles le transcrit de fusion EWS-FLI-1 a été inhibé, et traitées par les combinaisons de facteurs de différenciation adéquates, pouvaient se différencier dans ces lignées caractéristiques de tissus mésenchymateux. Effectivement, l’inhibition d’EWS-FLI1 dans des cellules cultivées en présence de molécules stimulant la différenciation adipocytaire conduit à une expression forte de trois protéines typiques de cette lignée (PPARg 2,
FABP4 et LPL) ainsi qu’à l’apparition de vésicules lipidiques visualisables par un marquage positif à l’Oil-Red-O. De la même façon, l’expression de trois marqueurs ostéocytaire (SPP1 ou anciennement ostéopontine, ALPL, phosphatase alcaline, et RUNX2) ainsi qu’un marquage positif à la réaction de Von-Kossa2 sont obtenus lorsque ces cellules d’Ewing dont le transcrit EWS-FLI-1 est inhibé sont cultivées en présence du cocktail de différenciation ostéocytaire. Nous avons ensuite analysé par cytométrie en flux l’expression de différents marqueurs de surface habituellement utilisés pour isoler les cellules souches mésenchymateuses. L’expression de CD44, CD59, CD73 (ecto 5’ nucléotidase), CD29 (chaîne β1 des intégrines) et CD54 (ICAM-1) est effectivement induite par l’inhibition d’EWS-FLI-1. CD90 (Thy1), CD105 (endogline) et CD166 (ALCAM) sont quant à eux exprimés mais non modulés par EWS-FLI1. L’ensemble de nos résultats suggère très fortement que l’inhibition de la protéine de fusion permet aux cellules d’Ewing de retrouver un phénotype originel de type cellule souche mésenchymateuse. |
Cette origine cellulaire permet de mieux comprendre la localisation des tumeurs d’Ewing au niveau des os et des tissus mous, deux sources majeures de cellules souches mésenchymateuses. Il est intéressant de souligner que la protéine de fusion EWS-FLI1 réprime la transcription de plusieurs protéines inhibitrice de la voie IGF1 (insulin growth factor 1) de type IGFBP (IGF binding protein) et potentialise ainsi cette voie de signalisation [
10] qui semble jouer un rôle majeur dans l’équilibre entre ces cellules souches et leur niche. De manière intéressante, une étude récente indique que la première vague de cellules mésenchymateuses proviendrait de cellules de la crête neurale [
11], ce qui pourrait réconcilier les hypothèses neurales et mésenchymateuses quant à l’origine des cellules d’Ewing. Ces résultats devraient permettre de développer des modèles cellulaires plus pertinents et d’accélérer le développement d’un modèle murin de cette tumeur. Ils constituent également une donnée essentielle pour l’identification et la caractérisation des cellules souches cancéreuses. |
Footnotes |
1. Delattre O, Zucman J, Plougastel B, et al. Gene fusion with an ETS DNA-binding domain caused by chromosome translocation in human tumours. Nature 1992; 359 : 162–5. 2. Ewing J. Diffuse endothelioma of bone. Proc New York Pathol Soc 1921; 21 : 17–24. 3. Noguera R, Navarro S, Peydro-Olaya A, Llombart-Bosch A. Patterns of differentiation in extraosseous Ewing’s sarcoma cells. An in vitro study. Cancer 1994; 73 : 616–24. 4. Cavazzana AO, Miser JS, Jefferson J, Triche TJ. Experimental evidence for a neural origin of Ewing’s sarcoma of bone. Am J Pathol 1987; 127 : 507–18. 5. Schuetz AN, Rubin BP, Goldblum JR, et al. Intercellular junctions in Ewing sarcoma/primitive neuroectodermal tumor: additional evidence of epithelial differentiation. Mod Pathol 2005; 18 : 1403–10. 6. Torchia EC, Jaishankar S, Baker SJ. Ewing tumor fusion proteins block the differentiation of pluripotent marrow stromal cells. Cancer Res 2003; 63 : 3464–8. 7. Riggi N, Cironi L, Provero P, et al. Development of Ewing’s sarcoma from primary bone marrow-derived mesenchymal progenitor cells. Cancer Res 2005; 65 : 11459–68. 8. Castillero-Trejo Y, Eliazer S, Xiang L, et al. Expression of the EWS/FLI-1 oncogene in murine primary bone-derived cells Results in EWS/FLI-1-dependent, ewing sarcoma-like tumors. Cancer Res 2005; 65 : 8698–705. 9. Tirode F, Laud-Duval K, Prieur A, et al. Mesenchymal stem cell features of ewing tumors. Cancer Cell 2007; 11 : 421–9. 10. Prieur A, Tirode F, Cohen P, Delattre O. EWS/FLI-1 silencing and gene profiling of Ewing cells reveal downstream oncogenic pathways and a crucial role for repression of insulin-like growth factor binding protein 3. Mol Cell Biol 2004; 24 : 7275–83. 11. Takashima Y, Era T, Nakao K, et al. Neuroepithelial cells supply an initial transient wave of MSC differentiation. Cell 2007; 129 : 1377–88. |