| |
| Med Sci (Paris). 2008 March; 24(3): 270–276. Published online 2008 March 15. doi: 10.1051/medsci/2008243270.Redondance génétique et synchronisation cellulaire dans les horloges circadiennes Hugues Dardente* School of Biological Sciences, Zoology Building, Tillydrone Avenue, Aberdeen AB24 2TZ, Écosse, Royaume-Uni |
Rôle et organisation hiérarchique du système circadien Chez les mammifères la rythmicité des fonctions cellulaires, physiologiques et comportementales, indispensables à la survie de l’espèce et au bien-être de l’individu, résulte d’un fonctionnement temporellement coordonné de nombreuses unités rythmiques (oscillateurs) présentes dans la plupart des tissus, centraux et périphériques [
1–
3] (Figure 1). Les noyaux suprachiasmatiques (NSC) de l’hypothalamus constituent l’horloge centrale dominante ; ils ont en charge l’intégration de signaux externes (en particulier les conditions lumineuses) puis la distribution de l’heure astronomique aux oscillateurs circadiens (fonctionnant de façon endogène et avec une période proche, mais différente, de 24h). Les données actuelles indiquent que cette synchronisation des oscillateurs par les NSC met en jeu des médiateurs humoraux (mélatonine ou glucocorticoïdes par exemple) ainsi que des projections vers les organes périphériques via le système nerveux autonome [
4–
6]. En retour, ces oscillateurs régulent divers aspects de la physiologie locale. C’est ce que démontrent plusieurs études dans lesquelles des gènes (gènes de l’horloge) indispensables à l’expression de la rythmicité circadienne ont été surexprimés ou supprimés de façon spécifique dans certains tissus [
7–
10,
43]. Cette synthèse fait le point sur les connaissances actuelles dans ces domaines, notamment sur le rôle exact d’un de ces gènes de l’horloge, Clock, qui a fait l’objet d’une série d’articles publiés récemment [
11–
13].
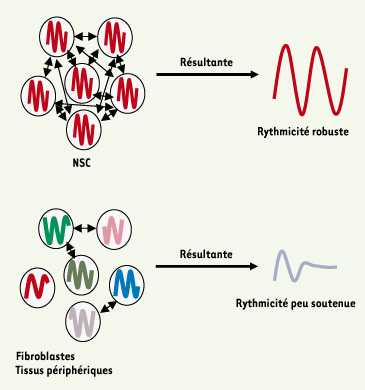
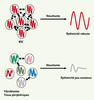 | Figure 1.
Synchronisation dans les noyaux suprachiasmatiques (NSC) et les tissus périphériques. Qu’elles proviennent des NSC ou qu’il s’agisse de fibroblastes, les cellules en culture présentent une activité rythmique persistante. Toutefois, au sein du tissu, il semble que les cellules des NSC soient très fortement couplées entre elles (flèches bidirectionnelles), ce qui permet d’assurer des phases d’expression cohérentes entre les neurones (rythmes en rouge, même phase). Il en résulte un message de sortie de forte amplitude. La résistance à diverses perturbations, notamment des mutations génétiques, semble être une des conséquences de cette organisation en un réseau serré (voir texte). Les cellules des tissus périphériques présentent également une rythmicité soutenue, mais les mécanismes de couplage (flèches bidirectionnelles) sont moins nombreux et/ou moins forts. Cela entraîne un déphasage rapide des cellules (rythmes de différentes couleurs, différentes phases). Il en résulte un message de sortie de moindre amplitude qui s’amortit plus rapidement que pour les NSC. Le rôle des NSC est à la fois de synchroniser les cellules au sein d’un tissu et les tissus entre eux (non représenté ici). |
|
Différents niveaux de contrôle cellulaire des rythmes circadiens Comme pour tout autre système biologique, il existe divers niveaux de contrôle possibles dans l’horloge circadienne :
-
Un contrôle transcriptionnel via des modifications de la chromatine (méthylations, acétylations ou phosphorylations…) permettant d’ouvrir ou de verrouiller la chromatine, autorisant ou interdisant de facto l’action de facteurs de transcription et de l’ARN polymérase [3]. L’emploi de promoteurs alternatifs constitue un autre niveau de contrôle transcriptionnel [
14,
15].
-
Un contrôle post-transcriptionnel notamment par épissage alternatif créant, à partir d’un même pré-ARN messager (ARNm), différents ARNm matures et donc différentes isoformes d’une même protéine (avec éventuellement différentes fonctions [
16]). Récemment, l’implication des micro-ARN (courts transcrits formant une structure secondaire complexe avec divers ARNm ayant pour conséquence leur dégradation rapide) dans le fonctionnement de l’horloge a également été proposée [
17]. Par ailleurs, la demi-vie des ARNm semble contrôlée de façon très précise par un équilibre entre des protéines qui stabilisent ou déstabilisent cet ARNm (par exemple LARK et Nocturnine [
18,
19]).
-
Un contrôle post-traductionnel, en particulier des phosphorylations, qui régule divers paramètres fonctionnels comme la capacité à dimériser, l’activité transcriptionnelle, la localisation subcellulaire et/ou la demi-vie de ces protéines [3,
20].
|
Mécanismes moléculaires des rythmes circadiens : les gènes de l’horloge Brièvement, les facteurs de transcription CLOCK et BMAL1 dimérisent (via leur domaine PAS, Per-ARNT-Sim1,) et contrôlent la transcription rythmique de certains gènes en se liant à l’ADN (via leurs domaines bHLH) sur des séquences de type « boîte E ». Certains de ces gènes, en particulier les gènes Period 1-3 (Per 1-3) et Cryptochromes 1-2 (Cry1-2) sont eux-mêmes des éléments de l’horloge. Les protéines PER1-3 et CRY 1-2 peuvent interagir et migrer dans le noyau où elles inhibent de façon rythmique l’activité transcriptionnelle du dimère CLOCK/BMAL1, bouclant ainsi la boucle [1, 3]. CLOCK/BMAL1 contrôle également l’expression rythmique d’autres gènes qui constituent des sorties « primaires » de l’horloge. Certains de ces gènes sont eux-mêmes des facteurs de transcription (REV-ERB, ROR ou DBP par exemple, voir plus loin). Il existe ainsi des sorties « secondaires » de l’horloge qui, à leur tour, définiront des sorties « tertiaires » et ainsi de suite. Cette cascade assure l’expression rythmique de très nombreux gènes suivant des phases distinctes [
21] (Figure 2).
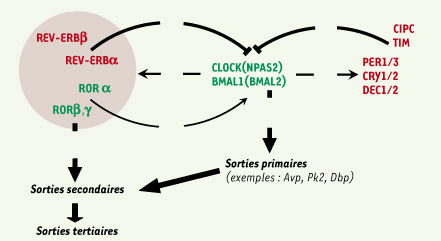
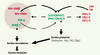 | Figure 2.
Mécanismes transcriptionnels de l’horloge moléculaire circadienne. Un dimère de facteurs de transcription CLOCK/BMAL1 (ou CLOCK/BMAL2, NPAS2/BMAL1, NPAS2/BMAL2) active l’expression de différents gènes via des séquences « boîtes E ». Les protéines PER, CRY, DEC, CIPC et TIM répriment l’activité du dimère. Les protéines REV-ERB et ROR répriment et activent la transcription du gène Bmal1 respectivement. Un contrôle transcriptionnel de CLOCK/BMAL1 sur les promoteurs de TIM, CIPC, REV-ERBβ, RORβ et RORγ reste encore à établir ; raison pour laquelle ils sont indiqués légèrement à l’écart. Voir le texte pour plus de précisions. AVP : arginine vasopressine ; DBP : D-site albumin binding protein ; PK2 : Prokineticine 2. |
Durant les dernières années, d’autres composants de l’horloge ont été identifiés, dont les récepteurs nucléaires orphelins des familles REV-ERB (2 membres) [
42] et ROR (3 membres), TIMELESS, les protéines DEC1-2, CIPC ainsi que diverses kinases et protéines liées à la voie du protéasome (Figures 2 et 3). Les approches de criblage génétique systématique développées par les équipes de J.S. Takahashi aux États-Unis et de P.M. Nolan en Grande-Bretagne ont déjà permis d’identifier les gènes Clock et Fbxl3 et il ne fait aucun doute que la liste d’acteurs moléculaires de l’horloge va encore s’étoffer.
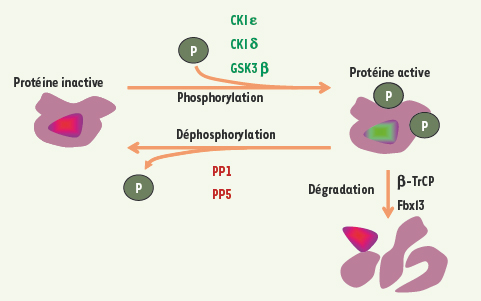
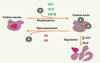 | Figure 3.
Mécanismes post-traductionnels de l’horloge moléculaire circadienne. Toute protéine de l’horloge est en équilibre entre une (ou plusieurs) forme active phosphorylée et une forme inactive déphosphorylée. La situation inverse, non représentée ici, est bien sûr possible. Trois kinases, CKIε, CKIδ, GSK3β (caséine kinase I epsilon et I delta, glycogène synthase kinase-3 beta) et deux phosphatases PP1 et PP5 (protéine phosphatases 1 et 5) ont été impliquées dans le fonctionnement de l’horloge. L’activation de la protéine modifie différents paramètres tels que : activité transcriptionnelle, localisation sub-cellulaire, capacité à dimériser. La protéine est ensuite dirigée vers les voies de dégradation. Cela implique les protéines β-TrCP ( beta-transducin repeat containing protein, pour la dégradation des protéines PER) et FBXL3 ( F-box and leucine-rich repeat protein 3 ; dégradation des protéines CRY), toutes deux membres du complexe E3 d’ubiquitine ligases [ 3, 20]. |
D’une manière générale la contribution de chacun de ces acteurs, considéré indépendamment, au mécanisme moléculaire, semble ne pas être fondamentale puisque les souris mutantes ou invalidées pour la plupart de ces gènes (plusieurs restent toutefois à caractériser) demeurent rythmiques, du moins sur le plan comportemental si l’on limite celui-ci à l’activité locomotrice de l’animal dans une roue (pour de plus amples explications [
2]). Cela semble être une conséquence de la redondance génétique du système. Par exemple, les protéines CLOCK et BMAL1 possèdent chacune un homologue structural et fonctionnel : NPAS2 (neuronal PAS domain protein 2) et BMAL2, respectivement [3]. Un des problèmes récurrents auquel se heurte l’étude des rythmes circadiens est celui de la définition même de gène de l’horloge : quel critère employer pour qualifier de façon univoque un nouveau composant présomptif de gène de l’horloge ? Le problème peut sembler assez trivial et d’ordre purement rhétorique. Il n’en est rien puisque cette question en cache une autre qui est centrale : quelle est la caractéristique fondamentale d’un rythme circadien ? Sa période ? Son amplitude relative ? Sa robustesse face à diverses perturbations ? Sa capacité à être synchronisé par un facteur externe ou un médiateur endocrine ? Deux critères sont généralement retenus comme marqueurs fiables du fonctionnement de l’horloge circadienne : la période du rythme et sa robustesse en condition d’obscurité constante (discussion dans [
22]). Donc, tout gène dont la suppression par invalidation génique (knock-out) entraîne une modification notable (plusieurs heures) de la période du rythme de l’activité locomotrice sur la roue, ou même son abolition totale (arythmie), est qualifié de gène de l’horloge. Quels sont alors les « vrais » gènes de l’horloge ? En 2006, trois gènes seulement répondaient à ce critère strict : Clock, Bmal1 et Per2. Qu’en est-il aujourd’hui ? |
Gènes de l’horloge : « À la fin il n’en restera qu’un (aucun ?) » Gène Bmal1
Bmal1 semble toujours solidement installé dans ce rôle puisque les souris invalidées pour ce gène (qui semblent être de vrais knock-out) sont immédiatement arythmiques en obscurité constante. Il y a donc certaines limites à la redondance fonctionnelle du système, puisque BMAL2, bien qu’il soit exprimé dans les NSC, n’est apparemment pas suffisant pour le sauvetage phénotypique de ces souris. Ces souris présentent par ailleurs une liste impressionnante de déficits et pathologies qui expliquent certainement la réduction drastique (environ 65 %) de leur espérance de vie [
23]. Ce phénotype peut être interprété comme une preuve de l’importance fonctionnelle des horloges circadiennes ; il peut aussi indiquer que la protéine BMAL1 joue un rôle majeur dans d’autres processus cellulaires. Par exemple, les souris double knock-out
Cry1/ Cry2 sont elles aussi arythmiques et pourtant leur espérance de vie est semblable à celle de souris sauvages. Cela pourrait être considéré comme la preuve d’un rôle non circadien de BMAL1 ; mais cette observation peut aussi être expliquée par le fait que l’horloge n’est pas bloquée au même niveau chez ces souris : activation transcriptionnelle constitutive par défaut de répression chez les souris knock-out pour Cry1/ Cry2, et absence d’activation transcriptionnelle pour les knock-out
Bmal1. La question d’éventuels rôles non circadiens pour les protéines de l’horloge demeure donc ouverte. Gène Per2
Pour Per2, les deux lignées murines invalidées initiales, issues de deux laboratoires indépendants, présentent un phénotype semblable : les souris deviennent rapidement arythmiques en obscurité constante. Toutefois, une troisième lignée knock-out pour laquelle la mutation est établie dans une autre souche murine, s’avère parfaitement rythmique [
24]. Gène Clock
Pour Clock la question a toujours été délicate. Les souris hétérozygotes pour la mutation du gène Clock présentent une période endogène d’environ 25h. Les souris homozygotes, initialement rythmiques en condition constante avec une période très longue (27-28h), deviennent rapidement arythmiques. Le point majeur ici est que cette souche murine, le « mutant Clock » n’est pas un knock-out du gène Clock. La protéine mutante (CLOCKΔ19) possède une délétion de 51 acides aminés correspondant à l’exon 19 du gène. La protéine CLOCKΔ19 se comporte comme un antimorphe, une forme de dominant négatif de la protéine CLOCK : elle peut toujours dimériser avec BMAL1 (car le domaine PAS est intact) et le dimère peut toujours se lier aux séquences « boîtes E » (car le domaine bHLH est lui aussi intact) mais elle semble dépourvue de potentiel transcriptionnel [
25,
26]. Le phénotype de ce mutant ne traduit peut-être pas un rôle direct de CLOCK dans l’horloge, mais pourrait être dû à la séquestration, par cette protéine tronquée, de certains facteurs importants (comme BMAL1). Il est aussi possible que CLOCKΔ19 occupe sur l’ADN des sites cruciaux en lieu et place d’autres facteurs, ou bien ne puisse pas lier certains cofacteurs nécessaires à l’activation de la transcription, bloquant ainsi l’horloge. Pour définir sans ambiguïté le rôle de CLOCK, il était nécessaire d’obtenir une invalidation du gène Clock, ce à quoi sont parvenus DeBruyne et al. [11]. Le résultat paraît, de prime abord, surprenant : les souris invalidées pour le gène Clock - qui n’ont aucune protéine détectable - sont rythmiques en obscurité constante contrairement au « mutant Clock ». Il semble donc que Clock ne soit pas un « vrai » gène de l’horloge. Toutefois, chez ces souris, les rythmes d’expression de nombreux gènes de l’horloge dans les NSC sont perturbés. Par ailleurs, l’amplitude des rythmes des ARNm de différentes sorties moléculaires (« primaires », donc directement contrôlées par CLOCK/BMAL1) de l’horloge suprachiasmatique (Avp, Pk2 et Dbp) est diminuée. Les réponses à la lumière sont également affectées. Pourquoi ces souris sont-elles toujours rythmiques ? Est-il envisageable qu’un autre composant de l’horloge puisse remplir les fonctions de CLOCK dans les NSC et ainsi effectuer un sauvetage phénotypique ? |
Redondance, redondance, tout est affaire de redondance… Comme nous l’avons mentionné précédemment, il existe une forte redondance dans les mécanismes de l’horloge circadienne. Alors que les souris knock-out pour Cry1 ou Cry2 ne sont pas arythmiques, les souris invalidées pour les deux gènes Cry1 et Cry2 le sont. Alors que Per2 n’est peut-être pas indispensable, les double knock-out Per1/Per2 sont arythmiques (pour revue des phénotypes, voir [22]). Or, Clock possède un homologue : Npas2. La protéine NPAS2 peut, à l’instar de CLOCK, dimériser avec BMAL1 et activer la transcription de gènes possédant des boîtes E [3]. Les souris invalidées pour le gène Npas2 ne présentent toutefois pas d’altération notable des rythmes circadiens [
27]. Néanmoins, NPAS2 a été impliquée dans le fonctionnement de divers oscillateurs hors des NSC, notamment dans le cerveau antérieur [
28] et dans le système vasculaire [
29]. Dans ces tissus NPAS2 remplit toutes les conditions requises pour être qualifiée d’homologue fonctionnel de CLOCK. Qu’en est-il dans les NSC ? D’après une étude antérieure [
30], Npas2 n’est pas détectable dans les NSC par hybridation in-situ (HIS) radioactive, technique toutefois peu sensible pour détecter des ARNm faiblement exprimés. En fait, bien qu’exprimé très faiblement, Npas2 est détectable dans les NSC par PCR quantitative [
12], une méthode plus sensible que l’HIS. Il était donc impossible d’exclure totalement un rôle de NPAS2 dans les NSC. DeBruyne et al. ont donc envisagé un possible phénomène de compensation chez ces souris. Confirmant le rôle présupposé de Npas2 comme analogue fonctionnel de Clock, et validant le mécanisme de compensation, DeBruyne et al. [11] ont montré que Npas2 était surexprimé dans le foie des souris invalidées pour Clock (voir aussi plus loin) comparativement aux souris de génotype « sauvage ». Malgré cela, de façon similaire à ce qui avait été observé dans les NSC, l’expression de divers gènes de l’horloge et de sorties de l’horloge demeure perturbée. Par contre aucune compensation n’a été observée dans les NSC : l’expression de Npas2 demeure indétectable par HIS. Comme noté précédemment il demeurait toutefois plausible qu’une expression très faible dans les NSC soit suffisante pour maintenir la rythmicité chez ces souris knock-out pour le gène Clock. |
Synchronisation et redondance sont nécessaires à une horloge robuste DeBruyne et al. [12]) ont donc produit des souris invalidées à la fois pour Clock et Npas2. Ces souris sont immédiatement arythmiques en obscurité constante validant le rôle de Npas2 comme analogue fonctionnel de Clock dans les NSC, et élargissant un peu plus la liste de redondances fonctionnelles dans l’horloge. Une chose semble inévitable : ce double knock-out va présenter une large palette de déficits et pathologies. Il sera très intéressant de voir si ces souris phénocopient le knock-out
Bmal1 : cela devrait théoriquement être le cas puisque les deux souches présentent une perturbation de l’horloge au même niveau de régulation, celui de l’activation transcriptionnelle centrale. Dans une troisième étude [13], ces mêmes auteurs se sont intéressé à un possible rôle différentiel, selon le tissu, des gènes Clock et Npas2. Comme nous l’avons vu, chez les animaux knock-out pour Clock, bien qu’assez sévèrement perturbés, les rythmes moléculaires persistent dans les NSC et dans le foie. Qu’en est-il dans un contexte ex-vivo ? Pour cette étude, les auteurs ont croisé les différentes lignées knock-out (Clock, Npas2 et Clock/Npas2) avec une lignée knock-in qui exprime une protéine de fusion PER2 ::Luciférase [
31]. Cela permet, en ajoutant de la luciférine dans le milieu de culture, une étude en temps réel du fonctionnement de l’horloge dans des explants. Comme attendu, la rythmicité persiste (aussi bien dans les NSC que dans le foie) en l’absence de Npas2 et est abolie pour les deux tissus dans les double knock-out
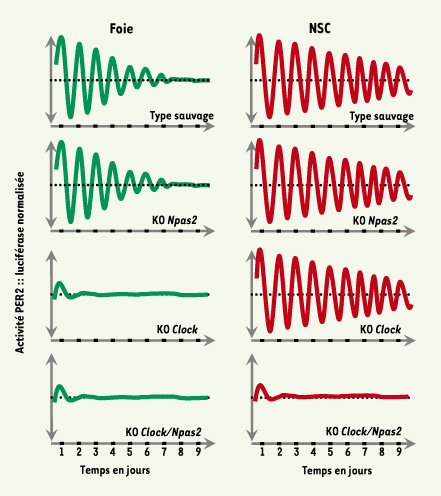
Clock/Npas2. En revanche, la rythmicité est soutenue en l’absence de Clock, mais seulement dans les NSC ; les explants de foie sont totalement arythmiques, et il est impossible de démarrer l’horloge hépatique (et ce en dépit de la forte compensation notée plus haut, Figure 4).  | Figure 4.
Conséquences de la délétion des gènes Clock et Npas2 sur la rythmicité. Lorsque des explants de NSC ou de foie sont placés en culture, ils présentent une oscillation circadienne qui persiste durant de nombreux cycles, en particulier dans les NSC ( voir aussi Figure 1). L’ablation de Npas2 a peu ou pas d’effet sur la rythmicité alors que l’ablation de Clock supprime la rythmicité du foie mais pas des NSC. Le double KO Clock/ Npas2 conduit à une arythmie totale pour les deux tissus ( voir texte) (adapté d’après [ 12, 13]). |
Au moins deux interprétations, qui ne sont pas mutuellement exclusives, sont envisageables : soit les mécanismes moléculaires sont différents dans le foie et les NSC et Clock s’avère indispensable au mécanisme dans le foie (mais pas dans les NSC), soit le maintien de la rythmicité dans les NSC en l’absence de Clock est une conséquence de la nature du tissu.
-
En faveur de la première hypothèse, une littérature déjà abondante indique que les gènes impliqués dans le fonctionnement des horloges présentent des profils d’expression différents. C’est notamment le cas pour les Ror [
32], les gènes Per1/2 [
33,
34] ou bien encore Timeless [33]. Par ailleurs, un composant peut être exprimé dans de multiples tissus mais en quantités différentes, comme c’est le cas pour les Rev-erb et les Ror [32,
35]. Chaque tissu possède donc une combinaison unique - qualitative et quantitative - de gènes de l’horloge (suivant une définition plus permissive que celle discutée précédemment). Cela affecte nécessairement les différentes caractéristiques de l’horloge (période, amplitude, sensibilité aux signaux synchroniseurs…). C’est peut-être l’une des raisons pour lesquelles les explants issus de différents tissus présentent des périodes différentes [31].
-
En faveur de la seconde hypothèse (nature du tissu), il est important de rappeler que les NSC forment une structure compacte, constituée de neurones établissant un réseau complexe et extrêmement dense d’interconnexions synaptiques et chimiques (gap-junctions) et d’interactions avec des cellules gliales elles aussi rythmiques [
36]. Il est concevable que ces propriétés histologiques uniques confèrent aux NSC une robustesse particulière, assortie d’une résistance accrue à diverses perturbations, notamment génétiques (Figure 1). Cela peut aussi expliquer la relative insensibilité des NSC à de nombreux synchroniseurs environnementaux et internes (hormis la lumière, peu de facteurs affectent la phase des NSC). Le couplage entre oscillateurs permet certainement d’amplifier le signal (par un phénomène de résonance) et d’assurer un fonctionnement synchronisé. Néanmoins, la présence de neurones oscillant en antiphase ainsi que celle de neurones apparemment insensibles à certaines mutations suggèrent également que tous les neurones des NSC ne sont pas identiques et que l’ensemble comprend diverses sous-unités fonctionnelles dont les rôles demeurent inconnus [
37–
39] (pour revues, voir [2,
40,
41]). Confirmant cette hypothèse et précisant un peu plus d’éventuelles différences entre les cellules des NSC, Liu et al. [39] ont démontré que des explants de NSC de souris invalidées pour Per1 ou Cry1 demeurent rythmiques alors que la rythmicité est perdue dans des explants de poumon. Observation encore plus intéressante, lorsque les cellules des NSC des souris knock-out
Per1 ou Cry1 sont dissociées, une très large majorité d’entre elles s’avère alors arythmique.
|
L’ensemble de ces résultats suggère que la robustesse rythmique des NSC est une conséquence de la redondance génétique dans les mécanismes moléculaires de l’horloge d’une part et d’un couplage particulièrement fort entre ses cellules d’autre part, résultante de son organisation en réseau. |
Merci à Nicolas Cermakian (Laboratoire de Chronobiologie Moléculaire, Centre de Recherche de l’Hôpital Douglas, Université McGill, Montréal, Québec), Mireille Masson-Pévet, Valérie Simonneaux et Étienne Challet (Département de Neurobiologie des Rythmes, UMR 7168/LC2, Université Louis Pasteur, Strasbourg, France) pour leurs commentaires sur cet article.
|
Footnotes |
1. Ko CH, Takahashi JS. Molecular components of the mammalian circadian clock. Hum Mol Genet 2006; 15 (suppl 2) : R271–7. 2. Dardente H, Cermakian N. Les noyaux suprachiasmatiques : une horloge circadienne composée. Med Sci (Paris) 2005; 21 : 66–72. 3. Dardente H, Cermakian N. Molecular circadian rhythms in central and peripheral clocks in mammals. Chronobiol Int 2007; 24 : 195–213. 4. Kalsbeek A, Palm IF, La Fleur SE, et al. SCN outputs and the hypothalamic balance of life. J Biol Rhythms 2006; 21 : 458–69. 5. Pando MP, Morse D, Cermakian N, et al. Phenotypic rescue of a peripheral clock genetic defect via SCN hierarchical dominance. Cell 2002; 110 : 107–17. 6. Guo H, McKinley Brewer J, Lehman MN, et al. Expression in hamster peripheral organs : effects of transplanting the pacemaker. J Neurosci 2006; 26 : 6406–12. 7. McDearmon EL, Patel KN, Ko CH, et al. Dissecting the functions of the mammalian clock protein BMAL1 by tissue-specific rescue in mice. Science 2006; 314 : 1304–8. 8. Hong HK, Chong JL, Song W, et al. Inducible and reversible Clock gene expression in brain using the tTA system for the study of circadian behavior. PLoS Genet 2007; 3 : e33. 9. Kornmann B, Schaad O, Bujard H, et al. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol 2007; 5 : e34. 10. Storch KF, Paz C, Signorovitch J, et al. Intrinsic circadian clock of the mammalian retina : importance for retinal processing of visual information. Cell 2007; 130 : 730–41. 11. Debruyne JP, Noton E, Lambert CM, et al. A clock shock : mouse CLOCK is not required for circadian oscillator function. Neuron 2006; 50 : 465–77. 12. DeBruyne JP, Weaver DR, Reppert SM. CLOCK and NPAS2 have overlapping roles in the suprachiasmatic circadian clock. Nat Neurosci 2007; 10 : 543–5. 13. DeBruyne JP, Weaver DR, Reppert SM. Peripheral circadian oscillators require CLOCK. Curr Biol 2007; 17 : R538–9. 14. Yamaguchi S, Mitsui S, Miyake S, et al. The 5’ upstream region of mPer1 gene contains two promoters and is responsible for circadian oscillation. Curr Biol 2000; 10 : 873–6. 15. Triqueneaux G, Thenot S, Kakizawa T, et al. The orphan receptor Rev-erbalpha gene is a target of the circadian clock pacemaker. J Mol Endocrinol 2004; 33 : 585–608. 16. Harms E, Kivimae S, Young MW, et al. Posttranscriptional and posttranslational regulation of clock genes. J Biol Rhythms 2004; 19 : 361–73. 17. Cheng HY, Papp JW, Varlamova O, et al. microRNA modulation of circadian-clock period and entrainment. Neuron 2007; 54 : 813–29. 18. Kojima S, Matsumoto K, Hirose M, et al. LARK activates posttranscriptional expression of an essential mammalian clock protein, PERIOD1. Proc Natl Acad Sci USA 2007; 104 : 1859–64. 19. Green CB, Douris N, Kojima S, et al. Loss of Nocturnin, a circadian deadenylase, confers resistance to hepatic steatosis and diet-induced obesity. Proc Natl Acad Sci USA 2007; 104 : 9888–93. 20. Gatfield D, Schibler U. Physiology. Proteasomes keep the circadian clock ticking. Science 2007; 316 : 1135–6. 21. Duffield GE. DNA microarray analyses of circadian timing : the genomic basis of biological time. J Neuroendocrinol 2003; 15 : 991–1002. 22. Takahashi JS. Finding new clock components : past and future. J Biol Rhythms 2004; 19 : 339–47. 23. Kondratov RV, Kondratova AA, Gorbacheva VY, et al. Early aging and age-related pathologies in mice deficient in BMAL1, the core componentof the circadian clock. Genes Dev 2006; 20 : 1868–73. 24. Xu Y, Toh KL, Jones CR, et al. Modeling of a human circadian mutation yields insights into clock regulation by PER2. Cell 2007; 128 : 59–70. 25. Gekakis N, Staknis D, Nguyen HB, et al. Role of the CLOCK protein in the mammalian circadian mechanism. Science 1998; 280 : 1564–9. 26. Lowrey PL, Takahashi JS. Mammalian circadian biology : elucidating genome-wide levels of temporal organization. Annu Rev Genomics Hum Genet 2004; 5 : 407–41. 27. Dudley CA, Erbel-Sieler C, Estill SJ, et al. Altered patterns of sleep and behavioral adaptability in NPAS2-deficient mice. Science 2003; 301 : 379–83. 28. Reick M, Garcia JA, Dudley C, et al. NPAS2 : an analog of clock operative in the mammalian forebrain. Science 2001; 293 : 506–9. 29. McNamara P, Seo SP, Rudic RD, et al. Regulation of CLOCK and MOP4 by nuclear hormone receptors in the vasculature : a humoral mechanism to reset a peripheral clock. Cell 2001; 105 : 877–89. 30. Shearman LP, Zylka MJ, Reppert SM, et al. Expression of basic helix-loop-helix/PAS genes in the mouse suprachiasmatic nucleus. Neuroscience 1999; 89 : 387–97. 31. Yoo SH, Yamazaki S, Lowrey PL, et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc Natl Acad Sci USA 2004; 101 : 5339–46. 32. Guillaumond F, Dardente H, Giguere V, et al. Differential control of Bmal1 circadian transcription by REV-ERB and ROR nuclear receptors. J Biol Rhythms 2005; 20 : 391–403. 33. Zylka MJ, Shearman LP, Levine JD, et al. Molecular analysis of mammalian timeless. Neuron 1998; 21 : 1115–22. 34. Feillet CA, Mendoza J, Albrecht U, et al. Forebrain oscillators ticking with different clock hands. Mol Cell Neurosci 2008; 37 : 209–21. 35. Yang X, Downes M, Yu RT, et al. Nuclear receptor expression links the circadian clock to metabolism. Cell 2006; 126 : 801–10. 36. Prolo LM, Takahashi JS, Herzog ED. Circadian rhythm generation and entrainment in astrocytes. J Neurosci 2005; 25 : 404–8. 37. Low-Zeddies SS, Takahashi JS. Chimera analysis of the Clock mutation in mice shows that complex cellular integration determines circadian behavior. Cell 2001; 105 : 25–42. 38. Herzog ED, Aton SJ, Numano R, et al. Temporal precision in the mammalian circadian system : a reliable clock from less reliable neurons. J Biol Rhythms 2004; 19 : 35–46. 39. Liu AC, Welsh DK, Ko CH, et al. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell 2007; 129 : 605–16. 40. Maywood ES, O’Neill JS, Chesham JE, et al. The circadian clockwork of the suprachiasmatic nuclei : analysis of a cellular oscillator that drives endocrine rhythms. Endocrinology 2007; 148 : 5624–34. 41. Aton SJ, Herzog ED. Come together, right… now : synchronization of rhythms in a mammalian circadian clock. Neuron 2005; 48 : 531–4. 42. Teboul M, Delaunay F. Le récepteur nucléaire orphelin Rev-erba oscille entre répression et activation. Med Sci (Paris) 2003; 19 : 411–3. 43. Kornmann B. L’horloge circadienne centrale et les horloges périphériques : décentralisation et contrôle hiérarchique. Med Sci (Paris) 2007; 23 : 349–50. |