| |
| Med Sci (Paris). 2008 May; 24(5): 541–544. Published online 2008 May 15. doi: 10.1051/medsci/2008245541.Succès de la première équipe française lors de la competition iGEM de biologie synthétique David Bikard,2* François Képès,1* and au nom de l’équipe iGEM Paris* 1Programme d’Épigénomique, Genopole®, CNRS, Université d’Évry, 523, Terrasses de l’Agora, 91034 Évry, France 2Département de Génomes et Génétique, Institut Pasteur, 25, rue du Docteur Roux, 75724 Paris MeSH keywords: Bactéries, Biologie, France, Modèles biologiques, Modèles génétiques |
Un concours de « legos » génétiques Les 3 et 4 novembre 2007 s’est tenu au MIT (Massachusetts Institute of Technology, États-Unis) la rencontre finale de iGEM (international Genetically Engineered Machine), la compétition étudiante internationale de biologie synthétique. Le but de la compétition est de concevoir rationnellement et de construire des systèmes biologiques remplissant une fonction spécifiée en assemblant, à la manière d’ingénieurs, des composants standard d’ADN appelés « Bio-briques » [
1](→).

(→) voir m/s n° 5, mai 2007, p. 551
La compétition iGEM est née en 2003 au MIT ; elle est devenue internationale en 2005. Le nombre d’équipes en compétition ne cesse d’augmenter d’année en année, au point que des « succursales » sont envisagées en Europe et en Asie. L’édition de cette année, qui a réuni 54 équipes venant du monde entier, est de loin la plus importante à ce jour (Figure 1). C’est devant un jury international prestigieux et un public d’un demi-millier de participants que chaque équipe a présenté son projet mené durant 3 mois au cours de l’été 2007.
 | Figure 1.
Les équipes iGEM 2007.
|
Cette année, des projets ambitieux ont vu le jour, comme le « Bactoblood » de Berkeley, des bactéries modifiées pour produire un substitut sanguin, ou encore « Vitrotrap » de Ljubljana, une ingénieuse approche de thérapie génique contre le Sida. Il faut bien entendu mentionner l’équipe de Pékin, victorieuse de cette édition grâce à son « interrupteur-poussoir » et son « compteur ». Le premier consiste en un petit réseau de molécules jouant le rôle d’un interrupteur permettant de passer d’un état stable d’expression génétique à un autre et inversement grâce à un unique signal. Le second tente de faire compter aux bactéries le nombre de fois qu’un signal a été transmis. Ces deux modules ont pour but de mettre en place une « chaîne de montage » bactérienne. Ils permettent une différenciation spatiale et temporelle à partir de conditions homogènes afin d’établir une division du travail. Pour la première fois une équipe française a participé et brillé en se voyant nominée parmi les 6 finalistes pour le grand prix, et en remportant le prix de la recherche fondamentale auquel concourraient aussi des universités comme Harvard, Caltech, et Cambridge. Bien d’autres projets mériteraient d’être mentionnés, mais il est aisé d’en prendre connaissance sur le « wiki » de la compétition (www.igem.org). En effet, la description des projets est rassemblée sur un site internet dédié qui joue un rôle important dans le processus d’évaluation. La communauté de biologie synthétique se veut ouverte. Partageons nos résultats, échangeons nos idées mais aussi nos constructions, car un point fort de cette nouvelle approche est de tenter une standardisation des méthodes de construction. Ainsi tout composant en ADN peut être simplement assemblé avec n’importe quel autre, facilitant les échanges et permettant un processus incrémental de construction. Au début de l’été, chaque équipe reçoit l’ensemble des « Bio-briques » du « Répertoire des composants biologiques standard » situé au MIT. Ces briques sont utilisées par les équipes pour construire des dispositifs, eux-mêmes assemblés en systèmes. Les équipes doivent en retour envoyer au « Répertoire » tous leurs assemblages ainsi que toute nouvelle brique construite. Ce « Répertoire » grandit pour le moment au rythme de la compétition et compte déjà plus d’un millier d’entrées. Dans un avenir proche, l’équipe du MIT qui s’en occupe espère bien en étendre la portée au-delà d’iGEM. |
L’équipe francilienne « iGEM Paris » L’initiative a été lancée par deux étudiants du master « approches interdisciplinaires du vivant » (AIV : ENS, Paris7, Paris5), David Bikard et Eimad Shotar. Ce master est hébergé par le Centre de recherche interdisciplinaire (CRI, faculté de Médecine Paris Descartes). Le CRI offre la liberté aux étudiants d’aborder de nombreux sujets : ceux-ci sont notamment incités à créer des clubs scientifiques sur des thèmes qui les intéressent. Les étudiants sont libres d’organiser leur club comme ils le souhaitent : étude d’articles, de livres, invitation de chercheurs, discussions ou débats, etc. C’est ainsi qu’un club de biologie synthétique est né au sein du CRI. Celui-ci a vite vu son affluence augmenter et le lien a rapidement été établi avec le réseau des chercheurs français s’intéressant à la biologie synthétique. Devant le succès du club, l’initiative d’organiser une participation francilienne à la compétition iGEM a naturellement été prise. L’équipe a alors organisé une recherche de sponsors, et a recruté des étudiants de toutes disciplines et formations dans les universités et écoles d’ingénieurs franciliennes. L’interdisciplinarité est en effet au cœur de la démarche de la biologie synthétique, qui allie dans son périmètre minimal l’ingénierie à la modélisation mathématique et à la génétique moléculaire. L’équipe de Berkeley comprenait même cette année un juriste afin de réfléchir à la brevetabilité du projet. Ainsi, les étudiants de l’équipe francilienne se répartissaient en 2 ingénieurs, 3 médecins, 2 bio-informaticiens, 1 biophysicien et 2 biologistes. Les étudiants du niveau L3 à M2 ont ainsi eu la chance de mener leur propre recherche selon des objectifs fixés par eux-mêmes. Tout au long du projet la prise de décision a été faite par les étudiants avec les conseils avisés d’une équipe d’encadrants tout aussi interdisciplinaire que celle des étudiants. Plusieurs sponsors ont participé (fondation Bettencourt Schueller, Commission Européenne, ambassade de France aux États-Unis, Sofinnova), et l’équipe a été accueillie par le laboratoire TaMaRa (Inserm U571). La somme recueillie a servi à la réalisation du projet au cours de l’été, et a permis à la totalité de l’équipe de partir à Boston pour la rencontre finale. Celle-ci s’est déroulée dans une excellente ambiance générale, et a été l’occasion de nombreuses rencontres avec les étudiants des autres équipes, mais surtout avec les chercheurs à la pointe de la discipline. C’était donc l’occasion de faire ses premiers pas dans une communauté scientifique à laquelle nombre des membres de l’équipe ont maintenant la ferme intention de participer. |
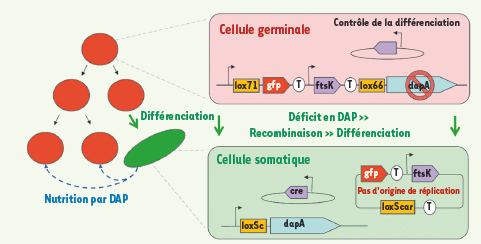
Cette année, la compétition iGEM a été divisée en 5 parcours à thème : Médecine et Santé, Environnement, Énergie, Traitement de l’Information et Recherche Fondamentale. C’est dans cette dernière catégorie que l’équipe francilienne a concouru avec pour projet de construire une bactérie multicellulaire synthétique. L’organisme imaginé est multicellulaire en ceci qu’il est composé de deux types cellulaires interdépendants (Figure 2). L’un, dédié à la reproduction (nommé lignée germinale), ne peut survivre qu’en présence du second type (la lignée somatique) qui provient par différenciation du premier. Cette dépendance vient de ce que les cellules germinales requièrent du diaminopimélate (DAP) pour la synthèse de leur paroi. Or le DAP leur est fourni par les cellules somatiques capables, elles, de le surproduire. Les cellules somatiques sont en revanche incapables de se diviser. Leur existence dépend donc entièrement de la différenciation de cellules de la lignée germinale. Ainsi l’interdépendance des deux types cellulaires est assurée.
 | Figure 2.
Principe de fonctionnement de l’organisme synthétique. Les cellules germinales (en rouge) expriment le gène ftsK essentiel à la division, mais n’expriment pas le gène dapA ce qui les rend dépendantes d’une source externe de DAP. L’expression de la recombinase CRE induit la différenciation par recombinaison entre les boites lox. Les cellules somatiques (en vert) n’expriment plus le gène ftsK et deviennent incapables de se diviser. En revanche, elles expriment dapA et produisent donc du DAP qui permet de nourrir les cellules germinales. |
Un défi pour la biologie synthétique : compartimenter les réactions d’un système Pourquoi une bactérie multicellulaire intéresse-t-elle la communauté de biologie synthétique ? Plusieurs utilisations prometteuses d’un tel organisme peuvent être envisagées. Un premier aspect est celui de la réalisation de systèmes biologiques complexes. Il peut en effet être utile de compartimenter des sous-systèmes biochimiques dont l’interaction doit être évitée. On aborde ici un des défis de l’application des notions d’ingénierie à la biologie. La démarche de biologie synthétique veut que l’on s’intéresse dans un premier temps au système global en faisant abstraction du niveau de base qui est le choix des composants et de la séquence d’ADN [
2]. Ainsi, un système va d’abord être construit en termes logiques. Souvent des capteurs (de pH, température, molécule particulière…) fournissent un signal d’entrée qui est traité par un réseau de portes logiques débouchant sur une réponse appropriée. De nombreuses portes logiques (voir Encadré) ont déjà été construites par différentes équipes. Certaines portes simples, comme le « NON » (ou inverseur), ont pu servir à la construction par exemple d’un oscillateur génétique [
3]. D’autres plus complexes (NON-ET, NON-OU…) ont valu à l’équipe de l’Université de Science et Technologie de Chine (USTC) une place en finale cette année. Le parallèle avec les circuits électriques est tentant, cependant il existe une différence fondamentale avec ces derniers. Dans un circuit électrique, on connecte facilement les différents composants de base sans que ceux-ci n’interagissent. La séparation physique des liens (fils, câbles…) permet d’utiliser un messager unique, le courant électrique. Au sein de la cellule, la séparation physique est beaucoup moins évidente, et implique donc l’utilisation d’autant de messagers moléculaires différents qu’il existe de liens dans le câblage. Séparer physiquement différentes réactions d’un système est donc un défi actuel de la biologie synthétique. C’est d’ailleurs un projet de création de compartiments artificiels chez la levure qui a valu également à l’Université de Californie à San Francisco (UCSF) une place en finale.

Le projet français : une bactérie « multicellulaire » Le projet français apporte une réponse différente à ce problème en permettant d’utiliser différents types cellulaires comme compartiments distincts. Au-delà, l’intérêt principal de l’organisme imaginé par les étudiants franciliens réside dans la possibilité de découpler l’expression d’un gène étranger ou transgène de la reproduction de l’organisme. Il ouvre ainsi la voie vers la production de composés toxiques pour la division cellulaire. L’expression de transgènes par des bactéries en bioréacteurs est devenue une méthode classique de production de certaines molécules (comme l’insuline) [
4]. Cependant dans bien des cas, la synthèse du composé souhaité est toxique pour la bactérie. Cela limite, de fait, le rendement possible car on doit trouver un compromis entre la croissance de la bactérie et la production. En revanche, dans ce nouvel organisme bi-cellulaire, si seules les cellules « somatiques » expriment le transgène, alors on doit en principe pouvoir atteindre dans ces cellules des concentrations bien supérieures à ce qu’il est possible d’atteindre dans les systèmes d’expression classiques. Certes existe toujours un équilibre entre la croissance et la production du composé toxique. En effet ce composé ne doit ni empêcher les cellules somatiques de nourrir les cellules germinales, ni empoisonner ces dernières en diffusant trop aisément. Mais cet équilibre est déplacé vers une plus forte production en comparaison avec un système d’expression classique. Une autre application consisterait à utiliser les cellules somatiques pour exprimer un transgène en dehors du laboratoire sans risque de fuite dans l’environnement. On peut en effet facilement induire la différenciation de toutes les cellules de la bactérie multicellulaire en cellules somatiques, incapables de proliférer. En revanche, elles continuent à grandir durant plusieurs heures en formant de longs filaments. Une application potentielle serait de faire stocker aux cellules somatiques des triglycérides dans des corps d’inclusion. Ces cellules pourraient être ingérées pendant le repas et absorberaient à notre place la graisse des aliments. Ce projet nommé « Diet Coli » (postiche du « Diet Cola ») est réalisable par expression de l’enzyme DGAT (diacylglycerol acyltransferase). L’équipe a montré que l’expression de la DGAT de Acinetobacter ADP1 [
5] par E. coli induit en effet la synthèse de triglycérides, dans des proportions encore augmentées si des acides gras sont présents dans le milieu. La conception de la bactérie multicellulaire synthétique a fait l’objet d’une étude théorique et expérimentale poussée. Cinq modèles mathématiques répondant à des questions différentes ont été réalisés afin de déterminer les paramètres critiques à prendre en compte lors de la construction (Figure 3). Ainsi, la fréquence de différenciation est un paramètre clé. Au-delà de 50 % de différenciation par génération, chaque cellule germinale donnera après division au mieux une autre cellule germinale, et la taille de la population ne pourra que diminuer. Au contraire, si le taux de différenciation est trop faible, trop peu de cellules somatiques seront présentes pour nourrir les cellules germinales et leur permettre de pousser. Il a également été démontré qu’un rétrocontrôle de la concentration du DAP sur le taux de différenciation pourrait grandement améliorer la robustesse du système. Les modèles ont ainsi permis de définir la gamme de viabilité du système pour certains paramètres. Ces paramètres ont ensuite été mesurés expérimentalement afin de vérifier si le « design » et les composants choisis s’y conformaient bien. Durant l’été l’équipe a conçu le système, l’a validé par modélisation, et a apporté la preuve de principe du fonctionnement des sous-systèmes. La réalisation du système complet est en cours de finition.
 | Figure 3.
Simulation spatiale et stochastique de la croissance de l’organisme synthétique. Le langage de programmation à base de règles « MGS » [
6] a été utilisé. Les cellules germinales (en rouge) se divisent et se différencient en cellules somatiques (en vert). Ces dernières ne se divisent plus mais fournissent du DAP aux cellules germinales, assurant leur croissance. |
Pour l’équipe entière, iGEM s’est révélée être une aventure pédagogique extrêmement formatrice, où la bonne humeur se mêle à l’émulation scientifique, l’envie de voir son projet réalisé, et éventuellement la satisfaction d’une reconnaissance internationale. Mais l’aventure ne s’arrête pas là. Les étudiants de l’équipe 2007 comptent bien s’investir cette année encore dans le club de biologie synthétique et dans la formation d’une équipe iGEM Paris pour 2008.

|
1. Peccoud J, Coulombel L. Une compétition de biologie synthétique ou comment créer l’« eau de E. coli » et les nano-barbies. Med Sci (Paris) 2007; 23 : 551–2. 2. Endy D. Foundations for engineering biology. Nature 2005; 438 : 449–53. 3. Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature 2000; 403 : 335–8. 4. Khosla C, Keasling JD. Metabolic engineering for drug discovery and development. Nat Rev Drug Discovery 2003; 2 : 1019–25. 5. Stoveken T, Kalscheuer R, Malkus U, et al. The wax ester synthase/acyl coenzyme A:diacylglycerol acyltransferase from Acinetobacter sp. strain ADP1: characterization of a novel type of acyltransferase. J Bacteriol 2005; 187 : 1369–76. 6. Michel O, Gavitto JL, Cohen J. MGS : transformer des collections complexes pour la simulation en biologie. Journées Francophones des Langages Applicatifs 2002; JFLA02. |