| |
| Med Sci (Paris). 2008 May; 24(5): 545–547. Published online 2008 May 15. doi: 10.1051/medsci/2008245545.Nouveaux outils de cartographie génétique et génomique pour les études des traits complexes chez le rat Dominique Gauguier* and Marie-Thérèse Bihoreau Wellcome Trust Centre for Human Genetics, Université d’Oxford, Oxford OX3 7BN, Royaume-Uni MeSH keywords: Animaux, Cartographie chromosomique, Marqueurs génétiques, Rats |
Le rat de laboratoire (Rattus Norvegicus) est un modèle clé dans des domaines variés de physiologie, pharmacologie, toxicologie et nutrition, et plus récemment dans les études génétiques et génomiques. Plus de 500 souches isogéniques de rat ont été développées et des recherches actives sont en cours dans des modèles de maladies humaines fréquentes, dont le diabète de type 2, l’obésité, l’asthme, l’hypertension, les maladies auto-immunes et les épilepsies [
1]. La raison pour laquelle les généticiens se sont intéressés au rat comme modèle d’étude tient à la facilité d’effectuer des tests physiologiques minutieux et des prélèvements sanguins volumineux et répétés, qui sont nécessaires pour quantifier des phénotypes représentatifs des traits complexes humains. Les progrès dans la production d’outils génétiques et génomiques chez le rat résultent d’efforts importants principalement au Royaume-Uni, aux États-Unis, au Japon, et en Allemagne. L’amélioration des méthodes de génotypage chez le rat, qui sont jusqu’à maintenant fondées sur l’utilisation de marqueurs génétiques microsatellites, reste l’élément limitant des études génétiques chez le rat. La caractérisation génotypique que vient de publier un consortium de laboratoires européens de plus de 20 000 polymorphismes nucléotidiques (single nucleotide polymorphisms, SNP) chez 167 souches isogéniques de rat, et des hybrides utilisés pour la cartographie génétique de traits complexes [
2], représente le progrès le plus significatif pour les études génétiques dans cette espèce depuis le séquençage du génome du rat de la souche Brown-Norway (BN) en 2004 [
3,
4]. |
Identification de marqueurs SNP dans le génome du rat Environ 3 millions de SNP ont été identifiés par comparaison de la séquence génomique du rat BN avec des données de séquençage du rat Sprague-Dawley et, pour augmenter la représentativité génétique de ces marqueurs, des souches de rats Salt Sensitive (SS), Wistar-Kyoto (WKY), Goto-Kakizaki (GK), Spontaneously Hypertensive Stroke Prone (SHRSP) et Fisher (F344). Sur la base de leur position dans le génome, ces nouveaux marqueurs renseignent sur des polymorphismes génétiques espacés d’environ 800 paires de bases. Ils sont appliqués au génotypage à haut débit et vont donc faciliter les études génétiques chez des hybrides issus de croisements classiques F2 ou backcross, mais également dans d’autres systèmes expérimentaux, dont le stock hétérogène qui est une mosaïque génétique de huit souches (ACI, BN, BUF, F344, M520, MR, WKY, WN) et qui permet la cartographie fine directe de traits quantitatifs [
5]. |
Études phylogénétiques des souches de rats isogéniques De manière à produire une collection de marqueurs de qualité dont le polymorphisme a été vérifié, et adaptée à des études génotypiques d’hybrides quelle que soit la combinaison de souches choisies, la détermination des génotypes de 20 283 SNP a été entreprise sur 167 souches isogéniques. L’identification des marqueurs polymorphes entre les souches de rats utilisées pour le typage des SNP, ainsi que la visualisation des blocs de génotypes conservés ou divergents, sont accessibles sur http://www.well.ox.ac.uk/rat_mapping_resources/SNPbased_maps.html. Les résultats du génotypage des marqueurs SNP indiquent l’éloignement phylogénétique de la souche de rats BN et l’articulation de la généalogie des autres souches de rats autour de onze groupes. En outre, l’hétérogénéité génétique importante des colonies de WKY, SHR et SHRSP (11-19 %), déjà démontrée par l’étude du polymorphisme de marqueurs microsatellites [
6], indique que ces souches, provenant d’un stock de rats Wistar, ont été distribuées avant que l’homogénéité génétique ait été atteinte. Cette hétérogénéité joue vraisemblablement un rôle majeur dans la réplication partielle des résultats des études de liaison génétique utilisant ces modèles. En revanche, les colonies d’autres souches maintenues indépendamment depuis de nombreuses générations, comme les souches BN ou GK, montrent, avec un taux de polymorphisme de moins de 1 %, une homogénéité génétique remarquable. |
Application des marqueurs SNP à la cartographie génétique des traits complexes L’évaluation de la qualité de cette série de SNP a été poursuivie pour vérifier que les polymorphismes sélectionnés ne sont pas des artefacts de séquence, que les technologies de génotypage de SNP permettent de discriminer hétérozygotes et homozygotes, et que la ségrégation mendélienne des allèles chez des hybrides est respectée. L’analyse génotypique a été entreprise chez des hybrides F2 d’un croisement entre rats GK et BN utilisé pour la cartographie de locus de contrôle de phénotypes représentatifs du diabète [
7,
8] et de traits métabonomiques [
9,
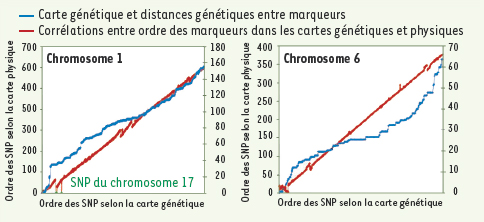
10]. Cette étude a été complémentée par le génotypage de deux collections de souches isogéniques recombinantes issues des rats SHR et BN (n = 31) et de rats F344 et Long-Evans (n = 34). Ces trois panels permettent la cartographie, et par conséquent la vérification de la qualité de plus de 80 % des 20 283 marqueurs SNP utilisés. La construction des cartes génétiques de SNP dans les rats issus du croisement F2 a été effectuée sans connaissance a priori de la position génomique des marqueurs. De manière générale, les cartes génétiques et physiques s’alignent et soulignent des variations de fréquence de recombinaison génétique le long des chromosomes (Figure 1). En revanche, la mise en évidence de régions où les cartes génétiques et physiques divergent suggère des anomalies dans l’assemblage de la séquence génomique du rat. Un exemple est la localisation dans la carte génétique du chromosome 1 de marqueurs SNP isolés d’une région télomérique du chromosome 17 (Figure 1). Les résultats de la cartographie génétique des marqueurs SNP génotypés dans les trois cohortes de rats indiquent l’existence d’autres erreurs dans l’assemblage de la séquence génomique sur les chromosomes 2, 4, 11, 12 et 14.
 | Figure 1.
Exemples d’alignements de cartes génétiques construites avec les marqueurs SNP et de cartes physiques résultant de l’assemblage du génome du rat. Les cartes génétiques ont été construites avec le programme JoinMap [ 5] en utilisant les données génotypiques des marqueurs SNP dans le croisement F2 GKxBN [ 6] sans connaissance à priori de leur localisation dans le génome. |
|
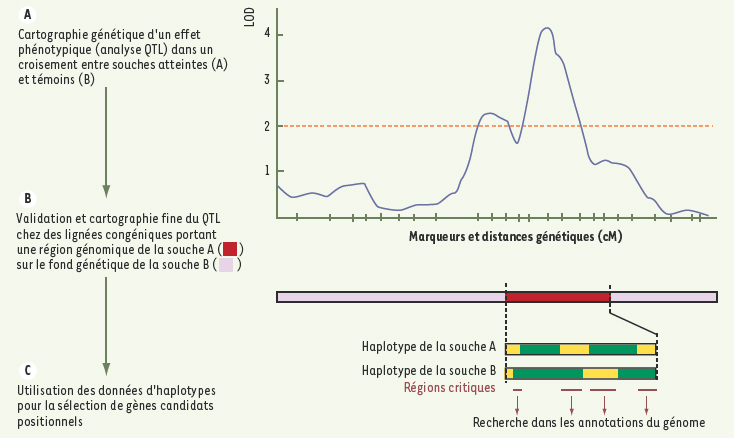
Définition d’haplotypes et identification de gènes de contrôle de traits complexes Les marqueurs SNP vont trouver une application importante dans le clonage de gènes de contrôle de traits quantitatifs (quantitative trait locus, QTL) dans des régions du génome identifiées dans une population hybride par étude de liaison génétique (Figure 2A) qui, par définition, associe sur des critères essentiellement statistiques un trait phénotypique avec un intervalle du génome. L’utilisation de souches congéniques, qui portent une région chromosomique couvrant tout ou partie du QTL transférée d’une souche sur le fond génétique d’une autre souche, permet de progresser vers la validation biologique de ces locus et la cartographie fine du ou des gènes responsables (Figure 2B) [
11]. La connaissance des génotypes des marqueurs SNP contigus sur la région du QTL et dans l’intervalle congénique, permet d’orienter la recherche de ces gènes vers des segments où les haplotypes entre les souches parentales sont divergents (Figure 2C). L’importance de cette stratégie est illustrée par les résultats de l’étude génétique du gène codant pour une protéine de contrôle du rythme circadien (BMAL1) [
12]. Cette étude a démontré le rôle de polymorphismes dans ce gène dans la régulation de son expression chez les rats congéniques dérivés des souches SHR et WKY pour un QTL d’hypertension, et dans la prédisposition à l’hypertension et au diabète chez l’homme [
13].
 | Figure 2.
Application des haplotypes déterminés par cartographie de SNP pour identifier les gènes de contrôle de traits complexes dans des souches congéniques.
|
|
La production de marqueurs SNP chez le rat et la caractérisation génotypique dans de nombreuses souches, fournissent de nouveaux outils essentiels pour corréler polymorphisme génétique et variabilité phénotypique [
14] et faciliter la cartographie génétique des traits complexes. Avec l’application de technologies de génotypage à haut débit, ces marqueurs vont promouvoir de nouvelles études génétiques de traits phénotypiques représentatifs de traits physiopathologiques humains et accélérer l’identification des variants génétiques responsables. |
Wellcome Functional Genomics Initiative CFG (Cardiovascular Functional Genomics ; 066780), FGENTCARD (Functional genomic diagnostic tools for coronary artery disease ; LSHG-CT-2006-037683) de la Communauté Européenne. D. Gauguier est Professeur de Génétique des Mammifères à l’Université d’Oxford et financé par un Senior Fellowship du Wellcome Trust (057733).
|
1. Gauguier D. The rat as a model physiological system. In : Jorde LB, Little P, Dunn M, Subramaniam S, eds. Encyclopedia of genetics. London, UK : Wiley, 2006 : 1154–71. 2. Saar K, Beck A, Bihoreau MT, et al. SNP and haplotype mapping for genetic analysis in the rat. Nat Genet 2008 (sous presse). 3. Aitman T, Critser JK, Cuppen E, et al. Progress and prospects in rat genetics: a community view. Nat Genet 2008 (sous presse). 4. Gibbs RA, Weinstock GM, Metzker ML, et al. Genome sequence of the Brown Norway rat yields insights into mammalion evolution. Nature 2004; 428 : 493–521 5. Valdar W, Solberg LC, Gauguier D, et al. Genome-wide genetic association of complex traits in heterogeneous stock mice. Nat Genet 2006; 38 : 879–87. 6. Bihoreau MT, Gauguier D, Kato N, et al. A linkage map of the rat genome derived from three F2 crosses. Genome Res 1997; 7 : 434–40. 7. Gauguier D, Froguel P, Parent V, et al. Chromosomal mapping of genetic loci associated with non-insulin dependent diabetes in the GK rat. Nat Genet 1996; 12 : 38–43. 8. Argoud K, Wilder SP, McAteer MA, et al. Genetic control of plasma lipid levels in a cross derived from normoglycaemic Brown Norway and spontaneously diabetic Goto-Kakizaki rats. Diabetologia 2006; 49 : 2679–88. 9. Dumas ME, Wilder SP, Bihoreau MT, et al. Direct quantitative trait locus mapping of mammalian metabolic phenotypes in diabetic and normoglycemic rat models. Nat Genet 2007; 39 : 666–72. 10. Gauguier D, Dumas ME. Metabonomique et cartographie génétique. Med Sci (Paris) 2007; 23 : 1091–4 11. Rogner UC, Avner P. Congenic mice: cutting tools for complex immune disorders. Nat Rev Immunol 2003; 3 : 243–52. 12. Dardente H. Redondance génétique et synchronisation cellulaire dans les horloges circadiennes. Med Sci (Paris) 2008; 24 : 270–6. 13. Woon PY, Kaisaki PJ, Braganca J, et al. Aryl hydrocarbon receptor nuclear translocator-like (BMAL1) is associated with susceptibility to hypertension and type 2 diabetes. Proc Natl Acad Sci USA 2007; 104 : 14412–7. 14. Fearnside JF, Dumas ME, Rothwell AR, et al. Phylometabonomic patterns of adaptation to high fat diet feeding in inbred mice. PLoS One 2008; 3 : e1668. |