| |
| Med Sci (Paris). 2008 August; 24(8-9): 700–702. Published online 2008 August 15. doi: 10.1051/medsci/20082489700.Les facteurs de transcription, garants de l’identité cellulaire Camille Du Roure* Friedrich Miescher Institute for Biomedical Research, Novartis Research Foundation, Basel, Suisse MeSH keywords: Lymphocytes B, Division cellulaire, Phénomènes physiologiques cellulaires, Humains, Lymphome B, Plasmocytes, Facteurs de transcription, Transcription génétique |
Toutes les cellules d’un organisme possèdent la même information génétique contenue dans les gènes dont la régulation sélective (activation des gènes caractéristiques d’un type cellulaire et répression des gènes inadéquats) confère aux cellules leur identité [
1]. Cette restriction transcriptionnelle implique une étroite coopération entre des facteurs de transcription, liés à l’ADN, et des facteurs épigénétiques qui modifient physiquement la chromatine. La plupart des mécanismes épigénétiques sont ubiquitaires et c’est par la présence de facteurs de transcription spécifiques que naît leur spécificité. D’ailleurs, chaque type cellulaire possède son propre réseau de facteurs de transcription nécessaires à l’établissement et au maintien de son identité [
2]. Leur dérégulation perturbe cette identité, et est associée au développement de tumeurs chez l’Homme. L’importance des facteurs de transcription est particulièrement bien illustrée dans le système hématopoïétique. Le développement des lymphocytes B offre, en particulier, un modèle de choix pour comprendre leur dynamique et leurs rôles dans la plasticité fonctionnelle des cellules dans un contexte sain et pathologique. |
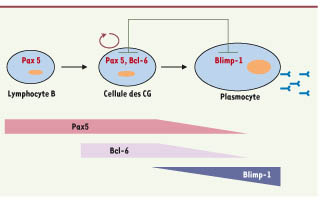
Les différentes étapes de la lymphopoïèse B Les lymphocytes B matures se différencient à partir de cellules souches hématopoïétiques dans la moelle osseuse puis dans la rate où ils attendent d’être activés par l’antigène. Les lymphocytes activés migrent dans les centres germinatifs (CG) où ils entament leur différenciation terminale en plasmocytes. A chacune de ces étapes (lymphocytes B matures, lymphocytes B des CG et plasmocytes), les cellules possèdent différentes fonctions. Par exemple, seules les cellules des CG prolifèrent, et seuls les plasmocytes sécrètent des anticorps. Pour leur permettre d’assurer ces fonctions, de nombreux facteurs de transcription adaptent le programme transcriptionnel des cellules. Trois d’entre eux, intervenant successivement lors de la différenciation, sont notablement importants : Pax5 (Paired box 5), Bcl-6 (B-cell lymphoma 6) et Blimp-1 (B-lymphocyte induced maturation protein 1) (Figure 1). En utilisant ces facteurs de transcription comme exemples, cet article vise à illustrer comment les facteurs de transcription assurent l’établissement et le maintien de l’identité cellulaire, et comment leur dérégulation peut conduire à des tumeurs.
 | Figure 1.
Développement des lymphocytes B en plasmocytes. Les lymphocytes B matures activés par l’antigène entrent dans les centres germinatifs (cellules B des CG) pour se différencier en plasmocytes. Les facteurs de transcription Pax5, Bcl-6 et Blimp-1 interviennent successivement lors de cette différenciation : Pax5 dans les cellules B matures et cellules B des CG, Bcl-6 dans les CG et Blimp-1 dans les plasmocytes. Pax5 et Bcl-6 répriment l’expression de prdm1/Blimp-1, et inversement. Les patrons d’expression de chacun des facteurs de transcription sont représentés dans la partie basse du schéma. |
|
Le subtil équilibre transcriptionnel d’un trio de facteurs de transcription Les souris déficientes pour Pax5 sont dépourvues de lymphocytes B, bien que les précurseurs conservent un potentiel de développement [
13]. En effet, ces progéniteurs sont capables de se différencier vers l’ensemble des lignages hématopoïétiques, à l’exception du lignage B [
3]. Différentes études montrent que pour établir et maintenir l’identité des cellules B, Pax5 non seulement active des gènes importants pour leurs fonctions (comme CD19 et Ebf1), mais aussi, et surtout, réprime des gènes nécessaires au développement d’autres lignages (tel que Notch1 pour la différenciation T [
14]) et ceux inadéquats pour les lymphocytes B [3]. La répression de ces gènes est importante pour préserver l’identité cellulaire et leur expression ectopique peut conduire à une perte de l’identité cellulaire ou à une transdifférenciation. Par exemple, l’expression forcée des facteurs de transcription myéloïdes CEBP α et β dans les lymphocytes B conduit à leur transdifférenciation en macrophages [
4]. Bcl-6, quant à lui, a de nombreux rôles dans les centres germinatifs (CG). Les CG sont des structures de la rate qui accueillent les lymphocytes B activés pour leur permettre de proliférer et de se différencier en plasmocytes. Au cours de cette prolifération, des mécanismes de modification de l’ADN qu’on appelle commutation isotypique et mutations hypersomatiques permettent la production d’anticorps adaptés et de haute affinité [
15]. Ces transformations impliquent à la fois des cassures double brin de l’ADN et des mutations ponctuelles. Bcl-6 permet aux cellules des CG de supporter ce stress génotoxique en réprimant des gènes impliqués dans la réponse au stress (Figure 2 [
5]). Parallèlement, Bcl-6 inhibe l’expression de gènes s’opposant à la prolifération cellulaire (arrêt du cycle cellulaire ou apoptose), et ceux permettant la différenciation plasmocytaire, en particulier prdm1 qui code pour Blimp-1 (Figure 2) [
6,
7]. Il a aussi été montré que prdm1 est réprimé par Pax5 dans les lymphocytes B matures, suggérant que Pax5 contribue, lui aussi, au maintien de l’identité B dans les CG.
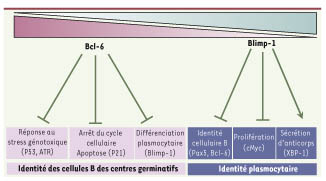
 | Figure 2.
Établissement de l’identité cellulaire par Bcl-6 et par Blimp-1. Les patrons d’expression (représentés par les triangles) de bcl-6 et blimp-1 sont inversement corrélés, permettant le contrôle de la différenciation des lymphocytes B en plasmocytes. Bcl-6 confère et maintient l’identité des cellules B des CG, en bloquant la réponse aux stress génotoxiques (répression de P53 et ATR par exemple), en réprimant des gènes impliqués dans l’arrêt du cycle cellulaire (P21) et dans l’apoptose, ainsi qu’en bloquant la différenciation plasmocytaire par la répression de prdm1. Inversement, Blimp-1 établit et maintient l’identité plasmocytaire en réprimant l’identité B (répression de pax5 et bcl-6), en inhibant la prolifération (c-Myc), et en activant l’expression de gènes importants pour la sécrétion d’anticorps (XBP-1 par exemple). |
De nombreuses études ont démontré le rôle prépondérant de Blimp-1 dans la différenciation terminale plasmocytaire [
8]. Ainsi, la surexpression expérimentale de ce facteur dans les lymphocytes B primaires suffit à induire leur différenciation en plasmocytes. Blimp-1 réprime l’identité cellulaire B (via la répression de Pax5 et bcl-6), permet la sortie du cycle cellulaire par la répression de c-myc, et induit la sécrétion d’anticorps par l’activation de XBP-1 (X-box binding protein1) (Figures 1, 2) [8]. |
Déséquilibre transcriptionnel dans les lymphomes B La stabilité des programmes transcriptionnels des cellules des CG et des plasmocytes repose sur la répression réciproque de Pax5/Bcl-6 et de Blimp-1, et cet équilibre est souvent remis en question dans les lymphomes B. Les cellules des CG, grâce à leur prolifération rapide, la commutation isotypique et les mutations hypersomatiques, sont particulièrement sujettes à des anomalies génétiques, comme des translocations chromosomiques ou mutations ponctuelles, qui conduisent à l’expression anormale de ces facteurs. Ainsi, en clinique, la plupart des lymphomes B ont pour origine des cellules des CG [7]. Le plus commun des lymphomes B (le lymphome diffus à grandes cellules B) est associé, dans 40 % des cas, à des translocations chromosomiques qui fusionnent le gène bcl-6 à différents promoteurs (plus de 20 identifiés) [6]. Dans 10 % des cas, une région régulatrice du gène bcl-6 est la cible d’hypermutations somatiques qui détruisent des sites de fixation à Bcl-6 nécessaires à son autorégulation négative [6]. Des translocations chromosomiques impliquant Pax5 et diverses altérations génétiques du gène prdm1 ont aussi été retrouvées dans différents lymphomes [
9–
11]. L’expression anormale, soutenue pour Bcl-6 et Pax5 et diminuée pour Blimp1, pourrait contribuer à la formation de ces lymphomes, en maintenant des cellules dans un état de prolifération et de stress génotoxique et dont la différenciation plasmocytaire est inhibée. L’ensemble de ces facteurs ont recours à différents mécanismes épigénétiques pour contrôler l’expression de leurs gènes cibles. Par exemple, Bcl-6 recrute différents complexes associés à une activité histone déacétylase (tel que le complexe NuRD) pour réprimer le gène prdm1 ainsi que des gènes impliqués dans l’arrêt du cycle cellulaire et l’apoptose [7]. Plus tôt dans la différenciation, Pax5 contribue aux réarrangements des gènes codant pour les immunoglobulines en aidant à la réorganisation tridimensionnelle de ce locus [3]. Finalement, une étude montre que Blimp-1 recrute l’histone méthyltransférase G9a pour réprimer l’expression de gènes cibles [
12]. Ainsi, la dérégulation de ces mécanismes épigénétiques pourrait amener, comme celle des facteurs de transcription, à la tumorigenèse. Des thérapies ciblant des mécanismes épigénétiques sont déjà utilisées ou en cours de développement pour traiter certains cancers, et représentent un domaine de recherche porteur d’espoir. |
À Remi Terrranova (FMI, Basel) et Olivia Du Roure (ESPCI, Paris) pour leurs critiques et suggestions lors de la préparation de cette nouvelle, ainsi qu’au Friedrich Miescher Instiute for Biomedical Research pour le soutien financier.
|
1. Fisher AG. Cellular identity and lineage choice. Nat Rev Immunol 2002; 2 : 977–82. 2. Sieweke MH, Graf T. A transcription factor party during blood cell differentiation. Curr Opin Genet Dev 1998; 8 : 545–51. 3. Cobaleda C, Schebesta A, Delogu A, Busslinger M. Pax5: the guardian of B cell identity and function. Nat Immunol 2007; 8 : 463–70. 4. Xie H, Ye M, Feng R, Graf T. Stepwise reprogramming of B cells into macrophages. Cell 2004; 117 : 663–76. 5. Phan RT, Dalla-Favera R. The BCL6 proto-oncogene suppresses p53 expression in germinal-centre B cells. Nature 2004; 432 : 635–9. 6. Parekh S, Polo JM, Shaknovich R, et al. BCL6 programs lymphoma cells for survival and differentiation through distinct biochemical mechanisms. Blood 2007; 110 : 2067–74. 7. Klein U, Dalla-Favera R. Germinal centres: role in B-cell physiology and malignancy. Nat Rev Immunol 2008; 8 : 22–33. 8. Kallies AS, Nutt L. Terminal differentiation of lymphocytes depends on Blimp-1. Curr Opin Immunol 2007; 19 : 156–62. 9. Poppe B, De Paepe P, Michaux L, et al. PAX5/IGH rearrangement is a recurrent finding in a subset of aggressive B-NHL with complex chromosomal rearrangements. Genes Chromosomes Cancer 2005; 44 : 218–23. 10. Tam W, Gomez M, Chadburn A, et al. Mutational analysis of PRDM1 indicates a tumor-suppressor role in diffuse large B-cell lymphomas. Blood 2006; 107 : 4090–100. 11. Pasqualucci L, Compagno M, Houldsworth J, et al. Inactivation of the PRDM1/BLIMP1 gene in diffuse large B cell lymphoma. J Exp Med 2006; 203 : 311–7. 12. Gyory I, Wu J, Fejer G, et al. PRDI-BF1 recruits the histone H3 methyltransferase G9a in transcriptional silencing. Nat Immunol 2004; 5 : 299–308. 13. Bouchard M. Les gènes Pax et la spécification cellulaire. Med Sci (Paris) 2003; 19 : 535–7. 14. Cavazzana-Calvo M, Six E, André-Schmutz I, Coulombel L. Hématopoïèse humaine : des cellules CD34 aux lymphocytes T. Med Sci (Paris) 2007; 23 : 151–9 15. Catalan N, Imai K, Revy P, Fischer A, Durandy A. Deux ans après, l’activation induced cytidine deaminase n’a pas livré tous ses secrets. Med Sci (Paris) 2003; 19 : 139–41. |