| |
| Med Sci (Paris). 2008 August; 24(8-9): 735–741. Published online 2008 August 15. doi: 10.1051/medsci/20082489735.L’intrusion des régulateurs de l’épigénome mâle dans les cellules somatiques cancéreuses Sophie Rousseaux,* Nicolas Reynoird, Jonathan Gaucher, and Saadi Khochbin* Équipe épigénétique et signalisation cellulaire.Université Joseph Fourier, Institut Albert Bonniot, Grenoble, F-38700 France Inserm U823, Institut Albert Bonniot, Grenoble, F-38706 France. Université Joseph Fourier, Institut Albert Bonniot, Grenoble, F-38700 France |
Anomalies de méthylation et expression illégitime de gènes dans les cellules cancéreuses La différenciation cellulaire peut être définie comme un processus de programmation du génome conduisant des cellules pluripotentes, telles que les cellules souches, vers un état d’organisation génomique propre à une expression spécifique de gènes. Cette programmation fait largement appel à des mécanismes assurant l’organisation structurale et fonctionnelle du génome incluant tous les mécanismes dits « épigénétiques ». En effet, des balisages moléculaires impliquant la méthylation de l’ADN et les modifications des histones contribuent de manière déterminante à la mise en place d’un génome différencié et sont à l’origine de l’expression des phénotypes définis par ces cellules. Il est maintenant très clairement établi que la transformation cellulaire bouleverse l’épigénome des cellules affectées et perturbe largement l’état de l’expression des gènes (pour revue, voir [
1]). En effet, l’une des perturbations épigénétiques les plus connues et étudiées dans ce contexte est celle de la méthylation de l’ADN [
47,
48] (→).
(→) Voir les articles de S. Laget et P.A. Defossez, page 725 de ce numéro, et l’article de M. Weber, page 731 de ce numéro
Nous connaissons depuis longtemps l’occurrence d’une méthylation aberrante de gènes dans les cellules cancéreuses, affectant, entre autres, les gènes codant pour des suppresseurs de tumeurs. Cette méthylation illégitime et la répression transcriptionnelle qui s’en suit, contribuent grandement à la transformation oncogénique des cellules [
2]. Les données les plus récentes laissent penser que ces anomalies épigénétiques se produisant dans les cellules cancéreuses ne sont pas restreintes à la seule méthylation de l’ADN et englobent également les histones et leurs modifications post-traductionnelles [
3]. Bien que l’on commence à caractériser de manière de plus en plus approfondie des perturbations épigénétiques observées dans les cellules cancéreuses, nous manquons de données sur l’origine de ces anomalies. Alors qu’un dysfonctionnement ou/et une dérégulation de l’expression des modulateurs épigénétiques peuvent aisément rendre compte de ces anomalies, il faudrait très probablement envisager aussi des scénarios à plusieurs étapes où des perturbations limitées et de faible impact initial sont amplifiées en réponse à l’activation ciblée de facteurs épigénétiques plus puissants, aboutissant à des altérations à grande échelle de l’épigénome et à la transformation cellulaire. Dans ce contexte, la dérégulation primaire ou secondaire de gènes normalement exprimés dans les cellules spermatogéniques pourrait avoir des conséquences dramatiques sur l’organisation génomique des cellules somatiques les exprimant de manière illégitime. L’une des réorganisations les plus spectaculaires du génome se produit lors de la spermatogenèse, associée à la méiose et plus particulièrement aux étapes post-méiotiques. En effet, en plus des recombinaisons et des réorganisations génomiques associées ayant lieu dans les cellules méiotiques, une reprogrammation massive du génome se déroule dans les cellules post-méiotiques, au cours de laquelle les histones sont enlevées et remplacées par des protéines basiques, spécifiques des spermatides et des spermatozoïdes (Figure 1) [
49] (→)
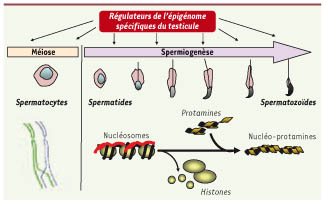
 | Figure 1.
Le génome subit deux étapes de re-organisation majeures et spécifiques au cours de la spermatogenèse. Premièrement, dans les spermatocytes diploïdes, le génome est brassé par les recombinaisons entre chromosomes homologues qui ont lieu lors de la prophase de méiose, puis les deux divisions nucléaires aboutissent à la formation des cellules germinales haploïdes. Deuxièmement, dans les cellules germinales mâles post-méiotiques (spermatides), la plupart des histones (associées à l’ADN dans la cellule somatique) sont enlevées et remplacées par des protéines spécifiques, les protamines, qui s’associent au génome (nucléo-protamines) et le compactent fortement dans le spermatozoïde. De nombreux facteurs régulant les différentes étapes de la méiose et la ré-organisation post-méiotique du génome haploïde sont exclusivement exprimés dans les cellules germinales, et réprimés dans les cellules somatiques normales. |
(→) Voir l’article de A. Henckel et R. Feil, page 747 de ce numéro
De ce fait, les cellules spermatogéniques expriment de manière spécifique un ensemble de régulateurs épigénétiques très puissants. Il est donc raisonnable de penser que leur expression illégitime pourrait grandement contribuer au bouleversement du paysage épigénétique des cellules somatiques et donc à la transformation cellulaire (Figure 2).
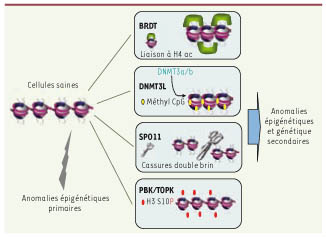
 | Figure 2.
Expression illégitime des régulateurs de l’épigénome mâle dans les cellules somatiques cancéreuses. Ces anomalies d’expression premièrement sont le reflet d’anomalies épigénétiques (primaires) et deuxièmement jouent un rôle dans la survenue d’anomalies génétiques et épigénétiques (secondaires). Les propriétés de quatre facteurs spécifiques du testicule pouvant être exprimés de manière illégitime dans les cancers somatiques illustrent cette deuxième hypothèse (voir texte). |
De plus, l’expression illégitime de ces gènes pourrait être considérée comme un excellent senseur d’anomalies épigénétiques se produisant dans les cellules cancéreuses. Une meilleure connaissance des mécanismes contrôlant la répression de ces gènes dans les cellules somatiques permettra de prédire la nature des anomalies épigénétiques affectant les cellules cancéreuses et à l’origine de la dérépression de leur transcription. Enfin, l’ensemble des gènes codant pour des protéines strictement spécifiques des cellules germinales mâles peut être considéré comme une source d’antigènes, susceptibles de déclencher une réaction immune lors de leur expression illégitime dans les cellules somatiques. La détection de leurs transcrits dans les prélèvements biologiques, ou bien d’anticorps dirigés contre les protéines codées par ces transcrits dans le sérum, donne accès à une large panoplie de marqueurs de tumeurs. Ces marqueurs, non seulement permettent de détecter et de suivre l’évolution des cancers, mais aussi d’envisager des approches préventives et thérapeutiques. Dans cette revue, nous développerons les implications associées à l’expression illégitime des gènes spécifiques du testicule dans les cellules pré-cancéreuses ou transformées. |
Reprogrammation du génome mâle lors de la méiose et durant les périodes post-méiotiques La spermatogenèse, via l’accomplissement d’un programme de différenciation unique impliquant une réorganisation spectaculaire cytoplasmique et nucléaire, aboutit à la génération des gamètes mâles matures ou spermatozoïdes. En particulier, les cellules engagées dans ce processus reprogramment leur génome en exprimant non seulement des facteurs spécifiques nécessaires au déroulement de la méiose, mais aussi ceux contrôlant le remplacement des histones par les protéines de transition (TP) et les protamines ainsi que la compaction extrême du génome qui en résulte (Figure 1). Alors que notre connaissance des facteurs méiotiques nous donne une idée relativement précise des mécanismes en jeu, nous ne connaissons presque rien de la reprogrammation post-méiotique du génome. L’analyse détaillée des évènements post-méiotiques a permis néanmoins de décrire certains aspects cruciaux de la réorganisation post-méiotique du génome. Une vague d’acétylation massive d’histones précède leur enlèvement et leur remplacement par les TP. Cette acétylation semble donner le signal de l’initiation d’une reprogrammation spécifique de différentes régions du génome, comme par exemple, celle des régions péricentromériques [
4]. Tous ces évènements impliquent des protéines structurales spécifiques exprimées exclusivement dans les cellules spermatogéniques, comme des variants d’histones ainsi que des puissants régulateurs de l’épigénome, comme le facteur à double bromodomaine Brdt, capable d’interagir, grâce à ses bromodomaines, avec les histones acétylées et de réorganiser la chromatine de manière spectaculaire. Il est donc facilement envisageable que l’expression illégitime de ces facteurs dans les cellules somatiques au cours de la transformation oncogénique puisse avoir un impact dramatique sur l’épigénome de ces cellules (Figure 2). La littérature est néanmoins très pauvre sur ce sujet et très peu d’études ont considéré le rôle de facteurs testiculaires dans la transformation de cellules somatiques. Nous décrivons les exemples les plus marquants en indiquant les possibles mécanismes cellulaires affectés par l’expression illégitime des facteurs testiculaires dans les cellules somatiques corroborant l’hypothèse de leur contribution déterminante dans la transformation maligne. |
Rôle oncogène des facteurs testiculaires dans les cellules somatiques L’activation illégitime dans les cellules somatiques de facteurs spécifiques du testicule, notamment de ceux qui sont impliqués dans la modulation de l’épigénome, peut avoir un effet amplificateur, aboutissant à des perturbations épigénétiques secondaires et déterminantes dans la transformation cellulaire. Le rôle de BORIS C’est le cas d’un paralogue du facteur CTCF ( CCCTC-binding factor), nommé BORIS, spécifiquement exprimé dans les cellules germinales mâles. CTCF est un facteur épigénétique (protéine à doigts de zinc) ayant un rôle déterminant dans la régulation spécifique d’allèle de gènes soumis à l’empreinte parentale [
46], une fonction de barrière transcriptionnelle ainsi qu’un rôle dans la régulation de l’expression des gènes en général [
5]. Dans les cellules germinales, BORIS est exprimé, alors que CTCF ne l’est pas [
6]. Bien que l’on ne connaisse pas précisément le rôle de BORIS au cours de la spermatogenèse, il est probable qu’il soit impliqué dans la régulation de l’expression d’autres gènes spécifiquement impliqués dans les voies de signalisation propres à la spermatogenèse, qui normalement sont aussi réprimés dans les cellules somatiques. L’expression illégitime de BORIS a été détectée dans un nombre significatif de cancers [
7–
11]. De manière intéressante, l’activation de BORIS dans les cellules somatiques induit l’expression d’autres facteurs C/T ( pour cancer/testis), plus particulièrement celle des gènes MAGE-A [ 7] et de CTAG1/NY-ESO1 [
12]. Dans ce dernier cas, BORIS recrute le facteur de transcription SP1 au promoteur de CTAG1 pour déréprimer sa transcription [
13]. Néanmoins, BORIS ne semble pas être le seul à agir pour induire l’activation secondaire des facteurs C/T [
10], et d’autres facteurs, comme par exemple le FGF ( fibroblast growth factor) [
14] et le récepteur à activité tyrosine kinase KIT [
15], contribuent grandement à favoriser l’expression illégitime de ces gènes. Parmi les différents gènes C/T, la dérépression primaire des membres de la famille MAGE peut mettre les cellules dans une configuration favorisant leur transformation maligne. En effet, il a été montré que la suppression de l’expression des MAGE de classe I (membres exprimés normalement uniquement dans les cellules germinales) induit l’apoptose des lignées cellulaires issues de mélanomes, positives pour l’expression illégitime des MAGE. Des études supplémentaires ont montré que cette résistance à l’apoptose était associée à une action neutralisante des MAGE sur P53 [
16]. En accord avec ces données, une autre étude a montré que MAGE-A2, un membre de la même famille, recrute l’histone-déacétylase 3 (HDAC3) et cible P53, et s’oppose ainsi à l’acétylation et à l’activation de P53 à la suite d’un traitement génotoxique [
17]. De plus, MAGE-A1 recrute l’histone désacétylase HDAC1 et ainsi transforme le régulateur transcriptionnel SKIP en un répresseur de transcription [
18]. … et celui de la protéine Brdt Un autre exemple concerne la protéine à double bromodomaine, Brdt. Cette protéine interagit fortement avec la chromatine acétylée et joue probablement un rôle déterminant dans le contrôle des évènements dépendants de l’acétylation lors de la maturation des spermatides. En effet, l’expression d’une protéine mutée dans son premier bromodomaine bloque la spermatogenèse aux stades qui suivent l’acétylation massive des histones [
19]. Brdt est également un facteur C/T. Bien que le gène BRDT soit exclusivement exprimé dans le testicule, la présence de l’ARN codant BRDT a été rapportée dans 12 cas (sur 47 étudiés) de cancers du poumon non à petites cellules, et dans un cas de carcinome à cellules squameuses [
20]. L’expression illégitime de BRDT dans les cancers broncho-pulmonaires, ainsi que sa capacité extraordinaire à remodeler et à condenser la chromatine acétylée, suggèrent qu’elle pourrait être impliquée dans la genèse et/ou l’évolution de ces cancers (Figure 2). Les mécanismes moléculaires impliqués restent à élucider. Expression illégitime d’autres protéines germinales dans les cellules cancéreuses Les cellules germinales possèdent un système spécifique de méthylation permettant l’établissement de novo de méthylations indispensables à la programmation de leur génome. La protéine DNMT3L, qui ne possède pas d’activité enzymatique propre, interagit avec et régule l’activité de deux méthyltransférases de l’ADN, DNMT3a et DNMT3b. DNMT3L est exprimé au cours de l’embryogenèse précoce et dans les cellules germinales et il est normalement réprimé dans les cellules somatiques. Une déméthylation de la région promotrice du gène DNMT3L est clairement détectée dans les cellules cancéreuses et associée à une expression aberrante du gène [
21]. Du fait du rôle régulateur important de DNMT3L dans la régulation de méthylations de novo [
22], il est facilement envisageable qu’une activation illégitime de DNMT3L puisse avoir un impact non négligeable sur l’épigénome en induisant une méthylation erronée du génome, et ainsi favoriser la transformation maligne des cellules affectées (Figure 2). Parmi les facteurs C/T, il y a également la protéine SPO11, qui induit des coupures double brin d’ADN lors des recombinaisons méiotiques (pour revue voir [
23]). Il est très probable que l’expression inappropriée de SPO11 dans les cellules somatiques favorise des translocations chromosomiques (Figure 2). Récemment les fonctions d’un orthologue chez la levure de deux C/T, CTAG1/NY-ESO1 et CTAG2/NY-ESO2, ont pu être définies. Cette protéine, PCC1, code pour un facteur de transcription capable de réguler l’expression d’un réseau de gènes impliqué dans le cycle cellulaire et la croissance polarisée chez la levure. Chez les mammifères, ces gènes sont uniquement exprimés dans les cellules germinales et leur dérépression aberrante pourrait affecter également des réseaux de gènes codant, comme chez la levure, pour des régulateurs cellulaires critiques [
24]. De manière intéressante, une kinase normalement exprimée dans les cellules germinales, PBK/TOPK, a été trouvée fréquemment dérégulée dans les cancers du sein. Les études fonctionnelles montrent que, dans les cellules somatiques, elle peut relayer la phosphorylation de la sérine 10 de l’histone H3 et donc avoir un rôle important dans les balisages erronés de cette marque épigénétique dans les cellules cancéreuses [
25]. Ces exemples sont donc très en faveur de l’hypothèse selon laquelle des dérégulations épigénétiques importantes peuvent surgir dans les cellules somatiques suivant une expression illégitime de gènes spécifiques de la gamétogenèse. Il apparaît également que des anomalies épigénétiques primaires dans les cellules pré-cancéreuses, dues à l’action « illégitime » des régulateurs du génome des gamètes, peuvent rapidement évoluer et aboutir à la transformation maligne. |
Activation aberrante des facteurs C/T, un indicateur des anomalies épigénétiques dans les cellules pré-cancéreuses et cancéreuses La perte, dans les cellules somatiques, de la répression des gènes spécifiquement exprimés dans les testicule peut être utilisée comme un bon détecteur des anomalies épigénétiques se produisant dans ces cellules. En effet, de nombreuses études ont montré que l’expression illégitime de plusieurs C/T est associée à une altération de la méthylation de leurs gènes. Par conséquent, lorsqu’une expression aberrante de ces gènes est détectée dans les cellules, elle peut être considérée comme le reflet direct de l’occurrence d’une méthylation anormale, et ainsi permettre de faire le diagnostic de cette anomalie épigénétique. De plus, de nouvelles données nous permettent d’entreprendre des analyses plus fines et de mieux comprendre les mécanismes épigénétiques responsables du maintien sous silence de ces gènes dans les cellules saines et d’identifier les perturbations épigénétiques se produisant dans les cancers. Par exemple, il a été montré que l’invalidation du gène G9a, ou du gène Glp/Eu-HMTase1, codant pour des méthyltransférases de la lysine 9 (K9) de l’histone H3, aboutit à une expression ectopique des gènes Mage [
26,
27]. Une analyse plus fine a permis de montrer qu’une mono-méthylation de K9, induite par l’expression d’un mutant de G9a dans les cellules G9a
−/− est suffisante pour réprimer l’expression du gène Mage-A [
28]. De plus, une acétylation des histones induite par le traitement des cellules par l’inhibiteur des HDAC, la trichostatine A (TSA), et la transcription qui en résulte, permettent une déméthylation du gène C/T GAGE et son expression stable dans les cellules somatiques [
29]. D’autres données suggèrent en revanche que la déméthylation ciblée des C/T se produit en premier lieu et peut être stabilisée par l’activité de ces promoteurs, les protégeant ainsi contre une re-méthylation et le rétablissement de la répression [
30,
31]. En effet, il a été montré que la déméthylation des régions promotrices de ces gènes et leur activation transcriptionnelle (due au ciblage des méthyltransférases de l’ADN, DNMT1/3b par des siARN), sont associées à une modification du code histone sur ces gènes en faveur d’un gain de marques actives, une augmentation de l’acétylation de K9 et de la méthylation de K4 de l’histone H3 [
32]. |
Les facteurs C/T, marqueurs de cancers et cibles de thérapie anti-cancéreuse À l’heure actuelle, on recense un peu plus d’une quarantaine de familles de gènes C/T, comprenant environ une centaine de gènes, dont l’expression anormale a été directement identifiée dans des cancers somatiques. La plupart de ces gènes sont répertoriés sur le site suivant : http://www.cancerimmunity.org/CTdatabase/. Les gènes C/T, dont l’expression est normalement restreinte aux cellules de la spermatogenèse méiotique ou post-méiotique, cellules protégées du système immunitaire par la barrière hémato-testiculaire, ont un statut immunitaire tout à fait particulier. En effet, dans des conditions physiologiques normales, en l’absence de cancer somatique ou d’effraction de la barrière hémato-testiculaire, les produits de ces gènes restent inconnus du système immunitaire autologue. En revanche, lorsqu’ils sont exprimés de manière aberrante dans les cancers somatiques, ils peuvent être reconnus par le système immunitaire qui les considère comme des antigènes étrangers, induisant une réaction cellulaire et/ou humorale contre les cellules tumorales. Ainsi, du fait de leur expression tissulaire restreinte et de leur caractère très immunogène, les gènes C/T et leurs produits constituent non seulement des marqueurs potentiels de choix pour le diagnostic des cancers, mais aussi des cibles idéales pour une immunothérapie anticancéreuse. En effet, les premiers antigènes C/T, de la famille MAGE-A, puis BAGE et GAGE, ont été découverts chez une patiente porteuse d’un mélanome dont l’évolution était anormalement favorable, du fait de la présence de lymphocytes T cytotoxiques reconnaissant les cellules tumorales autologues [
33], révélant déjà la nature fortement antigénique de ces facteurs. Dans les années qui suivirent, l’approche SEREX [
34]1 a mis à profit la réponse immunitaire humorale des patients cancéreux dirigée contre des protéines tumorales, pour identifier d’autres antigènes tumoraux, parmi lesquels une majorité de C/T (incluant les facteurs SSX, SCP1, NY-ESO-1). Enfin, un certain nombre d’antigènes C/T ont été identifiés sur la base de leur profil d’expression [
35,
36]. L’expression anormale d’un ou plusieurs C/T est détectée dans de nombreux cancers somatiques, tumeurs solides [
37–
40] ou hémopathies [
41,
42]. Cela peut être exploité pour le développement de marqueurs indiquant la présence et/ou de l’évolution d’un processus tumoral, basés sur la détection des transcrits correspondants [37], ou des protéines, ou même d’anticorps circulants dirigés contre ces C/T [
43]. À l’heure actuelle, on ne sait pas détecter des C/T dans tous les cancers somatiques (par exemple les C/T connus sont rarement exprimés dans les cancers colorectaux). Il est cependant possible que la découverte de nouveaux C/T permette de diagnostiquer de plus en plus de tumeurs. Plusieurs équipes ont exploité les propriétés immunogènes des C/T pour développer des stratégies vaccinales anticancéreuses. Les C/T MAGEA1 et NY-ESO-1 ont servi à faire des vaccins destinés à stimuler les réponses des cellules T, CD4+ et/ou CD8+, et un effet bénéfique a été démontré chez certains des patients traités [
44]. Afin d’assurer une efficacité optimale de ce type d’immunothérapie, des recherches en cours visent à optimiser la présentation de l’antigène aux cellules T, notamment en associant l’épitope à un adjuvant [
45]. De plus, certaines études récentes suggèrent d’associer à cette approche vaccinale un traitement épigénétique visant à induire ou augmenter l’expression de gènes C/T par les cellules tumorales, comme par exemple un agent déméthylant l’ADN ou inhibant les histones déacétylases [42]. |
Outre leur grand intérêt pour les applications médicales, l’étude des C/T représente une voie de recherche novatrice et puissante pour la compréhension des mécanismes fondamentaux régulant la programmation du génome au cours de la différenciation cellulaire, et des anomalies épigénétiques associées à la transformation cellulaire et au développement des cancers. En effet, l’expression des gènes C/T dans les cancers somatiques est le reflet d’une dérégulation épigénétique globale dans la cellule cancéreuse dont les mécanismes moléculaires restent à élucider. Il est également très important de souligner que, du fait du statut immunitaire tout à fait particulier des produits de ces gènes et de leur caractère hautement immunogène, les C/T sont une source de marqueurs de grande valeur pour la détection des cancers, ainsi que des cibles potentielles très intéressantes pour une thérapie vaccinale, comme le démontrent des études pilotes. Ainsi l’identification de C/T supplémentaires pourrait ouvrir la voie à une approche globale pour la détection d’une pathologie cancéreuse ainsi que pour une thérapie vaccinale. |
SR et SK remercient l’ANR, l’INCa, l’ARC (programme ARECA), la région Rhône-Alpes et la délégation à la recherche clinique du CHU de Grenoble (DRRC) pour leur soutien aux projets du laboratoire, et plus spécifiquement au projet C/T. NR et JG bénéficient d’une allocation doctorale de la région Rhône-Alpes.
|
Footnotes |
1. Deltour S, Chopin V, Leprince D. Modifications épigénétiques et cancer. Med Sci (Paris) 2005; 21 : 405–11. 2. Baylin SB. DNA methylation and gene silencing in cancer. Nat Clin Pract Oncol 2005; 2 (suppl 1) : S4–11. 3. Fraga MF, Ballestar E, Villar-Garea A, et al. Loss of acetylation at Lys16 and trimethylation at Lys20 of histone H4 is a common hallmark of human cancer. Nat Genet 2005; 37 : 391–400. 4. Govin J, Escoffier E, Rousseaux S, et al. Pericentric heterochromatin reprogramming by new histone variants during mouse spermiogenesis. J Cell Biol 2007; 176 : 283–94. 5. Wallace JA, Felsenfeld G. We gather together: insulators and genome organization. Curr Opin Genet Dev 2007; 17 : 400–7. 6. Loukinov DI, Pugacheva E, Vatolin S, et al. BORIS, a novel male germ-line-specific protein associated with epigenetic reprogramming events, shares the same 11-zinc-finger domain with CTCF, the insulator protein involved in reading imprinting marks in the soma. Proc Natl Acad Sci USA 2002; 99 : 6806–11. 7. Vatolin S, Abdullaev Z, Pack SD, et al. Conditional expression of the CTCF-paralogous transcriptional factor BORIS in normal cells results in demethylation and derepression of MAGE-A1 and reactivation of other cancer-testis genes. Cancer Res 2005; 65 : 7751–62. 8. D’Arcy V, Abdullaev ZK, Pore N, et al. The potential of BORIS detected in the leukocytes of breast cancer patients as an early marker of tumorigenesis. Clin Cancer Res 2006; 12 : 5978–86. 9. D’Arcy V, Pore N, Docquier F, et al. BORIS, a paralogue of the transcription factor, CTCF, is aberrantly expressed in breast tumours. Br J Cancer 2008; 98 : 571–9. 10. Kholmanskikh O, Loriot A, Brasseur F, et al. Expression of BORIS in melanoma: lack of association with MAGE-A1 activation. Int J Cancer 2008; 122 : 777–84. 11. Risinger JI, Chandramouli GV, Maxwell GL, et al. Global expression analysis of cancer/testis genes in uterine cancers reveals a high incidence of BORIS expression. Clin Cancer Res 2007; 13 : 1713–9. 12. Hong JA, Kang Y, Abdullaev Z, et al. Reciprocal binding of CTCF and BORIS to the NY-ESO-1 promoter coincides with derepression of this cancer-testis gene in lung cancer cells. Cancer Res 2005; 65 : 7763–74. 13. Kang Y, Hong JA, Chen GA, et al. Dynamic transcriptional regulatory complexes including BORIS, CTCF and Sp1 modulate NY-ESO-1 expression in lung cancer cells. Oncogene 2007; 26 : 4394–403. 14. Kondo T, Zhu X, Asa SL, Ezzat S. The cancer/testis antigen melanoma-associated antigen-A3/A6 is a novel target of fibroblast growth factor receptor 2-IIIb through histone H3 modifications in thyroid cancer. Clin Cancer Res 2007; 13 : 4713–20. 15. Yang B, Wu J, Maddodi N, et al. Epigenetic control of MAGE gene expression by the KIT tyrosine kinase. J Invest Dermatol 2007; 127 : 2123–8. 16. Yang B, O’Herrin SM, Wu J, et al. MAGE-A, mMage-b, and MAGE-C proteins form complexes with KAP1 and suppress p53-dependent apoptosis in MAGE-positive cell lines. Cancer Res 2007; 67 : 9954–62. 17. Monte M, Simonatto M, Peche LY, et al. MAGE-A tumor antigens target p53 transactivation function through histone deacetylase recruitment and confer resistance to chemotherapeutic agents. Proc Natl Acad Sci USA 2006; 103 : 11160–5. 18. Laduron S, Deplus R, Zhou S, et al. MAGE-A1 interacts with adaptor SKIP and the deacetylase HDAC1 to repress transcription. Nucleic Acids Res 2004; 32 : 4340–50. 19. Shang E, Nickerson HD, Wen D, et al. The first bromodomain of Brdt, a testis-specific member of the BET sub-family of double-bromodomain-containing proteins, is essential for male germ cell differentiation. Development 2007; 134 : 3507–15. 20. Scanlan MJ, Altorki NK, Gure AO, et al. Expression of cancer-testis antigens in lung cancer: definition of bromodomain testis-specific gene (BRDT) as a new CT gene, CT9. Cancer Lett 2000; 150 : 155–64. 21. Gokul G, Gautami B, Malathi S, et al. DNA Methylation profile at the DNMT3L promoter: A potential biomarker for cervical cancer. Epigenetics 2007; 2 : 80–5. 22. Ooi SK, Qiu C, Bernstein E, et al. DNMT3L connects unmethylated lysine 4 of histone H3 to de novo methylation of DNA. Nature 2007; 448 : 714–7. 23. Baudat F, de Massy B. SPO11 : une activité de coupure de l’ADN indispensable à la méiose. Med Sci (Paris) 2004; 20 : 213–8. 24. Kisseleva-Romanova E, Lopreiato R, Baudin-Baillieu A, et al. Yeast homolog of a cancer-testis antigen defines a new transcription complex. EMBO J 2006; 25 : 3576–85. 25. Park JH, Lin ML, Nishidate T, et al. PDZ-binding kinase/T-LAK cell-originated protein kinase, a putative cancer/testis antigen with an oncogenic activity in breast cancer. Cancer Res 2006; 66 : 9186–95. 26. Dodge JE, Kang YK, Beppu H, et al. Histone H3-K9 methyltransferase ESET is essential for early development. Mol Cell Biol 2004; 24 : 2478–86. 27. Tachibana M, Ueda J, Fukuda M, et al. Histone methyltransferases G9a and GLP form heteromeric complexes and are both crucial for methylation of euchromatin at H3-K9. Genes Dev 2005; 19 : 815–26. 28. Collins RE, Tachibana M, Tamaru H, et al. In vitro and in vivo analyses of a Phe/Tyr switch controlling product specificity of histone lysine methyltransferases. J Biol Chem 2005; 280 : 5563–70. 29. D’Alessio AC, Weaver IC, Szyf M. Acetylation-induced transcription is required for active DNA demethylation in methylation-silenced genes. Mol Cell Biol 2007; 27 : 7462–74. 30. De Smet C, Loriot A, Boon T. Promoter-dependent mechanism leading to selective hypomethylation within the 5’ region of gene MAGE-A1 in tumor cells. Mol Cell Biol 2004; 24 : 4781–90. 31. Loriot A, De Plaen E, Boon T, De Smet C. Transient down-regulation of DNMT1 methyltransferase leads to activation and stable hypomethylation of MAGE-A1 in melanoma cells. J Biol Chem 2006; 281 : 10118–26. 32. James SR, Link PA, Karpf AR. Epigenetic regulation of X-linked cancer/germline antigen genes by DNMT1 and DNMT3b. Oncogene 2006; 25 : 6975–85. 33. Van der Bruggen P, Traversari C, Chomez P, et al. A gene encoding an antigen recognized by cytolytic T lymphocytes on a human melanoma. Science 1991; 254 : 1643–7. 34. Sahin U, Tureci O, Schmitt H, et al. Human neoplasms elicit multiple specific immune responses in the autologous host. Proc Natl Acad Sci USA 1995; 92 : 11810–3. 35. Scanlan MJ, Gordon CM, Williamson B, et al. Identification of cancer/testis genes by database mining and mRNA expression analysis. Int J Cancer 2002; 98 : 485–92. 36. Bock-Axelsen J, Lotem J, Sachs L, Domany E. Genes overexpressed in different human solid cancers exhibit different tissue-specific expression profiles. Proc Natl Acad Sci USA 2007; 104 : 13122–7. 37. Scanlan MJ, Simpson AJ, Old LJ. The cancer/testis genes: review, standardization, and commentary. Cancer Immun 2004; 4 : 1–15. 38. Kalejs M, Erenpreisa J. Cancer/testis antigens and gametogenesis: a review and brain-storming session. Cancer Cell Int 2005; 5 : 4. 39. Simpson AJ, Caballero OL, Jungbluth A, et al. Cancer/testis antigens, gametogenesis and cancer. Nat Rev Cancer 2005; 5 : 615–25. 40. Costa FF, Le Blanc K, Brodin B. Concise review: cancer/testis antigens, stem cells, and cancer. Stem Cells 2007; 25 : 707–11. 41. Condomines M, Hose D, Raynaud P, et al. Cancer/testis genes in multiple myeloma: expression patterns and prognosis value determined by microarray analysis. J Immunol 2007; 178 : 3307–15. 42. Meklat F, Li Z, Wang Z, et al. Cancer-testis antigens in haematological malignancies. Br J Haematol 2007; 136 : 769–76. 43. Chen YT, Scanlan MJ, Sahin U, et al. A testicular antigen aberrantly expressed in human cancers detected by autologous antibody screening. Proc Natl Acad Sci USA 1997; 94 : 1914–8. 44. Odunsi K, Qian F, Matsuzaki J, et al. Vaccination with an NY-ESO-1 peptide of HLA class I/II specificities induces integrated humoral and T cell responses in ovarian cancer. Proc Natl Acad Sci USA 2007; 104 : 12837–42. 45. Susumu S, Nagata Y, Ito S, et al. Cross-presentation of NY-ESO-1 cytotoxic T lymphocyte epitope fused to human heat shock cognate protein 70 by dendritic cells. Cancer Sci 2008; 99 : 107–12. 46. Gabory A, Dandolo L. Épigénétique et développement : l’empreinte parentale. Med Sci (Paris) 2005; 21 : 390–5. 47. Laget S, Defossez PA. Le double jeu de l’épigénétique : cible et acteur du cancer. Med Sci (Paris) 2008; 24 : 725–30. 48. Weber M. Profils de méthylation de l’ADN dans les cellules normales et cancéreuses. Med Sci (Paris) 2008; 24 : 731–4. 49. Henckel A, Feil R. Asymétrie des génomes parentaux : implications en pathologie. Med Sci (Paris) 2008; 24 : 747–52. |