| |
| Med Sci (Paris). 2009 January; 25(1): 69–76. Published online 2009 January 15. doi: 10.1051/medsci/200925169.Anémie de Diamond-Blackfan Le côté obscur de la biogenèse des ribosomes Almass-Houd Aguissa-Touré,1* Lydie Da Costa,2 Thierry Leblanc,3 Gil Tchernia,4 Sébastien Fribourg,5 and Pierre-Emmanuel Gleizes1* 1Laboratoire de Biologie Moléculaire Eucaryote, Université de Toulouse et CNRS, 118, route de Narbonne, 31062 Toulouse, France 2Inserm U790, Université Paris-Sud, 94805 Villejuif, France et AP-HP, Hôpital Robert-Debré, 75935 Paris, France 3AP-HP, Hôpital Saint-Louis, 75475 Paris, France 4Centre d’information et de dépistage de la drépanocytose, Mairie de Paris, 75013 Paris, France 5Inserm U869, Institut Européen de Chimie et Biologie, Université de Bordeaux, Victor Segalen, 33607 Pessac, France |
Biogenèse des ribosomes et érythropoïèse : un lien inattendu La biogenèse des ribosomes est l’activité métabolique la plus consommatrice d’énergie dans une cellule qui prolifère. La synthèse des 4 ARN et des 79 protéines ribosomiques qui composent les deux sous-unités ribosomiques mobilise l’essentiel de l’activité transcriptionnelle, les ARN messagers (ARNm) codant les protéines ribosomiques comptant pour la moitié des ARNm cellulaires. Par ailleurs, la production des ribosomes est étroitement connectée à la régulation du cycle cellulaire [
1]. Malgré son rôle clé, peu de pathologies ont été liées à la mutation de gènes impliqués dans ce processus. Récemment, plusieurs maladies génétiques rares et un syndrome hématologique acquis ont été associés à la mutation de gènes codant une protéine ribosomique ou l’un des 300 facteurs requis pour la maturation des pré-ribosomes (particules précurseurs des sous-unités ribosomiques) [
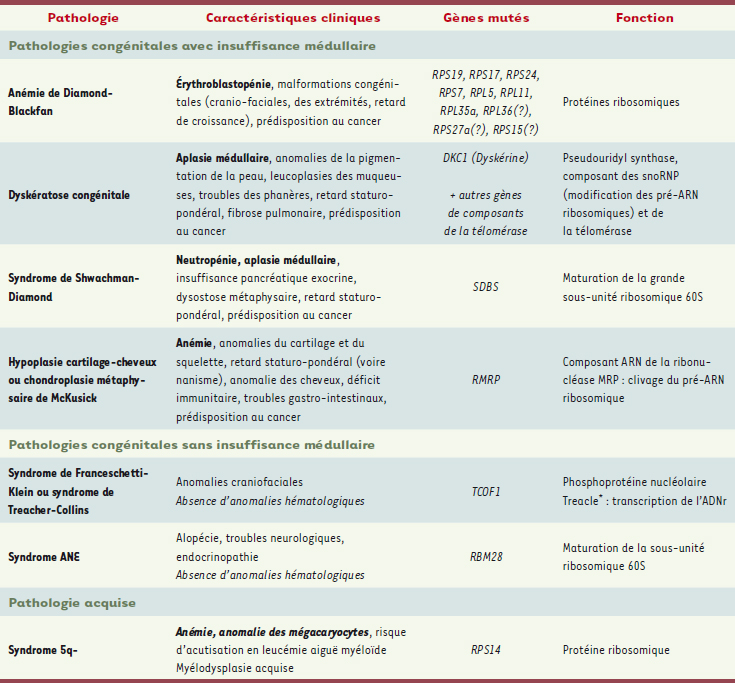
2]. De manière surprenante, ces pathologies sont presque toutes caractérisées par une insuffisance médullaire (Tableau I). Cette relation inattendue entre la biogenèse des ribosomes, un processus ubiquitaire, et le fonctionnement du tissu hématopoïétique est particulièrement bien caractérisée dans le cas de l’anémie de Diamond-Blackfan, la première maladie génétique associée à la mutation de gènes codant des protéines ribosomiques.  | Tableau I.
Pathologies liées à la mutation d’un gène codant une protéine ribosomique ou un facteur impliqué dans la biogenèse des ribosomes. Pour la dyskératose congénitale, quatre autres gènes impliqués dans le complexe de la télomérase peuvent être mutés [
44]. * Treacle : codée par le gène TCOF1 ( Teacher-Collins-Franceschetti syndrome 1). |
|
L’anémie de Diamond-Blackfan L’anémie de Diamond-Blackfan ou ADB (Diamond-Blackfan anemia) est une maladie rare (5 à 7 cas par million de naissances vivantes) qui résulte d’un déficit sévère de la prolifération et de la différenciation des précurseurs érythroïdes. La moelle osseuse présente un nombre très réduit d’érythroblastes (moins de 5 % des cellules nucléées), alors que les autres lignées hématopoïétiques se différencient normalement. L’anémie apparaît dans les semaines qui suivent la naissance et est associée chez 40 % des patients à des malformations plus ou moins sévères au niveau de la face, des extrémités des membres, de la sphère urogénitale et du cœur [
3–
5]. La plupart des patients présentent un retard de croissance qui peut être aggravé par l’anémie chronique et les traitements. On note également un risque augmenté d’hémopathies malignes et de certaines tumeurs solides [
6]. Plus de 60 % des patients répondent à un traitement par des corticoïdes à faible dose, suivant un mécanisme mal compris. Les patients réfractaires à ce traitement sont régulièrement transfusés et une greffe de moelle allogénique ou de sang de cordon HLA-identique est actuellement la seule voie curative. La possibilité de résolution spontanée, permettant d’arrêter le traitement, sans que l’on puisse en prédire le moment ou la durée, rend néanmoins difficile l’indication d’une greffe qui, en France, est proposée dans les formes graves dépendantes de transfusions, ou les formes compliquées. Un article de consensus sur le diagnostic et le traitement de l’ADB a été récemment publié par une communauté internationale de cliniciens [
7]. L’ADB est liée à un défaut intrinsèque des progéniteurs érythroïdes et non pas du stroma médullaire, comme l’indiquent à la fois des données in vitro et le succès curatif des greffes de moelle. L’analyse de la formation de colonies érythroïdes ex vivo à partir des progéniteurs hématopoïétiques de patients a mis en évidence un blocage de la différenciation des progéniteurs érythroïdes. D’après une étude récente, la différenciation s’arrêterait à la transition entre les progéniteurs BFU-e (burst-forming unit-erythroid) et CFU-e (colony-forming unit-erythroid)
1, c’est-à-dire au moment où s’exprime une dépendance à l’érythropoïétine [
8]. Néanmoins, cette question reste discutée : d’autres travaux ont fait le constat d’une réduction du nombre de progéniteurs précoces BFU-e, ce qui suggère un défaut plus en amont dans la hiérarchie de l’hématopoïèse [
9,
10]. En accord avec cette idée, il a été montré que la différenciation d’autres lignages hématopoïétiques pouvait être affectée chez certains patients [
11,
12]. |
L’ADB est associée à des mutations dans des protéines ribosomiques La découverte d’un caryotype anormal avec une translocation équilibrée chez une patiente suédoise a permis d’identifier le premier gène impliqué dans l’ADB, le gène codant la protéine ribosomique RPS19 [
13]. Des mutations, toutes hétérozygotes, ont alors été identifiées chez 25 % des patients inscrits dans l’ensemble des registres internationaux. L’introduction par transgenèse de copies surnuméraires du gène RPS19 dans des cellules médullaires de ces patients (et donc dans les progéniteurs érythroïdes) complémente en grande partie le défaut de différenciation érythroïde observé dans des cultures ex vivo [
14]. La cartographie pangénomique des SNP (single nucleotide polymorphisms) réalisée dans 215 familles a révélé d’autres locus en lien avec l’ADB : une région de 18 Mb sur le chromosome 8 (8p23.3-22) et des régions plus petites sur les chromosomes 6 et 10. Concernant le chromosome 10, le séquençage du gène codant la protéine ribosomique RPS24 a mis en évidence chez trois patients deux mutations non-sens et une délétion de la région codante, absentes lors de l’analyse de 200 génomes contrôles [
15]. L’analyse de délétions chromosomiques chevauchantes dans le chromosome 3 chez deux patients a permis de mettre en cause le gène RPL35A [
16]. Indépendamment, le séquençage systématique de plusieurs gènes codant des protéines ribosomiques a montré chez des patients la présence de mutations hétérozygotes dans les gènes RPS17 [
17,
18], RPS7, RPS15, RPS27a, RPL5, RPL11 et RPL36 [18]. La mutation de chacun de ces gènes touche moins de 1-2 % des patients (un seul cas répertorié pour RPS27a, RPS15 et RPL36), sauf dans le cas de RPL5 et RPL11 qui sont affectés chez 6,5 % et 5 % des patients respectivement. Il est notable que ces gènes codent aussi bien des protéines de la sous-unité 40S (gènes RPS) que de la sous unité 60S (gènes RPL). Sans être aussi complètes que les études concernant la protéine RPS19, ces observations récentes montrent l’accroissement du nombre de protéines ribosomiques liées à l’ADB et confortent l’hypothèse d’un lien entre cette maladie et la biogenèse ou la fonction des ribosomes. Les modèles animaux de la maladie sont encore en développement. L’interruption du gène RPS19 chez la souris n’a pas produit de phénotype à l’état hétérozygote, alors qu’elle est létale à l’état homozygote [
19]. L’extinction de la synthèse de RPS19 dans l’embryon de poisson zèbre grâce à des oligonucléotides de type morpholino conduit à de nombreux défauts de développement, en particulier de l’hématopoïèse [
20]. Cet organisme vertébré pourrait donc s’avérer intéressant pour l’étude de la fonction des protéines ribosomiques dans un modèle intégré. |
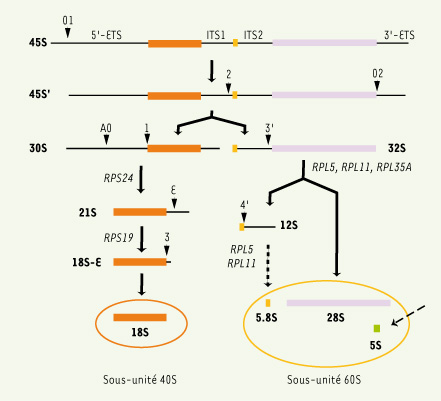
La biogenèse des ribosomes Les sous-unités ribosomiques sont le fruit de l’assemblage de 4 ARN, qui composent l’essentiel de la masse du ribosome, et d’environ 80 protéines. Ce processus se déroule principalement dans le nucléole, un domaine spécialisé du noyau [
21]. Trois des 4 ARN ribosomiques (ARNr) sont issus de la maturation d’un long transcrit précurseur appelé pré-ARN ribosomique (pré-ARNr). Ce transcrit primaire contient les ARN ribosomiques 18S, 5,8S et 28S et subit plusieurs étapes de clivage (Figure 1) qui éliminent les séquences transcrites additionnelles (ETS et ITS pour external et internal transcribed spacer). Par ailleurs, il est modifié enzymatiquement par les snoRNP (RNP pour ribonucleoprotein) qui catalysent l’isomérisation d’uridines en pseudo-uridines et la méthylation des riboses de plus de 200 nucléotides. 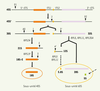 | Figure 1.
Maturation du pré-ARN ribosomique chez l’homme. La transcription des gènes ribosomiques par l’ARN polymérase I conduit à la synthèse d’un ARN précurseur dans lequel les portions d’ARN ribosomique 18S, 5.8S, et 28S sont incluses. Les têtes de flèche indiquent les sites des clivages qui amènent progressivement à l’élimination des espaceurs transcrits externes (ETS) et internes (ITS). L’élimination de la séquence 5’-ETS peut également intervenir avant le clivage de l’ITS1 au site 2, ce qui conduit à la formation de l’ARN intermédiaire 41S qui est ensuite résolu en ARN 21S et 32S. l’ARN 5S est synthétisé indépendament par l’ARN polymérase III. Les noms des protéines ribosomiques indiquent les étapes auxquelles ces protéines sont requises. |
Au cours de ce processus, les pré-ARNr sont associés avec des co-facteurs (protéines et snoRNP) dans des particules ribonucléoprotéiques appelées pré-ribosomes. La particule initiale ou particule 90S est scindée en deux à la suite du clivage de l’ITS1, donnant naissance aux particules pré-40S et pré-60S qui suivent des voies de maturation indépendantes. L’analyse protéomique de ces particules chez la levure Saccharomyces cerevisiae a révélé que plus de 150 protéines participent de manière transitoire à la composition des pré-ribosomes à différents stades, un nombre qui pourrait être plus élevé chez les mammifères. Les étapes ultimes de la synthèse des sous-unités 40S et 60S interviennent dans le cytoplasme, comme la maturation de l’extrémité 3’ de l’ARN 18S [
22]. Les modalités des clivages des pré-ARNr chez les mammifères ne sont pas aussi bien connues que chez la levure. On peut néanmoins noter que la maturation de l’ARN 18S comprend des étapes supplémentaires chez les mammifères. |
RPS19 et RPS24 sont essentielles à la formation des particules pré-40S L’étude de la fonction de RPS19 dans la biogenèse des ribosomes a révélé que cette protéine intervient à la transition entre la particule 90S et la particule pré-40S. Chez S. cerevisiae, l’interruption de la synthèse de RPS19 entraîne un clivage anormal dans l’ITS1 [
23]. Plusieurs facteurs pré-ribosomiques spécifiquement associés à la particule pré-40S ne sont plus incorporés. Les particules pré-40S anormales formées en l’absence de RPS19 ne quittent pas le noyau et sont dégradées rapidement, et l’ARN 18S n’est pas produit. Un défaut de maturation similaire est observé après l’interruption de la synthèse de RPS19 grâce à des siARN dans des cellules humaines en culture [
24–
26]. Dans ce cas, le pré-ARN ribosomique 21S n’est plus clivé en ARN 18S-E, le dernier précurseur de l’ARN 18S. Les particules pré-40S contenant de l’ARN 21S s’accumulent et semblent bloquées dans le nucléole, ce qui entraîne l’effondrement de la production des sous-unités 40S. Récemment, nous avons montré que la mutation de RPS24 bloque l’élimination du 5’-ETS [
27]. Les autres protéines ribosomiques impliquées dans l’ADB sont également nécessaires à la production de la petite ou de la grande sous-unité [16, 18]. Ainsi, en amont de leur rôle potentiel dans la traduction, ces protéines sont d’abord nécessaires à la biogenèse des ribosomes. |
Altération de la biogenèse des ribosomes chez les patients En accord avec les données obtenues in vitro, des cellules de patients atteints d’ADB présentent un défaut de maturation du pré-ARNr reproduisant l’effet des siRNA contre la protéine ribosomique considérée [16, 24–27]. Ainsi, on retrouve un défaut de conversion de l’ARN pré-ribosomique 21S en ARN 18S-E dans des cellules médullaires CD34+ (cellules souches et progéniteurs hématopoïétiques), ou dans des fibroblastes cutanés de patients porteurs de mutations dans RPS19 [24–26]. Un défaut similaire peut être observé dans des lignées lymphoblastoïdes établies à partir des lymphocytes de ces patients [24, 26, 27]. En revanche, des lignées lymphoblastoïdes dont le gène RPS24 est altéré montrent un taux élevé d’ARN 30S [27], alors que c’est la maturation des ARNr 5,8S et 28S qui est retardée en présence de la mutation de RPL35A, RPL5 et RPL11 [16, 18]. La présence d’un retard de maturation des ARN ribosomiques dans des fibroblastes cutanés est en accord avec la nature ubiquitaire de la biogenèse des ribosomes. De fait, les malformations constitutives de certains patients atteints d’ADB, comme le risque augmenté de néoplasies, indiquent que l’effet des mutations ne se limite pas à l’érythropoïèse. |
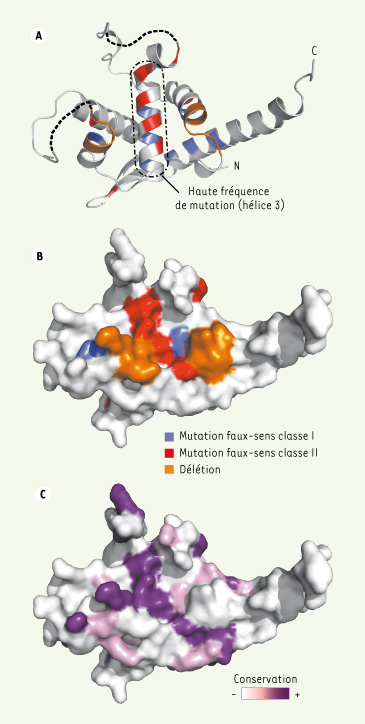
La structure de RPS19 révèle l’impact des mutations au cours de l’ADB Les mutations répertoriées dans le gène le plus fréquemment muté, RPS19, affectent la région promotrice, les séquences introniques et l’ensemble de la séquence codante : mutations faux-sens, non-sens, insertions, délétions [
28]. Si certaines de ces mutations aboutissent clairement à un allèle nul, l’impact des nombreuses mutations ponctuelles faux-sens est plus difficile à évaluer a priori. Pour répondre à cette question, nous avons récemment résolu la structure cristalline de la protéine S19e de l’archéon hyperthermophile Pyrococcus abyssi [
29]. Cette protéine présente plus de 50 % de similitude avec son homologue humain et s’est avérée être un bien meilleur candidat à la cristallisation. Cette structure a révélé deux classes de mutations faux-sens (Figure 2). Un premier groupe affecte des résidus enfouis dans le coeur hydrophobe de la protéine et impliqués dans le maintien de sa structure. De fait, la mutation de ces acides aminés affecte la stabilité de RPS19 et provoque la dégradation de la protéine par le protéasome dans des cellules en culture [
30–
32]. Une deuxième classe de mutations touche des acides aminés localisés à la surface de la protéine qui définissent des domaines basiques essentiels pour l’incorporation de RPS19 dans la particule pré-40S, comme l’ont confirmé des expériences chez la levure S. cerevisiae [29] et dans des cellules humaines en culture [30].  | Figure 2.
Position des mutations faux-sens liées à l’ADB dans la structure de la protéine RPS19. La structure de RPS19 (Pyroccocus abyssi) est composée d’un ensemble d’hélices α organisé autour de l’hélice 3 (A : vue en ruban ; B : vue en surface). L’hélice 3 correspond à la zone hyper-mutée de la protéine. Les mutations peuvent être réparties en deux classes : certaines affectent des résidus dans le coeur hydrophobe de la protéine et la déstabilisent (classe I, en bleu) ; d’autres touchent des acides aminés en surface de la protéine et perturbent probablement des interactions intermoléculaires (classe II, en rouge). Les résidus en orange indiquent deux délétions détectées chez des patients. Les acides aminés mutés sont proches dans la structure. Les mutations de classe II définissent un sillon très conservé dans la famille des protéines RPS19 (C). |
Ainsi, qu’elles diminuent la synthèse de RPS19, provoquent sa dégradation ou affectent ses surfaces d’interaction, les mutations pathogéniques de RPS19 entraînent toutes un déficit d’incorporation de RPS19 dans les pré-ribosomes, et par conséquent affectent la production des sous-unités 40S. |
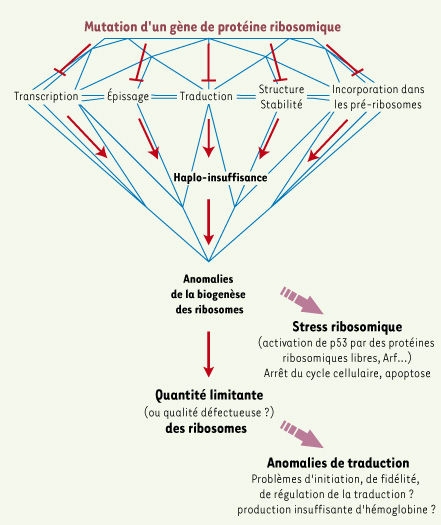
Les progéniteurs érythroïdes victimes d’un stress ribosomique ? Les données récentes nous conduisent à envisager un mécanisme physiopathologique commun aux mutations des différents gènes de protéines ribosomiques dans l’ADB (Figure 3). Si l’on extrapole les résultats obtenus pour RPS19, on peut envisager que la perte de fonction de chacune des protéines codées par ces gènes provoque une anomalie de la biogenèse des ribosomes, ce qui peut avoir deux conséquences directes : d’une part l’établissement d’un stress ribosomique, encore appelé stress nucléolaire, et d’autre part une limitation de la quantité de ribosomes fonctionnels.  | Figure 3.
Modèle de l’impact de la mutation des protéines ribosomiques dans l’anémie de Diamond-Blackfan. Les mutations d’un gène de protéine ribosomique affectent soit la synthèse de la protéine à différents niveaux, soit son incorporation dans les ribosomes. Ces situations aboutissent toutes à une insuffisance en protéine dont la conséquence première est d’affecter la biogenèse des ribosomes. Le stress qui en résulte active une voie de réponse susceptible de ralentir ou d’arrêter le cycle cellulaire. De plus, la limitation de la quantité de ribosomes produits peut conduire à des défauts qualitatifs ou quantitatifs de la traduction. |
La notion de stress ribosomique a été largement documentée, même si elle reste mal définie sur le plan mécanistique. Ainsi, l’arrêt de la transcription des gènes ribosomiques ou l’altération de la fonction de protéines nucléolaires provoque un arrêt du cycle cellulaire à la transition G1/S dans des cellules en culture [
33]. Ces résultats sont corroborés par plusieurs exemples in vivo indiquant que l’altération de la biogenèse des ribosomes se traduit par des défauts de contrôle du cycle cellulaire [
34]. L’arrêt du cycle cellulaire, sous l’effet d’un défaut de biogenèse des ribosomes, pourrait mettre en jeu des protéines ribosomiques libres : RPL5, RPL11 et RPL23 peuvent ainsi activer la protéine p53 en séquestrant la protéine HDM2, une E3 ubiquitine-ligase qui conjugue l’ubiquitine à p53 et active sa dégradation par le protéasome [
35–
37]. Il a aussi été proposé que la protéine ribosomique RPL11 inhibe directement l’activité du facteur de transcription c-Myc [
38]. Ainsi, un défaut de synthèse ou de maturation des ribosomes pourrait conduire à l’accumulation de protéines ribosomiques libres qui contribueraient à ces mécanismes anti-prolifératifs. À ce titre, la mutation de RPL5 et RPL11 chez certains patients atteints d’ADB [18] rend plausible l’implication directe de ces mécanismes dans la physiopathologie de l’ADB. Une autre piste conduit directement au suppresseur de tumeur Arf (p19Arf chez la souris, p14Arf chez l’homme) qui peut bloquer la progression en phase G1 indépendamment de p53 [
39]. Arf entre en jeu à différents niveaux de la biogenèse des ribosomes et pourrait intervenir dans le contrôle de qualité de ce processus [
40]. Plus largement, les études protéomiques de la composition du nucléole et des données fonctionnelles suggèrent fortement que ce domaine nucléaire joue un rôle central dans la régulation de la prolifération cellulaire et agit comme un senseur concentrant des protéines permettant de répondre à différents stress [
41,
42]. Si l’hypothèse d’un stress ribosomique est une piste importante pour comprendre comment la mutation de protéines ribosomiques peut interrompre un processus de différenciation, elle ne rend pas compte du lien physiopathologique privilégié entre l’altération de la biogenèse des ribosomes et l’érythropoïèse. Il est possible qu’un stress ribosomique devienne critique dans certaines situations physiologiques, au cours du développement et lors de l’érythropoïèse. En effet, le processus de différenciation érythroblastique provoque un cataclysme fonctionnel et architectural dans le noyau cellulaire : la condensation de la chromatine préparant l’expulsion du noyau aboutit à un arrêt progressif de la transcription et à un remaniement important de la structure nucléolaire, ce qui pourrait amplifier l’effet de stress. En aval de la biogenèse des ribosomes, la limitation de la quantité de ribosomes ou, plus indirectement, un défaut qualitatif des ribosomes consécutif à la désorganisation de leur synthèse, pourrait affecter l’efficacité ou la régulation de la traduction à cette étape de forte prolifération cellulaire et de production massive d’hémoglobine. Plusieurs facteurs pourraient alors concourir à l’arrêt du cycle cellulaire, voire au déclenchement de l’apoptose. |
L’étude de l’ADB a permis de faire émerger le concept nouveau de « maladie ribosomique » et mis en évidence une relation physiologique particulière entre biogenèse des ribosomes et érythropoïèse. Cette relation a été récemment renforcée par l’association d’un syndrome myélodysplasique acquis, le syndrome 5q-, à l’haploinsuffisance du gène de la protéine ribosomique RPS14 [
43]. Ces données appellent désormais une étude plus spécifique des mécanismes et de la régulation de la synthèse des ribosomes dans les progéniteurs érythroïdes et de son articulation avec le processus de différenciation érythroïde. Par ailleurs, la caractérisation des voies de stress activées en réponse à un défaut de biogenèse des ribosomes est nécessaire pour comprendre leur implication dans l’ADB ou dans d’autres maladies impliquant des facteurs de la biogenèse des ribosomes. Il reste également à préciser quel est l’impact sur la traduction de l’haploinsuffisance d’un gène de protéine ribosomique. L’exploration de ces différentes pistes de travail nécessitera de nouveaux modèles d’étude, en particulier animaux. Derrière ces mécanismes physiopathologiques se trouvent probablement des clés pour comprendre l’intégration de la biogenèse des ribosomes dans la régulation du destin cellulaire. |
Les travaux des auteurs sont financés par l’ANR (programme « Maladies rares »/projet RIBODBA), l’ARC et la FRM. Nous remercions nos collaborateurs pour leur aide dans la relecture critique de ce manuscrit.
|
Footnotes |
1. Dez C, Tollervey D. Ribosome synthesis meets the cell cycle. Curr Opin Microbiol 2004; 7 : 631–7. 2. Liu JM, Ellis SR. Ribosomes and marrow failure: coincidental association or molecular paradigm ? Blood 2006; 107 : 4583–8. 3. Chen S, Warszawski J, Bader-Meunier B, et al. Diamond-blackfan anemia and growth status: the French registry. J Pediatr 2005; 147 : 669–73. 4. Leblanc T, Gluckman E, Brauner R. Growth hormone deficiency caused by pituitary stalk interruption in Diamond-Blackfan anemia. J Pediatr 2003; 142 : 358. 5. Willig TN, Gazda H, Sieff CA. Diamond-Blackfan anemia. Curr Opin Hematol 2000; 7 : 85–94. 6. Lipton JM, Federman N, Khabbaze, et al. Osteogenic sarcoma associated with Diamond-Blackfan anemia: a report from the Diamond-Blackfan Anemia Registry. J Pediatr Hematol Oncol 2001; 23 : 39–44. 7. Vlachos A, Ball S, Dahl N, et al. Diagnosing and treating Diamond Blackfan anaemia: results of an international clinical consensus conference. Br J Haematol 2008; 142 : 859–76. 8. Ohene-Abuakwa Y, Orfali KA, Marius C, Ball SE. Two-phase culture in Diamond Blackfan anemia: localization of erythroid defect. Blood 2005; 105 : 838–46. 9. Bagnara GP, Zauli G, Vitale L, et al.
In vitro growth and regulation of bone marrow enriched CD34+ hematopoietic progenitors in Diamond-blackfan Anemia. Blood 1991; 78 : 10. 10. Nathan DG, Clarke BJ, Hillman DG, et al. Erythroid precursors in congenital hypoplastic (Diamond-Blackfan) anemia. J Clin Invest 1978; 61 : 489–98. 11. Casadevall N, Croisille L, Auffray I, et al. Age-related alterations in erythroid and granulopoietic progenitors in Diamond-Blackfan anaemia. Br J Haematol 1994; 87 : 369–75. 12. Giri N, Kang E, Tisdale JF, et al. Clinical and laboratory evidence for a trilineage haematopoietic defect in patients with refractory Diamond-Blackfan anaemia. Br J Haematol 2000; 108 : 167–75. 13. Draptchinskaia N, Gustavsson P, Andersson B, et al. The gene encoding ribosomal protein S19 is mutated in Diamond-Blackfan anaemia. Nat Genet 1999; 21 : 169–75. 14. Hamaguchi I, Ooka A, Brun A, et al. Gene transfer improves erythroid development in ribosomal protein S19-deficient Diamond-Blackfan anemia. Blood 2002; 100 : 2724–31. 15. Gazda HT, Grabowska A, Merida-Long LB, et al. Ribosomal protein S24 gene is mutated in Diamond-Blackfan anemia. Am J Hum Genet 2006; 79 : 1110–8. 16. Farrar JE, Nater M, Caywood E, et al. Abnormalities of the large ribosomal subunit protein, Rpl35A, in diamond-blackfan anemia. Blood 2008; 112 : 1582–92. 17. Cmejla R, Cmejlova J, Handrkova H, et al. Ribosomal protein S17 gene (RPS17) is mutated in Diamond-Blackfan anemia. Hum Mutat 2007; 28 : 1178–82. 18. Gazda HT, Sheen MR, Vlachos A, et al. Ribosomal protein L5 and L11 mutations are associated with cleft palate and abnormal thumbs in Diamond-Blackfan anemia patients. Am J Hum Genet 2008; 83 : 769–80. 19. Matsson H, Davey EJ, Draptchinskaia N, et al. Targeted disruption of the ribosomal protein S19 gene is lethal prior to implantation. Mol Cell Biol 2004; 24 : 4032–7. 20. Danilova N, Sakamoto K, Lin S. Ribosomal protein S19 deficiency in zebrafish leads to developmental abnormalities and defective erythropoiesis through activation of p53 protein family. Blood 2008; 112 : 5228–37. 21. Hernandez-Verdun D, Louvet E. Le nucléole : structure, fonctions et maladies associées Med Sci (Paris) 2004; 20 : 37–44. 22. Rouquette J, Choesmel V, Gleizes PE. Nuclear export and cytoplasmic processing of precursors to the 40S ribosomal subunits in mammalian cells. EMBO J 2005; 24 : 2862–72. 23. Léger-Silvestre I, Caffrey JM, Dawaliby R, et al. Specific role for yeast homologs of the Diamond-Blackfan anemia-associated Rps19 protein in ribosome synthesis. J Biol Chem 2005; 280 : 38177–85. 24. Choesmel V, Bacqueville D, Rouquette J, et al. Impaired ribosome biogenesis in Diamond-Blackfan anemia. Blood 2007; 109 : 1275–83. 25. Flygare J, Aspesi A, Bailey JC, et al. Human RPS19, the gene mutated in Diamond-Blackfan anemia, encodes a ribosomal protein required for the maturation of 40S ribosomal subunits. Blood 2007; 109 : 980–6. 26. Idol RA, Robledo S, Du HY, et al. Cells depleted for RPS19, a protein associated with Diamond-Blackfan Anemia, show defects in 18S ribosomal RNA synthesis and small ribosomal subunit production. Blood Cells Mol Dis 2007; 39 : 35–43. 27. Choesmel V, Fribourg S, Aguissa-Touré AH, et al. Mutation of ribosomal protein RPS24 in Diamond-Blackfan anemia results in a ribosome biogenesis disorder. Hum Mol Genet 2008; 17 : 1253–63. 28. Campagnoli MF, Ramenghi U, Armiraglio M, et al. RPS19 mutations in patients with Diamond-Blackfan anemia. Hum Mutat 2008; 29 : 911–20. 29. Gregory LA, Aguissa-Touré AH, Pinaud N, et al. Molecular basis of Diamond-Blackfan anemia: structure and function analysis of RPS19. Nucleic Acids Res 2007; 35 : 5913–21. 30. Angelini M, Cannata S, Mercaldo V, et al. Missense mutations associated with Diamond-Blackfan anemia affect the assembly of ribosomal protein S19 into the ribosome. Hum Mol Genet 2007; 16 : 1720–7. 31. Da Costa L, Tchernia G, Gascard P, et al. Nucleolar localization of RPS19 protein in normal cells and mislocalization due to mutations in the nucleolar localization signals in 2 Diamond-Blackfan anemia patients: potential insights into pathophysiology. Blood 2003; 101 : 5039–45. 32. Crétien A, Hurtaud C, Moniz H, et al. Study of the effects of proteasome inhibitors on ribosomal protein S19 (RPS19) mutants, identified in Diamond-Blackfan anemia patients. Haematologica 2008; 93 : 1627–34. 33. Pestov DG, Strezoska Z, Lau LF. Evidence of p53-dependent cross-talk between ribosome biogenesis and the cell cycle: effects of nucleolar protein Bop1 on G(1)/S transition. Mol Cell Biol 2001; 21 : 4246–55. 34. Sulic S, Panic L, Barkic M, et al. Inactivation of S6 ribosomal protein gene in T lymphocytes activates a p53-dependent checkpoint response. Genes Dev 2005; 19 : 3070–82. 35. Dai MS, Lu H. Inhibition of MDM2-mediated p53 ubiquitination and degradation by ribosomal protein L5. J Biol Chem 2004; 279 : 44475–82. 36. Dai MS, Zeng SX, Jin Y, et al. Ribosomal protein L23 activates p53 by inhibiting MDM2 function in response to ribosomal perturbation but not to translation inhibition. Mol Cell Biol 2004; 24 : 7654–68. 37. Lohrum MA, Ludwig RL, Kubbutat MH, et al. Regulation of HDM2 activity by the ribosomal protein L11. Cancer Cell 2003; 3 : 577–87. 38. Dai MS, Arnold H, Sun XX, Sears R, Lu H. Inhibition of c-Myc activity by ribosomal protein L11. EMBO J 2007; 26 : 3332–45. 39. Carnero A, Hudson JD, Price CM, Beach DH. p16INK4A and p19ARF act in overlapping pathways in cellular immortalization. Nat Cell Biol 2000; 2 : 148–55. 40. Ayrault O, Andrique L, Larsen CJ, Séité P. La régulation négative de la biogenèse des ribosomes : une nouvelle voie de contrôle du cycle celullaire par le suppresseur de tumeur Arf ? Med Sci (Paris) 2006; 22 : 519–24. 41. Olson MO. Sensing cellular stress: another new function for the nucleolus ? Sci STKE 2004; 2004 : pe10. 42. Rubbi CP, Milner J. Disruption of the nucleolus mediates stabilization of p53 in response to DNA damage and other stresses. EMBO J 2003; 22 : 6068–77. 43. Ebert BL, Pretz J, Bosco J, et al. Identification of RPS14 as a 5q- syndrome gene by RNA interference screen. Nature 2008; 451 : 335–9. 44. Hoareau-Aveilla C, Henry Y, Leblanc T. La dyskératose congénitale : une maladie méconnue due à un maintien défectueux des télomères. Med Sci (Paris) 2008; 24 : 390–8. |