| |
| Med Sci (Paris). 2009 March; 25(3): 307–310. Published online 2009 March 15. doi: 10.1051/medsci/2009253307.L’information cachée dans le repliement des protéines Il n’y a pas que la protéine prion de mammifère qui se « prionise » Karine Toupet1,2,3* and Sylvain Lehmann4,5,6* 1Université Montpellier 2, 34095 Montpellier, France 2Inserm, U710, 34095 Montpellier, France 3EPHE, Paris, 75007 Paris, France 4Institut de Génétique Humaine, CNRS, UPR 1142, 34396 Montpellier, France 5CHU de Montpellier, 34295 Montpellier 6Université Montpellier 1, 141, rue de la Cardonille, Bâtiment 24, CC105, Place Eugène Bataillon, 34095 Montpellier France |
Les maladies à prions sont l’exemple type des pathologies « conformationnelles » également appelées protéinopathies, dans lesquelles une protéine ou un fragment de protéine acquiert une conformation anormale et forme des agrégats sous la forme d’amyloïde. On retrouve dans cette famille une trentaine d’autres maladies : des maladies neurodégénératives, maladie de Parkinson, d’Huntington et d’Alzheimer, mais aussi d’autres affections systémiques comme l’amylose et le diabète de type 2. Dans ces protéinopathies, on observe des phénomènes de repliement anormaux associés à une accumulation de protéines qui résistent à la dégradation et s’organisent sous formes d’agrégats insolubles. C’est le cas en particulier du peptide Aβ pour la maladie d’Alzheimer, de l’α-synucléine pour la maladie de Parkinson ou bien encore de la huntingtine pour la maladie d’Huntington. Le repliement des protéines et son contrôle sont essentiels dans l’organisme pour obtenir des protéines stables et fonctionnelles. De nombreux mécanismes cellulaires entrent en jeu pour cela (rôle des chaperons moléculaires, du protéasome, de l’ubiquitinylation…). Pourtant, dans les protéinopathies, il existe une mise en défaut de ces mécanismes. On constate en fait que les phénomènes de conformation/repliement anormaux et d’agrégation sont liés, et qu’ils procèdent de façon similaire dans toutes les protéinopathies. La compréhension des mécanismes moléculaires sous-jacents dans l’une de ces affections va pouvoir ainsi enrichir la compréhension des autres. Nous illustrons dans cet article trois de ces mécanismes en rapportant les données récentes présentées par trois orateurs aux « Journées inter-IFR » de Montpellier (RIMO, 2008). |
Des prions de mammifères aux prions de levure ou comment un modèle prion chez la levure permet de trouver des agents thérapeutiques potentiels chez l’homme La nature exacte de l’agent infectieux responsable des maladies à prions est toujours l’objet de controverses mais l’hypothèse des « prions » pour « Proteinaceous infectious particle » proposé dans les années 1980 par Stanley Prusiner reste la plus communément admise [
1]. Selon cette hypothèse, l’agent infectieux est une protéine appelée PrPSc qui correspond à une conformation anormale d’une protéine physiologique appelée PrPC. La transmission des prions se ferait parce que la PrPSc induirait un changement de conformation de la PrPC normale en PrPSc pathologique caractérisé par l’acquisition d’une structure riche en feuillets β. Ce phénomène « prion », ou transmission d’une information conformationelle, n’est pas limité à la protéine prion et aux mammifères. En effet, certains phénotypes de transmission non mendélienne chez la levure Saccharomyces cerevisiae [
2] ou chez certains champignons comme Podospora anserina [
3] mettent en jeu des « mécanismes prions ». Dans ces modèles prions dits « analogues », des protéines (Ure2p, Sup35p…) sont capables d’exister sous forme « prionisée ». Une fois converties elles peuvent alors transmettre leur conformation différente associée à des fonctions biologiques modifiées. Les similitudes entre prions de mammifères et prions de levure ne s’arrêtent pas là : tout comme la PrPC, la protéine Sup35p, par exemple, est initialement soluble et forme des agrégats amyloïdes fibrillaires une fois convertie en « prion », comme le fait la PrPSc. Ces similitudes entre les prions de mammifères et les prions de levure, ajoutées à la grande facilité d’emploi, au faible coût et à l’innocuité de la levure, font de cet outil un modèle de choix pour l’étude du mécanisme « prion » et, au-delà, des mécanismes d’agrégation à l’œuvre dans les protéinopathies. |
Rôle des chaperons moléculaires dans la propagation des prions Le travail sur les prions de levure a permis de mettre en évidence l’importance de protéines chaperons, et en particulier de la protéine Hsp104, dont la sur- ou sous-expression stoppe la propagation. Cette protéine fait partie d’une famille de protéines ubiquitaires dont le rôle principal est de lutter contre le dépliement partiel, le repliement alternatif et l’agrégation des protéines. L’un de ses modes d’action proposés afin d’expliquer l’importance des chaperons moléculaires dans la formation des prions consisterait à induire la fragmentation des larges agrégats protéiques en oligomères, offrant alors de multiples sites capables d’induire de nouvelles polymérisations associées aux changements conformationnels liés à la conversion [
4]. Cette importance de Hsp104 dans la propagation des prions de levure et la production de nouveaux « propagons » in vivo, a été démontrée par Mick Tuite et son équipe (Université de Kent, Canterbury, Royaume-Uni) en utilisant du chlorure de guanidium qui inhibe l’activité ATPasique de Hsp104 [
5] et différents mutants de Hsp104 [
6]. Outre Hsp104, d’autres chaperons moléculaires semblent aussi intervenir dans la propagation des prions de levure (Hsp70, Hsp40…) [
7]. Même si Hsp104 joue un rôle central chez la levure, cette molécule n’a pas d’analogue chez l’homme. Ceci rend difficile les approches visant à manipuler l’expression des chaperons à des fins thérapeutiques dans les prions de mammifères et, par extension, dans les protéinopathies. |
Les prions de levure : un modèle de choix pour la recherche de composés thérapeutiques Les prions de levure sont un modèle de choix non seulement pour l’étude des mécanismes prions mais aussi pour réaliser des criblages à haut débit dans le but de rechercher des molécules à visée thérapeutiques. Ainsi, Marc Blondel (Inserm U613, Brest) et son équipe ont développé une méthode simple et rapide de criblage chez Saccharomyces cerevisiae basée sur le fait qu’en présence de formes prions, on peut obtenir des colonies blanches et autotrophes pour l’adénine, alors qu’en l’absence de conversion, les colonies sont rouges et auxotrophes pour l’adénine [
8] (Figure 1). Un criblage primaire d’une chimiothèque a été effectué sur le prion [PSI+
] ; les molécules actives ont ensuite été testées contre le prion [URE3]. Étant donné la grande divergence tant fonctionnelle que de séquence primaire des protéines impliquées dans la formation de ces deux prions, les composés actifs contre les deux sont potentiellement intéressants pour bloquer les mécanismes prions de façon générale et par extension dans les protéinopathies. Parmi les molécules identifiées par Marc Blondel et ses collaborateurs, la 6-aminophénanthridine (6AP) et le guanabenz (GA) se sont aussi révélées actives contre le prion de mammifère, non seulement ex vivo dans des cultures de cellules chroniquement infectées par le prion, mais aussi in vivo dans un modèle animal (murin) pour les maladies à prions [
9,
10]. Des essais sont en cours dans des modèles plus complexes (animaux transgéniques…) pour évaluer l’intérêt des molécules ainsi sélectionnées. Ces résultats valident ainsi le modèle de la levure et ses prions pour isoler des molécules actives contre les prions de mammifère. Ils suggèrent, en outre, que des mécanismes de prionisation sont conservés de la levure aux mammifères, motivant ainsi la recherche des cibles intracellulaires de la 6AP et du GA.  | Figure 1.
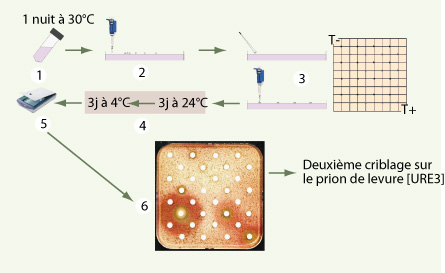
Principe du test de criblage pour la recherche de molécules anti-prions.
Un aliquot provenant d’une culture en milieu liquide (1) d’une souche de prion [PSI+
] est réparti sur une boîte de Pétri contenant un milieu complémenté avec 200 µM de guanidine hydrochloride. De petits filtres sont alors disposés sur la surface d’agar (2). Les molécules à tester sont ensuite appliquées sur chaque filtre (3), le filtre situé en haut à gauche étant réservé au contrôle négatif (DMSO) et celui en bas à droite au contrôle positif (5µl d’une solution de GuHCL à 300 mM). Quand une molécule est active contre le prion de levure [PSI+
], un halo rouge de colonies apparaît autour de la région du filtre où la molécule a été déposée (6). Les molécules positives sont ensuite testées sur le prion de levure [URE3]. (d’après Pr M. Blondel, Inserm U613, Brest, France). |
|
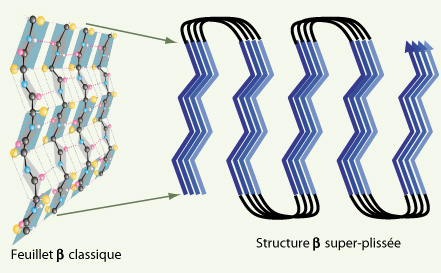
Comment l’étude biophysique et in silico des phénomènes d’agrégation permettent de rassembler les protéinopathies sous une même entité moléculaire La compréhension des repliements pathogéniques et l’étude structurale des agrégats et des fibrilles amyloïdes présents dans les protéinopathies et les prions sont des étapes clés pour le développement de nouvelles stratégies thérapeutiques. Depuis dix ans, de nombreux progrès ont été faits via l’utilisation de nouvelles techniques telles que la spectroscopie RMN (résonance magnétique nucéaire) à l’état solide, la microscopie électronique en transmission à balayage, ou encore la spectroscopie par résonance paramagnétique électronique. L’intégration des informations issues de ces différentes techniques grâce à des approches de modélisation moléculaire a permis à A. Kajava et ses collaborateurs (CRBM, Montpellier) de suggérer que les structures amyloïdes retrouvées dans les protéinopathies, qu’elles soient constituées de tau, α-synucléine, Ure2p, Sup35p, huntingtine ou encore de l’amyline humaine, ont une structure commune appelée « structure β super-plissée » (association en parallèle de plusieurs feuillets β) (Figure 2) [
11,
12]. Les modèles proposés par d’autres groupes pour les fibres amyloïdes de Aβ [
13], HET-s [
14] et Prion [
15] ont également des feuillets β parallèles. Toutes ces données ouvrent la voie vers le développement de nouvelles thérapeutiques communes pour ces maladies.  | Figure 2.
Modèle structural proposé pour différents amyloïdes.
De nouvelles techniques expérimentales et approches bioinformatiques suggèrent que les différentes amyloïdes retrouvées dans les protéinopathies sont constituées de l’association en parallèle de plusieurs feuillets β (structure β super-plissée) (d’après Dr A. Kajava, CRBM, Montpellier, France). |
|
Les études sur les prions de mammifères et celles sur les « prions » présents chez les levures et les champignons filamenteux ont permis de confirmer l’existence de la transmission d’une information biologique basée sur la conformation des protéines qui représente une nouvelle facette des mécanismes épigénétiques. Ces travaux ont également permis des avancées sur les mécanismes moléculaires des changements conformationels et d’agrégations protéiques conduisant à la formation d’amyloïdes et à des phénotypes pathologiques observés dans les « protéinopathies ». Cela explique les connexions importantes entre les recherches diagnostiques et thérapeutiques communes à toutes ces pathologies. |
Footnotes |
1. Prusiner SB. Novel proteinaceous infectious particles cause scrapie. Science 1982; 216 : 136–44. 2. Wickner RB. [URE3] as an altered URE2 protein: evidence for a prion analog in Saccharomyces cerevisiae. Science 1994; 264 : 566–9. 3. Coustou V, Deleu C, Saupe S, Begueret J. The protein product of the het-s heterokaryon incompatibility gene of the fungus Podospora anserina behaves as a prion analog. Proc Natl Acad Sci USA 1997; 94 : 9773–8. 4. Tuite MF, Cox BS. Propagation of yeast prions. Nat Rev Mol Cell Biol 2003; 4 : 878–90. 5. Ferreira PC, Ness F, Edwards SR, et al. The elimination of the yeast [PSI+] prion by guanidine hydrochloride is the result of Hsp104 inactivation. Mol Microbiol 2001; 40 : 1357–69. 6. Zenthon JF, Ness F, Cox B, Tuite MF. The [PSI+] prion of Saccharomyces cerevisiae can be propagated by an Hsp104 orthologue from Candida albicans. Eukaryot Cell 2006; 5 : 217–25. 7. Kryndushkin DS, Smirnov VN, Ter-Avanesyan MD, Kushnirov VV. Increased expression of Hsp40 chaperones, transcriptional factors, and ribosomal protein Rpp0 can cure yeast prions. J Biol Chem 2002; 277 : 23702–8. 8. Tribouillard D, Bach S, Gug F, et al. Using budding yeast to screen for anti-prion drugs. Biotechnol J 2006; 1 : 58–67. 9. Bach S, Talarek N, Andrieu T, et al. Isolation of drugs active against mammalian prions using a yeast-based screening assay. Nat Biotechnol 2003; 21 : 1075–81. 10. Tribouillard-Tanvier D, Beringue V.Antihypertensive drug Guanabenz is active in vivo against both yeast and mammalian prion. PLoS ONE 2008; 3 : e1981 11. Kajava AV, Baxa U, Wickner RB, Steven AC. A model for Ure2p prion filaments and other amyloids: the parallel superpleated beta-structure. Proc Natl Acad Sci USA 2004; 101 : 7885–90. 12. Kajava AV, Aebi U, Steven AC. The parallel superpleated beta-structure as a model for amyloid fibrils of human amylin. J Mol Biol 2005; 348 : 247–252. 13. Petkova AT, Ishii Y, Balbach JJ, et al. A structural model for Alzheimer’s beta -amyloid fibrils based on experimental constraints from solid state NMR. Proc Natl Acad Sci USA 2002; 99 : 16742–7. 14. Ritter C, Maddelein ML, Siemer AB, et al. Correlation of structural elements and infectivity of the HET-s prion. Nature. 2005; 435 : 844–8. 15. Govaerts C, Wille H, Prusiner SB, Cohen FE. Evidence for assembly of prions with left-handed beta-helices into trimers. Proc Natl Acad Sci USA 2004; 101 : 8342–7. |