Approches globales des systèmes biologiques
Pour relever les défis de l’innovation thérapeutique et saisir de telles opportunités, l’industrie pharmaceutique doit évoluer vers un nouveau système de pensée dans la découverte et vers un développement du médicament basé sur une compréhension globale des systèmes biologiques. En effet, une des clés du succès de ces médicaments de demain consiste en une meilleure connaissance de la maladie, incluant une connaissance approfondie des mécanismes moléculaires impliqués, ainsi que l’identification de fils conducteurs entre la préclinique et la clinique, entre les modèles animaux et les futurs patients, et ceci dès les stades de recherche
(Voir Encadré ci-contre). En effet, si de multiples éléments peuvent être à l’origine des échecs des candidats médicaments en développement clinique, il apparaît de plus en plus fondamental de pouvoir définir les caractéristiques biologiques (biochimiques et physiologiques) de « l’état normal » et d’approfondir les mécanismes physiopathologiques et étiologiques impliqués dans la genèse et la progression des maladies chez l’homme.
En effet, la première étape dans cette démarche d’une meilleure connaissance des maladies est l’identification des cibles moléculaires impliquées dans le processus physiopathologique. Elle nécessite un lien fort et précoce entre la découverte du médicament et la recherche clinique dans un processus de recherche translationnelle (faisant intervenir les différentes compétences) pour lequel l’industrie crée peu à peu un nouvel environnement et instaure un nouveau mode de fonctionnement. Elle implique également la constitution de réseaux avec des partenaires académiques et hospitaliers, les autorités réglementaires ainsi que des consortiums de chercheurs à l’échelle européenne. Cette recherche innovatrice devra de plus être positionnée sur l’ensemble du cycle de vie d’un médicament, de la découverte jusqu’au lit du malade (« bench to bedside »).
La deuxième étape, sur la base d’une meilleure connaissance des mécanismes impliqués, est d’identifier des fils conducteurs, (bio)marqueurs permettant un suivi plus précis de l’évolution de la maladie et de l’activité pharmacologique et thérapeutique des futurs candidats médicaments. De plus, cette connaissance sera mieux traduite dans les nouveaux modèles animaux afin d’améliorer leur pouvoir prédictif en termes d’efficacité ou de toxicité clinique.
Ce continuum entre la recherche préclinique et clinique situe clairement les patients et la physiopathologie moléculaire au centre du processus de recherche afin de développer des médicaments innovants et plus efficaces et d’intégrer, dès le stade de recherche, la prévention, le diagnostic et le traitement des maladies. C’est dans ce cadre de recherche translationnelle et de médecine moléculaire que les nouvelles technologies développées après le séquençage du génome (ère post-génomique) apportent des outils permettant de mieux appréhender la complexité des pathologies chez l’homme. Elle regroupe les technologies de séquençage, de génétique moléculaire, de génomique fonctionnelle, de protéomique, de biologie structurale, de métabolomique, ainsi que les technologies d’imageries (IRM, SPECT, single photon emission computed tomography, PET, positron emission tomography) soutenues par de nouvelles méthodes d’analyse en bio-informatique, en biostatistique et en modélisation mathématique.

Ces approches permettent principalement de décrire, et par conséquent, de décortiquer l’entièreté d’un système biologique, allant du gène à son expression finale, la fonction. Elles permettent de mieux comprendre le mécanisme associé à la réponse aux médicaments (efficacité et effets secondaires) chez le patient et éventuellement de prédire les différentes sources de variabilité entre les individus. Ces nouvelles connaissances sont des sources importantes de propriétés intellectuelles.
Il faut avoir à l’esprit que les fonctions biologiques, qu’elles soient normales, pathologiques ou modulées par un médicament, sont la résultante d’une régulation coordonnée et dynamique de centaines de gènes ayant des répercussions sur l’expression, les modifications post-translationnelles, la localisation et la diversité de fonction des protéines. Par conséquent, les interprétations de ces processus, que l’on pourrait qualifier de « classiques » et canoniques, restent nécessairement des simplifications à la lumière des connaissances acquises ces dernières années. De plus, la multiplicité fonctionnelle des protéines ne peut s’imaginer que dans un flux permanent, dépendant de leur contexte cellulaire/tissulaire, de l’état physiologique/pathologique, des modifications post-traductionnelles, ou de leur interaction avec d’autres protéines.
C’est pourquoi une empreinte moléculaire obtenue à partir d’une analyse des ARN (transcriptome), des protéines (protéome) ou des métabolites (métabolome), ne représente qu’un instantané d’un phénomène biologique. La survenue de ces acteurs cellulaires est de plus échelonnée dans le temps et c’est pourquoi ils doivent nécessairement être mis en regard d’autres paramètres (qualitatifs et quantitatifs) comme les fonctions anatomiques, biochimiques, phénotypiques, cliniques et fonctionnelles du système étudié. On comprend alors mieux pourquoi il est nécessaire d’avoir une compréhension globale d’un système biologique dans une approche qualifiée aujourd’hui de biologie de système (systems biology). Elle représente en fait l’intégration de ces disciplines et de ces expertises nouvelles avec des connaissances plus établies et surtout la compréhension de leur imbrication.
Intégration des données
Ces nouvelles technologies ne peuvent cependant à elles seules résoudre tous les problèmes et si elles permettent indéniablement de mieux apprivoiser la complexité des processus en jeu, elles doivent nécessairement être appliquées en parallèle à d’autres approches et être à la portée (utilisable et compréhensible) des différents partenaires impliqués dans le projet.
En effet, la puissance de ces technologies en termes de genèse de données et d’informations oblige à être encore plus précis et plus rigoureux dans leur application et leur interprétation.
La première règle d’or est de préciser l’objectif scientifique que l’on veut atteindre et de déterminer si l’approche proposée permet de répondre à la question posée. L’intégration de ces méthodes doit se faire lors de la mise en place du plan d’expérience permettant de définir les analyses statistiques les plus appropriées.
La deuxième règle d’or est la mise en place d’une bonne organisation et d’un tri intelligent de ces données. En effet la quantité vertigineuse de données publiées quotidiennement sur ces sujets dans la recherche biomédicale (~ 1 700 publications par jour sont publiées dans PubMed) et la masse de données générées par des nouvelles technologies, nécessitent la mise en place d’outils de tri et de synthèse ainsi que des bases de données permettant de gérer, organiser, comparer, fédérer et analyser ces informations via des processus standardisés.
La bonne construction de cette architecture relationnelle est vitale pour une bonne exploitation et une bonne valorisation des données. Elle a souvent été négligée par le passé, mais représente la clé de voûte de l’approche des systèmes biologiques, bien avant les techniques d’analyses de laboratoire elles-mêmes.
Il en est de même pour les nouvelles méthodes d’analyses et d’expertises en biostatistique, mathématique de modélisation et en bio-informatique qui sont essentielles pour transformer ces informations en connaissances moléculaires utilisables.
Malgré les efforts pour mettre en place une organisation rationnelle, ces avancées technologiques peuvent donner quelque peu le tournis car elles vont au-delà des barrières des disciplines traditionnellement acceptées et forcent les scientifiques à trouver une nouvelle manière de travailler et de partager l’information. Cette évolution implique aujourd’hui, encore plus qu’hier, une discussion, une explication et une information quant au potentiel de ces approches et c’est dès la mise en place d’un nouveau projet thérapeutique que ces rôles et ces interconnexions de disciplines doivent être définis.
Finalement, la « validation » d’une nouvelle cible thérapeutique a souvent à tort été assimilée à un puzzle, où il fallait attendre la mise en place de la dernière pièce (fin de la phase 3), avant de valider son rôle dans le processus physiopathologique de la maladie principalement par le biais de l’efficacité ou de l’inefficacité du candidat médicament.
Il faut se rappeler que beaucoup de médicaments n’ont pas de cibles réelles identifiées et dans certains cas les composés ont des cibles multiples qui exigent une optimisation dès le stade de la recherche afin d’obtenir une efficacité et une sécurité clinique optimales.
De plus, la complexité de certaines maladies dégénératives et la limite actuelle des connaissances des processus moléculaires impliqués rendent extrêmement difficile le développement des médicaments ayant un impact majeur sur l’étiologie ou sur la progression de la maladie et en conséquence, limitent les remèdes à des traitements symptomatiques.
Or, si l’accès au tissu d’origine humaine (normal/pathologique) via des biobanques, la recherche clinique, les études médico-épidémiologiques, les essais cliniques et les observations médicales servent à enrichir notre connaissance des maladies, cela ne peut qu’aller de pair avec une meilleure connaissance des processus moléculaires à l’origine des maladies ainsi que de l’identification des cibles qui jouent un rôle majeur dans le processus physiopathologique. Cette démarche permet de faire émerger de nouvelles hypothèses et de consolider la pertinence du choix d’une cible pour un futur médicament, augmentant ainsi les chances de succès d’avoir un impact sur la pathologie et l’état clinique du patient. Cette approche offre l’opportunité d’identifier les sous-populations de patients susceptibles de mieux bénéficier du traitement.
Pour relever le défi de l’intégration et de l’application de ces nouvelles technologies au sein du groupe Servier, il a été créé un ensemble de plates-formes, véritables « boîtes à outils » technologiques, mises à la disposition des partenaires de la Recherche et Développement (Figure 1). Au sein de la recherche, la division de pharmacologie et physiopathologie moléculaires fédère de multiples expertises et technologies couvrant les domaines de la génomique, de la protéomique, de la bio-informatique et de la pathologie moléculaire. Elle a pour objectif d’améliorer la validation du choix des cibles thérapeutiques. Elle permet une meilleure caractérisation et un suivi des modèles précliniques afin d’améliorer leur pouvoir prédictif par rapport à la pathologie humaine. Ces outils permettent le développement et l’intégration de biomarqueurs pharmacodynamiques au sein des projets de recherche et d’approfondir la connaissance du mécanisme d’action moléculaire de nos futurs médicaments.
 | Figure 1.
Fédération au sein du groupe Servier de l’ensemble des expertises et technologies nécessaires à une démarche translationnelle. Elle couvre les domaines de la génomique, de la protéomique, de la biochimie métabolique, de la bio-informatique et la pathologie moléculaire. |
L’intégration de ces nouvelles technologies dans les programmes de développement de nouveaux médicaments se poursuit dans les domaines de la biopharmacie et de la clinique. Elle concerne notamment la sécurité d’emploi des médicaments (toxicogénomique), le métabolisme, avec la biochimie métabolique (métabolomique), qui permettent de suivre les modifications métaboliques exogènes et endogènes induites ou reliées au traitement ainsi que les études de pharmacogénétiques pour la caractérisation des variabilités de réponses aux traitements.
Cette synergie de compétences et de moyens est essentielle pour mieux apprivoiser la complexité du système biologique du gène jusqu’aux effets fonctionnels moléculaires physiologiques et/ou pathologiques. Mais cette démarche d’innovation thérapeutique et de recherche exploratoire doit être synchronisée avec les programmes de recherche afin de mieux comprendre les mécanismes en jeu et à terme de faciliter la prise de décision dans l’avancée des projets.
Le pouvoir prédictif des modèles animaux est au centre de nos préoccupations car il est un des éléments fortement liés aux échecs en clinique [
4–
7]. C’est pourquoi nous étudions très en amont le tissu pathologique humain ce qui nous permet, dès le stade de découverte, de valider le rôle fonctionnel de la cible dans la pathologie humaine et de mieux sélectionner et développer les modèles précliniques. Nous nous appliquons également à déterminer si ces modèles animaux sont adaptés à la cible moléculaire étudiée (relation cible, processus physiopathologique et symptomatologique). Souvent, il nous manque de nombreux éléments fondamentaux permettant de caractériser ces modèles. Il faut donc déterminer des facteurs aussi divers que le niveau d’expression, la régulation et le rôle fonctionnel de la cible, les études d’homologies entre protéines, l’extrapolation entre espèces (orthologie), les variations génétiques qui influent sur la fonction ainsi que la localisation tissulaire et cellulaire de ces cibles.
Mais la compréhension de la similarité/dissimilarité entre espèces ne s’arrête pas au niveau moléculaire, il faut y intégrer la dimension histopathologique. C’est pourquoi nous pensons que les expertises en histologie et anatomopathologie sont un atout majeur de cette stratégie. L’introduction des modèles animaux (pathology-driven) par l’utilisation de modèles transgéniques, qui reproduisent aux niveaux moléculaire et pathologique la génétique, l’étiologie et le développement de la maladie, est une voie d’amélioration des modèles « génériques » très utilisée dans la recherche biomédicale [
8].
Le domaine du diabète est caractéristique de cette démarche d’amélioration de connaissances du modèle animal. Par exemple, les souris db/db présentent une mutation dans le gène pour le récepteur de la leptine et développent plusieurs phénotypes et perturbations métaboliques typiques du diabète de type 2 comme l’hyperglycémie, l’hyperinsulinémie, l’hyperlipidémie et la résistance à l’insuline [
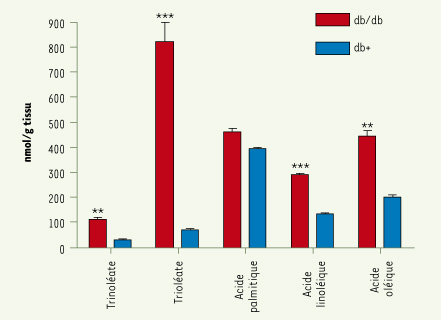
9]. En effet, nous avons montré de très importantes modifications transcriptionnelles avec un changement d’expression d’environ 4 000-8 000 gènes en fonction du tissu analysé entre les db (animaux témoins) et les db/db (animaux pathologiques) (Tableau I). Si ce véritable embrasement transcriptionel est lié pour une part au processus physiopathologique, de nombreuses modifications concernent aussi des mécanismes d’adaptation de l’organisme à la progression de la pathologie. Il est à noter une augmentation significative des taux de plusieurs triglycérides, notamment du trilinoléate, trioléate, acide linoléique et de l’acide oléique du foie chez les souris db/db par rapport aux souris db+ (Figure 2). Une analyse conjointe des données de métabolomique (suivi des fonctions métaboliques) et de transcriptomique (modifications dans les transcriptions de gènes) nous permet de faire l’hypothèse suivante : l’augmentation de la densité de triglycérides et d’acide gras libres de souris db/db versus db+, dans le foie, pourrait être associée à l’augmentation des ARNm des gènes codant des transporteurs d’acides gras (CD36 ; × 13,6), ou des enzymes impliquées dans la voie de synthèse des acides gras telles que l’acétyl-CoA carboxylase a (ACACA ; × 1,5), l’enzyme de synthèse des acides gras (FAS ; × 2) et des triglycérides tels que la glycérol 3-phosphate déshydrogénase 3 (Gpd1 ; × 1,5) et l’acylglycérol-3- phosphate O- acyltransférase (Agpata2 ; × 1,6). Il est donc important de se focaliser sur des processus cellulaires et moléculaires associés aux manifestations fonctionnelles de la pathologie (insulinémie, stéatose, glycémie, etc.) et de faire le lien avec la cible thérapeutique étudiée dans ce modèle animal.
 | Figure 2.
Profil métabolomique des triglycérides au niveau du foie des souris diabétiques (db/db) et des souris témoins (db+) à l’âge de 12 semaines. Données : n = 6 animaux/groupe (moyenne ± ESM) ; test de Student (**p > 0,01, ***p < 0,001). |
 | Tableau I.
Changements d’expression tissulaires des ARN (modifications transcriptionnelles - augmentation [induction] ou diminution [répression]) entre les souris diabétiques (db/db) et les souris témoins (db+) à 12 semaines. Données : n = 6 animaux/groupe. Nombre de séquences : facteur de régulation > 1,5 ; p < 0,01. |