| |
| Med Sci (Paris). 2009 May; 25(5): 497–504. Published online 2009 May 15. doi: 10.1051/medsci/2009255497.Mécanismes de défense du nématode C. elegans
Katja Ziegler* and Nathalie Pujol* Centre d’immunologie de Marseille-Luminy (CIML), Inserm U631, CNRS, UMR6102, Université de la Méditerranée, Campus Luminy, Case 906, 13288 Marseille Cedex 9, France |
C. elegans est un nématode non parasitaire vivant dans le sol, souvent trouvé sur des fruits en état de décomposition [
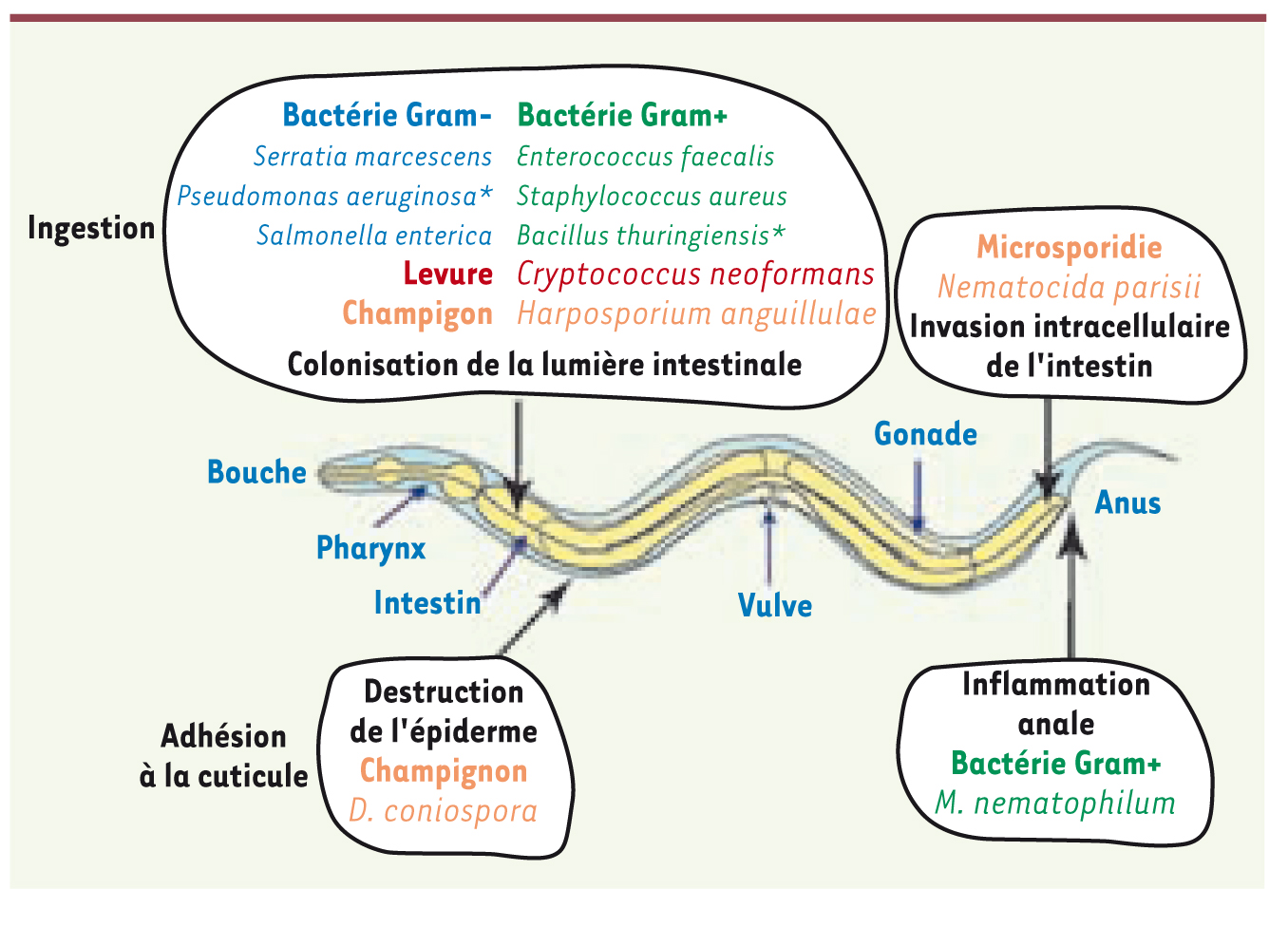
1]. Il se nourrit de bactéries et est constamment exposé à de multiples pathogènes potentiels (Figure 1).  | Figure 1.
Schéma d’un nématode adulte et des différents modes d’infection.
La plupart des pathogènes sont ingérés et établissent une infection dans la lumière intestinale. Certaines bactéries peuvent tuer le ver en libérant des toxines (*). La bactérie M. nematophilum et le champignon D. coniospora adhèrent à la cuticule et infectent C. elegans par son épiderme. La liste des pathogènes n’est pas exhaustive. |
Certains pathogènes, bactéries à Gram négatif, Gram positif ou levures (comme Cryptococcus neoformans) sont ingérés comme source de nourriture et provoquent une infection dans l’intestin. D’autres bactéries produisent des toxines, comme Bacillus thuringiensis ou Pseudomonas aeruginosa dans certaines conditions de culture. Microbacterium nematophilum adhère à la région anale et provoque un type de gonflement de l’épiderme [
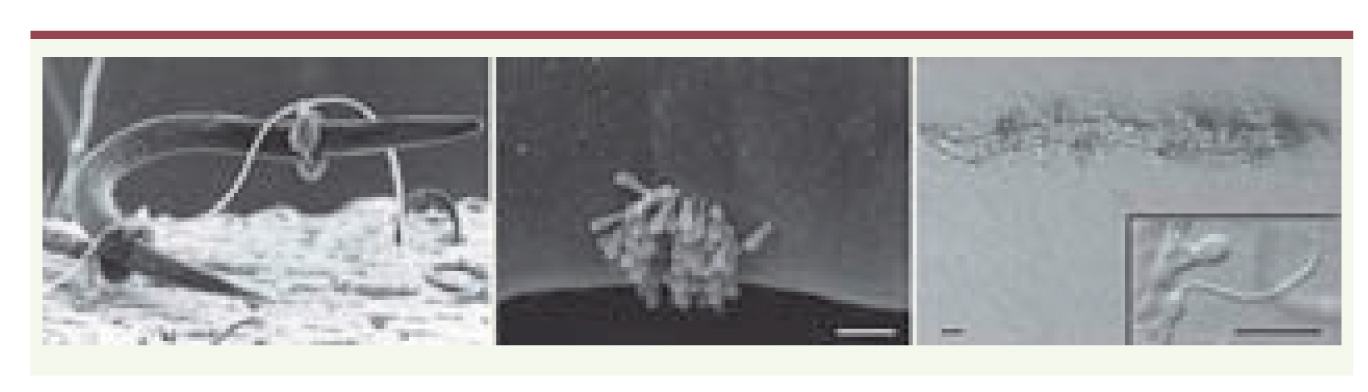
2]. Des champignons ont, eux, élaboré une variété remarquable de pièges pour nématodes, en forme de lasso constricteur (Drechslerella), d’hameçon (Harposporium) ou de simples quilles adhésives, comme Drechmeria coniospora (Figure 2). Un autre eucaryote de type microsporidie, identifié récemment, est un parasite intracellulaire naturel du nématode, Nematocida parisii [
3].  | Figure 2.
Illustration de divers types d’attaque de champignon nématophage. Drechslerella anchonia à gauche (Pr. G. Barron, University of Delph, Canada), Drechmeria coniospora au centre (Jürgen Berger, MPI for Developmental Biology, Tübingen, Germany), Harposporium anguillulae. Échelle : 10 µm. |
Comme c’est le cas pour tous les invertébrés, la défense contre les pathogènes de C. elegans dépend uniquement de son système immunitaire inné. Le ver possède trois principaux mécanismes de défense. (1) Le premier est un comportement de fuite. Alors que la plupart des bactéries, sources potentielles de nourriture, attirent le nématode, certaines bactéries pathogènes provoquent au contraire un comportement d’évitement. Cette réponse de fuite est déclenchée par les neurones olfactifs et implique l’unique TLR (toll-like receptor) de C. elegans, TOL-1 [
4] et une voie de signalisation en aval des récepteurs couplés aux protéines G (GPCR,G protein coupled receptor) [
5]. (2) Des barrières physiques représentent le second mécanisme de défense. La cuticule, exosquelette du ver, formée de collagène et de chitine, est une première ligne de défense. Autre barrière, lors de l’ingestion de nourriture, le broyeur du pharynx empêche les bactéries ou levures de pénétrer intactes dans l’intestin (Figure 1), et des mutants dont le broyeur est défectueux sont plus sensibles aux infections bactériennes [
6,
7]. (3) Enfin, C. elegans met en jeu des mécanismes inductibles de défense que nous discutons ci-dessous. Il faut noter que, contrairement aux insectes ou aux mammifères, C. elegans ne possède pas d’immunité cellulaire. Les cœlomocytes, seules cellules libres dans la cavité du corps, ne semblent pas capables de phagocyter des bactéries [
8]. |
Reconnaissance des pathogènes Les récepteurs dédiés à la reconnaissance des pathogènes sont appelés des PRR pour pattern recognition receptor. Les gènes codant pour des familles principales de PRR comme PGRP (peptidoglycan recognition proteins) ou GNBP (Gram-negative binding proteins) chez la drosophile ou NOD (nucleotide-binding oligomerization domain) et les protéines à domaine NACHT chez les mammifères [
9] sont absents du génome de C. elegans. Le seul TLR présent chez le ver ne semble pas directement impliqué dans la résistance aux infections [4]. Chez les mammifères, certaines lectines de type C comme la Dectine-1 ont été impliquées dans la reconnaissance de β-glucan exprimé dans la paroi des champignons [9]. Il y a 278 gènes codant des lectines de type C chez le nématode, dont l’expression de certaines est modifiée lors d’infections par différentes bactéries [
10]. La façon dont le nématode perçoit l’infection reste à découvrir, mais il est clair que selon la nature de l’infection, C. elegans développe une réaction de défense ciblée [
11–
13]. Une étude récente faite en parallèle chez le nématode et la souris propose une liaison directe des récepteurs scavenger (poubelle), CED-1/SCARF1 (C. elegans) et C03F11.3/CD36 (souris) aux β-glucanes de la paroi des levures Candida albicans et neoformans [
14,
46] (→) Par ailleurs, un mutant du gène fshr-1 codant un GPCR à motif LRR apparaît plus sensible à l’infection par les bactéries à Gram positif et négatif, et exprime plus faiblement plusieurs gènes effecteurs de la réponse immunitaire. Les auteurs proposent l’hypothèse selon laquelle le récepteur FSHR-1 (un analogue du mammalian follicle stimulating hormone receptor) pourrait soit agir comme un récepteur hormonal aidant la réponse immunitaire, soit servir directement de PRR [
15]. (→) Voir l’article de Daniel Poulain et al., p. 473 de ce numéro
La perception de l’infection se fera probablement via la reconnaissance directe des pathogènes ou de leurs facteurs de virulence, mais aussi des dommages cellulaires et du stress causés par les pathogènes [11,
16], selon la théorie du danger [
17], comme cela a été montré chez les plantes, la drosophile [
18] et les mammifères [
19]. |
Plusieurs cascades de signalisation sont activées en fonction du pathogène conduisant soit à la production de molécules effectrices, par exemple les lysozymes, qui pourront détruire les pathogènes, soit à d’autres réponses cellulaires qui protégeront l’organisme, telles que la production de ROS (reactive oxygen species) [
20] ou la réponse UPR (unfolded protein response) [
21]. Impliquées dans l’immunité des animaux et des plantes, les voies mettant en jeu les MAPK (mitogen-activated protein kinase) sont considérées comme les plus anciennes cascades de transduction du signal. Chez C. elegans, un crible génétique à partir de mutants hypersensibles à l’infection par P. aeruginosa a révélé un rôle pour la voie de la p38 MAPK dans la défense antimicrobienne [
22]. Depuis, il a été montré que cette voie était impliquée dans la protection contre d’autres bactéries à Gram négatif et positif et semble être l’une des principales cascades de signalisation intervenant dans la réponse immunitaire innée [
23]. Une autre voie MAPK, la voie ERK (extracellular signal-regulated kinase) est impliquée dans la résistance à l’infection par la bactérie à Gram positif M. nematophilum [
24]. Enfin, la MAPK MEK-1 de la voie JNK (c-Jun N-terminal kinase) est nécessaire à l’activation complète de la p38, révélant une interaction entre les différentes voies MAPK [
25]. Plusieurs études transcriptionnelles ont été réalisées comparant des vers infectés par diverses bactéries. La réponse du ver à une infection par S. marcescens a permis de mettre en évidence l’induction de plusieurs molécules effectrices telles que des lectines et certains lysozymes, la voie du TGF-β (transforming growth factor-β) étant nécessaire à cette induction [
26]. La voie de l’insuline impliquant le récepteur DAF-2 et le facteur de transcription de type FOXO DAF-16 (un membre de la famille forkhead transcription factor) sont impliqués dans beaucoup de processus biologiques chez le nématode, notamment la longévité. Garsin et al. ont montré que les mutants daf-2 sont résistants à l’infection par E. faecalis, S. aureus et P. aeruginosa [
27]. Mais des études récentes comparant des profils d’expression de gènes, sous le contrôle de daf-16 d’une part, et après infection par diverses bactéries d’autre part, révèlent peu de gènes communs, suggérant que daf-16 régulerait l’expression de gènes impliqués dans la défense constitutive du nématode contre les pathogènes [16, 23]. Enfin, la voie de la mort cellulaire programmée (PCD) semble protéger C. elegans contre l’infection par S. typhimurium [
28]. |
Une absence remarquée : celle de NF-κB Chez les insectes et les vertébrés, le facteur de transcription NF-κB représente le lien principal entre la réception et la transmission du signal d’infection et l’activation des effecteurs. Son absence chez le ver est étonnante mais ouvre une fenêtre d’investigation sur d’autres mécanismes de transcription potentiellement conservés chez les autres espèces. Une grande partie des effecteurs induits dans l’intestin en réponse à P. aeruginosa est sous le contrôle du facteur de transcription de type GATA ELT-2 [
29]. Le groupe d’Aballay a montré qu’une élévation de température augmentait la résistance du ver en activant le facteur de transcription HSF-1 (heat shock factor) [
30]. Récemment, le cofacteur de transcription BAR-1/β-caténine a été démontré comme étant nécessaire à la résistance contre S. aureus [
31]. |
Modulation par le système nerveux Deux articles récents fournissent la preuve d’une interaction chez C. elegans entre le système nerveux et le système immunitaire. Kawli et Tan ont démontré que la sécrétion des DCV (dense core vesicles) par les neurones régule négativement la réponse immunitaire intestinale contre P. aeruginosa et que ce mécanisme de contrôle neuronal est transmis par la voie de l’insuline dans l’intestin [
32]. Styer et ses collègues ont montré par ailleurs que NPR-1, un GPCR apparenté au récepteur du neuropeptide Y chez les mammifères, régule négativement la réponse à P. aeruginosa, probablement en agissant en amont de la voie de p38 MAPK [
33]. Mais cette étude est controversée, une autre équipe ayant attribué la réduction de la susceptibilité dans la souche mutante npr-1 à un phénomène mécanique. Les mutants npr-1 passent en effet plus de temps en groupe et seraient plus en contact avec les bactéries pathogènes [
34]. |
La réponse antifongique chez C. elegans
La plupart des pathogènes bactériens décrits précédemment infectent le ver en envahissant la lumière de l’intestin dont la fonction naturelle est la digestion des bactéries. Il est de ce fait difficile de séparer digestion et réponse immunitaire de l’hôte. Notre équipe étudie surtout un autre type de pathogène qui attaque le ver par l’extérieur à travers sa cuticule. Le champignon Drechmeria coniospora infecte le nématode à travers l’épiderme Le champignon D. coniospora est un parasite nématophage strict. Il produit des spores en forme de petites quilles. La base renflée appelée bouton adhésif s’attache sur la cuticule du ver, notamment au niveau des interfaces entre l’organisme et le milieu environnant comme la bouche, la vulve et l’anus (Figure 2).
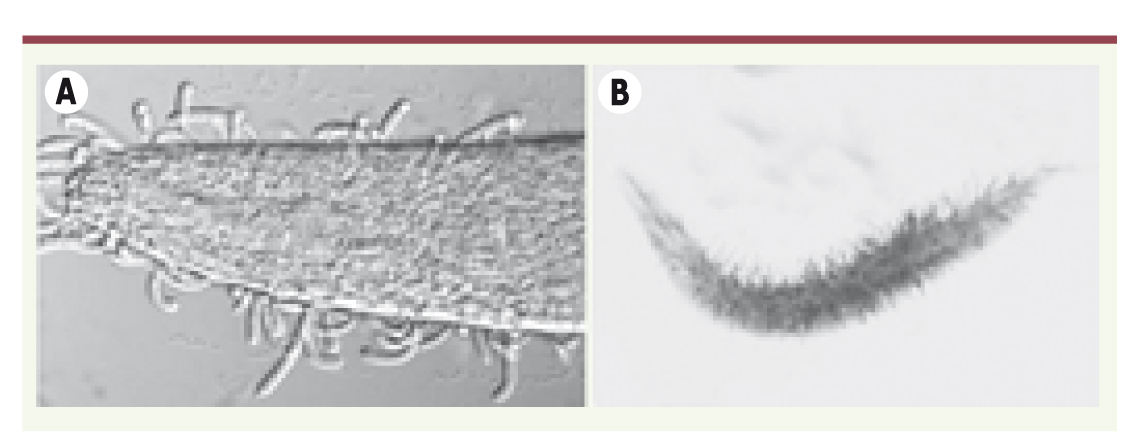
Des enzymes sécrétées par le champignon vont permettre de percer la cuticule et d’envoyer des hyphes qui vont croître dans l’épiderme et envahir tout le ver [
35]. L’infection est rapide, le nématode peut être tué en 48 heures (Figure 3).
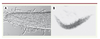 | Figure 3.
Infection de C. elegans par D. coniospora. Après 48 heures d’infection, les hyphes ont envahi le ver et commencent à sortir du nématode (A), le ver n’est plus qu’un cadavre poilu (B), les hyphes commencent à germer en conidia qui seront dispersées prêtes à établir une nouvelle infection. |
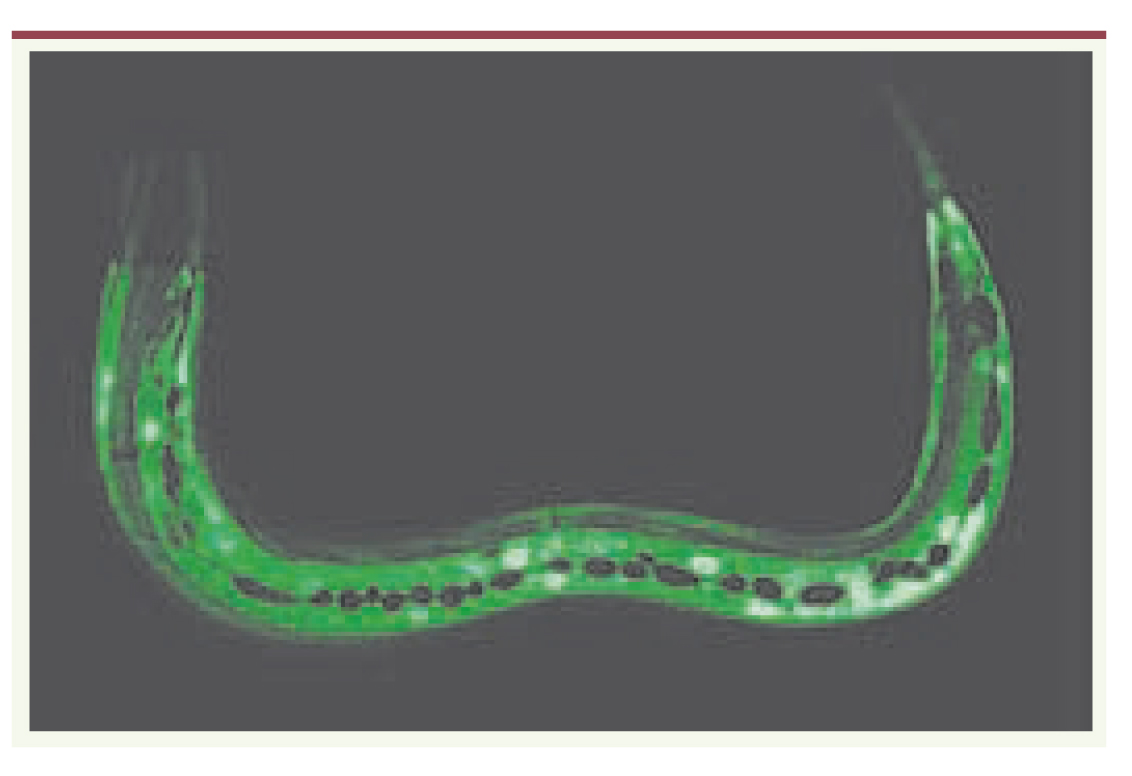
Réponse transcriptionelle de C. elegans à l’infection fongique Une étude d’expression différentielle par puces à ADN a montré que, en réponse à une infection par D. coniospora, le nématode induit l’expression de plusieurs gènes codant une famille de peptides riches en glycine et tyrosine appelée NLP, pour neuropeptide-like protein et CNC pour caenacin. Des études d’expression à l’aide de gènes rapporteurs comme la GFP ( green fluorescent protein) ou la DsRed ont montré que la majorité de ces gènes étaient induits après l’infection et exprimés dans l’épiderme du ver, site de l’attaque du champignon (Figure 4) [ 13,
36].  | Figure 4.
Expression du gène nlp-31.
Image en microscopie confocale d’une jeune larve exprimant la GFP (green fluoresenct protein) dans l’épiderme sous le contrôle du promoteur du gène nlp-31.
|
Amplification et sélection positive des gènes nlp et cnc
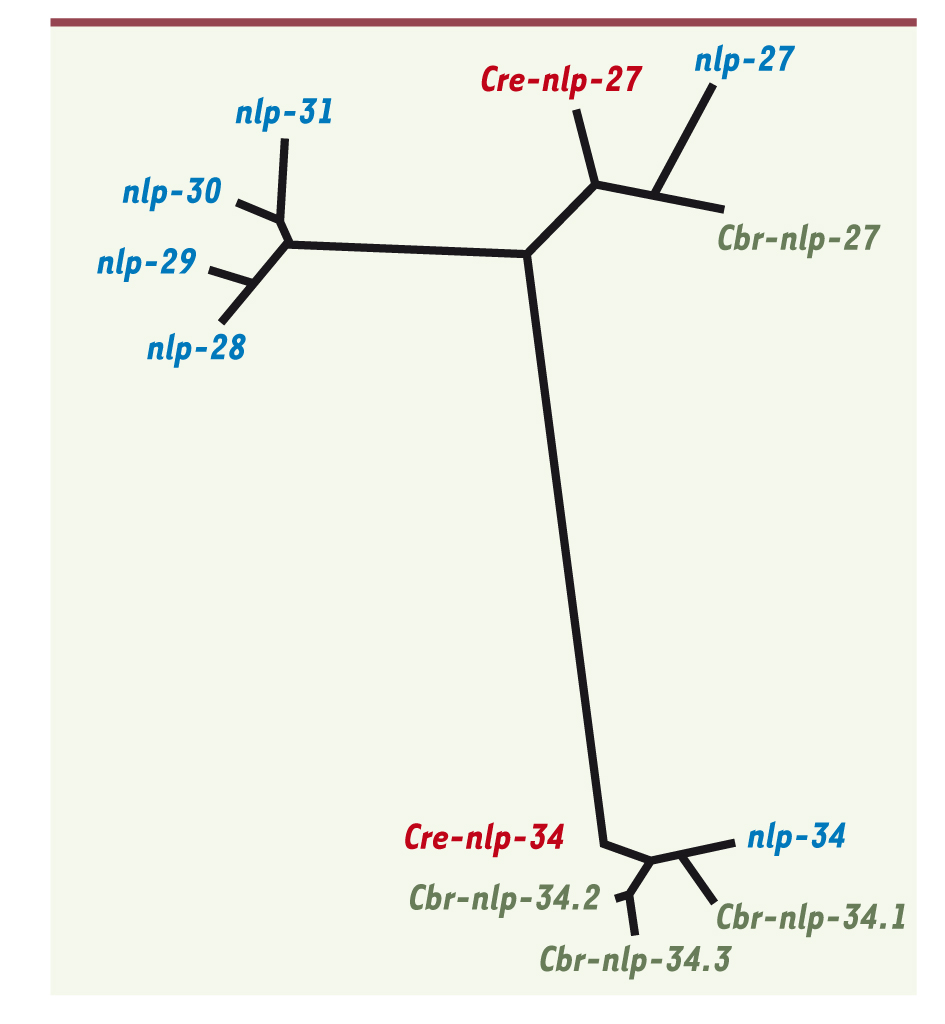
La majeure partie des gènes nlp et cnc induits après infection est positionnée dans le génome en deux groupes distincts, appelés groupe nlp-29 et cnc-2, sur le chromosome V. L’identification de groupes synténiques dans deux espèces de nématodes, C. briggsae et C. remanei, montre que lors de la divergence entre les espèces, il y avait probablement deux gènes dans le groupe nlp-29 et que l’un d’eux, nlp-27, a subi une expansion chez C. elegans, l’autre, nlp-34, chez C. briggsae. Des études d’adaptations évolutives basées sur le taux de remplacement de bases provoquant des mutations versus des remplacements silencieux, faites en collaboration avec H. Schulenberg (Kiel, Allemagne), suggèrent en effet une évolution adaptative positive du groupe nlp-29 [ 13] (Figure 5). La théorie contemporaine sur l’évolution des gènes de l’immunité innée propose que l’hôte et son pathogène partageant le même habitat fassent la « course » pour essayer d’échapper aux innovations de l’autre [
37]. Une amplification par duplication en tandem et une diversification de gènes apparaissent comme les premières armes pour faire face aux stratégies échappatoires des pathogènes. 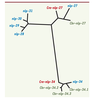 | Figure 5.
Arbre phylogénétique des gènes du locus nlp.
Chez C. elegans (bleu), C. briggsae (vert) et C. remanei (rouge). C. remanei représente probablement la situation ancestrale avec deux gènes nlp-27 et nlp-34, alors qu’une amplification de nlp-27 s’est produite chez C. elegans et de nlp-34 chez C. briggsae [ 13]. |
Les peptides des groupes nlp-29 et cnc-2 ont des propriétés antimicrobiennes Un des peptides du groupe nlp-29, NLP-31, a été synthétisé et son activité antifongique a été démontrée in vitro. De plus, lorsque des vers infectés par D. coniospora sont incubés avec ce peptide, celui-ci est capable d’arrêter la sporulation du champignon [ 36]. Enfin, des vers transgéniques surexprimant des peptides du groupe entier nlp-29 ou cnc-2 sont plus résistants à l’infection fongique [ 13,
38], ce qui confirme une activité antimicrobienne in vivo. Nous désignerons ces peptides sous le nom de peptides antimicrobiens (AMP). Une blessure de l’épiderme induit l’expression des gènes AMP
Comme l’infection par le champignon implique l’ouverture d’une brèche dans la cuticule et l’épiderme du ver, nous avons testé la possibilité qu’une piqûre stérile puisse provoquer l’induction de l’expression des gènes AMP. En réponse à une simple piqûre avec une aiguille fine ou en utilisant un faisceau laser au niveau de l’épiderme, certains gènes AMP sont rapidement induits ; c’est le cas de la majorité des gènes du groupe nlp-29 et de certains gènes du groupe cnc-2. Par ailleurs, une expression constitutive forte de ces gènes est observée dans certains mutants présentant des défauts développementaux de l’épiderme. Cette expression pourrait protéger le nématode contre d’éventuelles attaques à travers un épiderme fragilisé [ 13,
39,
40]. Il en est certainement de même dans la peau des mammifères où une piqûre stérile provoque l’induction d’AMP [
41]. La voie de la p38 MAPK contrôle l’expression des gènes AMP du groupe nlp-29 dans l’épiderme Les molécules à domaine TIR ( Toll-interleukin 1 receptor) étant connues pour leur implication dans l’immunité innée [
42], nous avons testé le rôle des deux protéines contenant un tel domaine : le récepteur TLR TOL-1 et la molécule cytoplasmique TIR-1, homologue de SARM chez les mammifères. Si l’induction des gènes AMP est indépendante de TOL-1, TIR-1 régule uniquement le groupe de gènes nlp-29. Des vers transgéniques surexprimant le gène sek-1 (p38 MAPKK) spécifiquement dans l’épiderme sont capables de sauver les mutants sek-1 mais aussi tir-1 et nsy-1 (p38 MAPKKK). Cela montre que tir-1 agit en amont de la voie de la p38/MAPK, comme lors de l’infection par le pathogène intestinal P. aeruginosa [
43], et que cette voie agit de façon intrinsèque à la cellule. L’induction de ces gènes par la piqûre est aussi régulée par la même voie [ 39]. Une kinase de type Tribbles est spécifiquement requise pour la réponse à l’infection Une approche non biaisée par crible génétique a permis d’identifier plusieurs gènes responsables de l’induction des peptides en isolant des mutations récessives produites au hasard lors d’une mutagenèse par un agent chimique, mutations qui bloquent l’induction du peptide nlp-29 en réponse à l’infection (Figure 6). L’un de ces mutants, nipi-3, est particulièrement intéressant car il bloque la réponse à l’infection mais pas à la piqûre. Des études épistatiques ont montré que nipi-3 agissait en amont de la voie TIR-1/p38 dans l’épiderme. nipi-3 code une kinase homologue à Tribbles qui, chez les mammifères, agit aussi en amont de la voie de la p38 [
44]. Ces données renforcent encore la réalité d’une conservation des molécules et des voies de signalisation intervenant chez le nématode et les mammifères et révèlent que les deux processus d’induction des peptides après piqûre et après infection sont contrôlés séparément au niveau génétique [ 39].  | Figure 6.
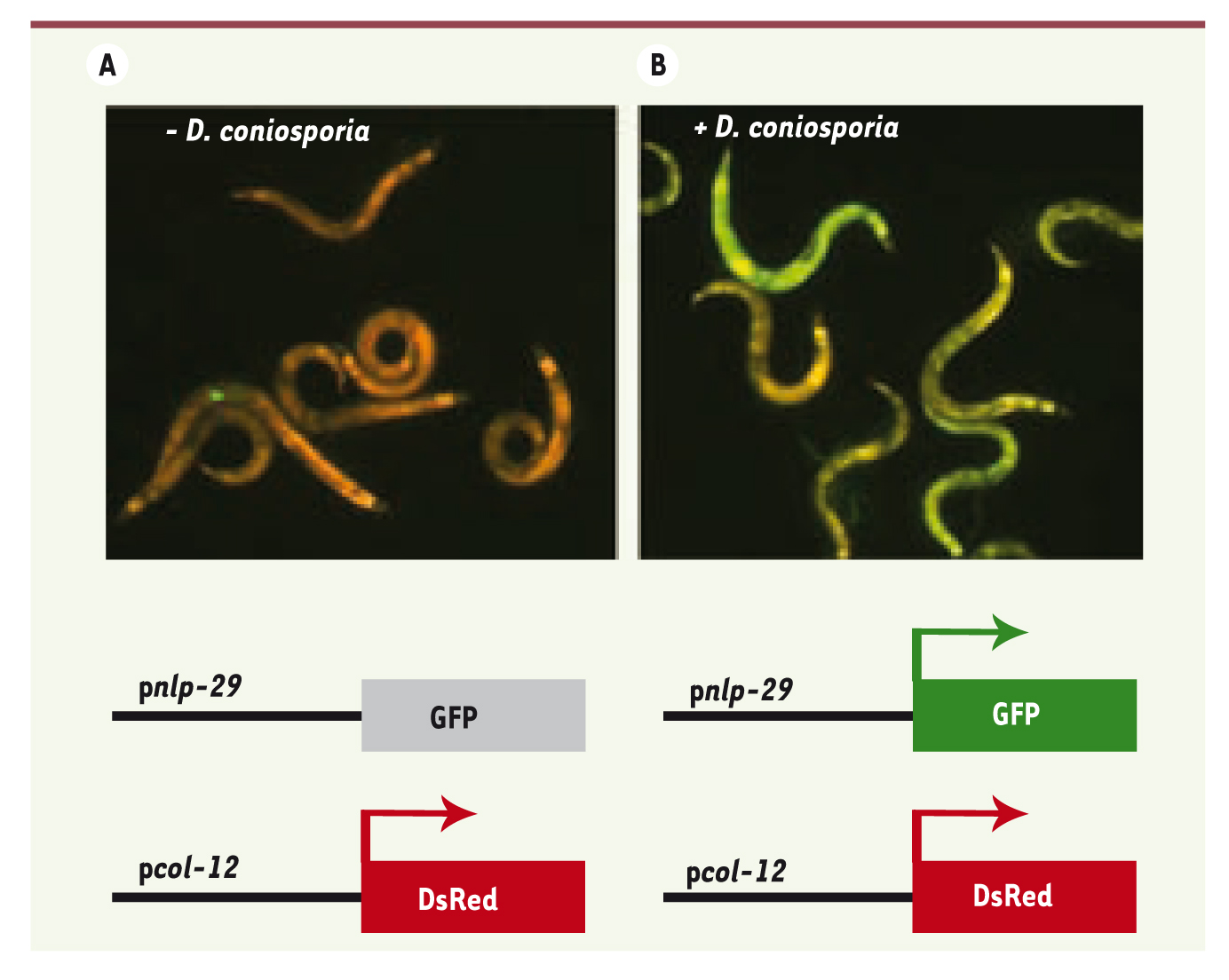
Illustration de l’induction des gènes nlp-29 après infection.
Une souche transgénique, contenant un promoteur contrôlant la DsRed exprimé de façon constitutive dans l’épiderme et le promoteur inductible du gène nlp-29 contrôlant la GFP, est rouge en l’absence d’infection (gauche) et devient verte après 12 heures d’infection par D. coniospora (droite). |
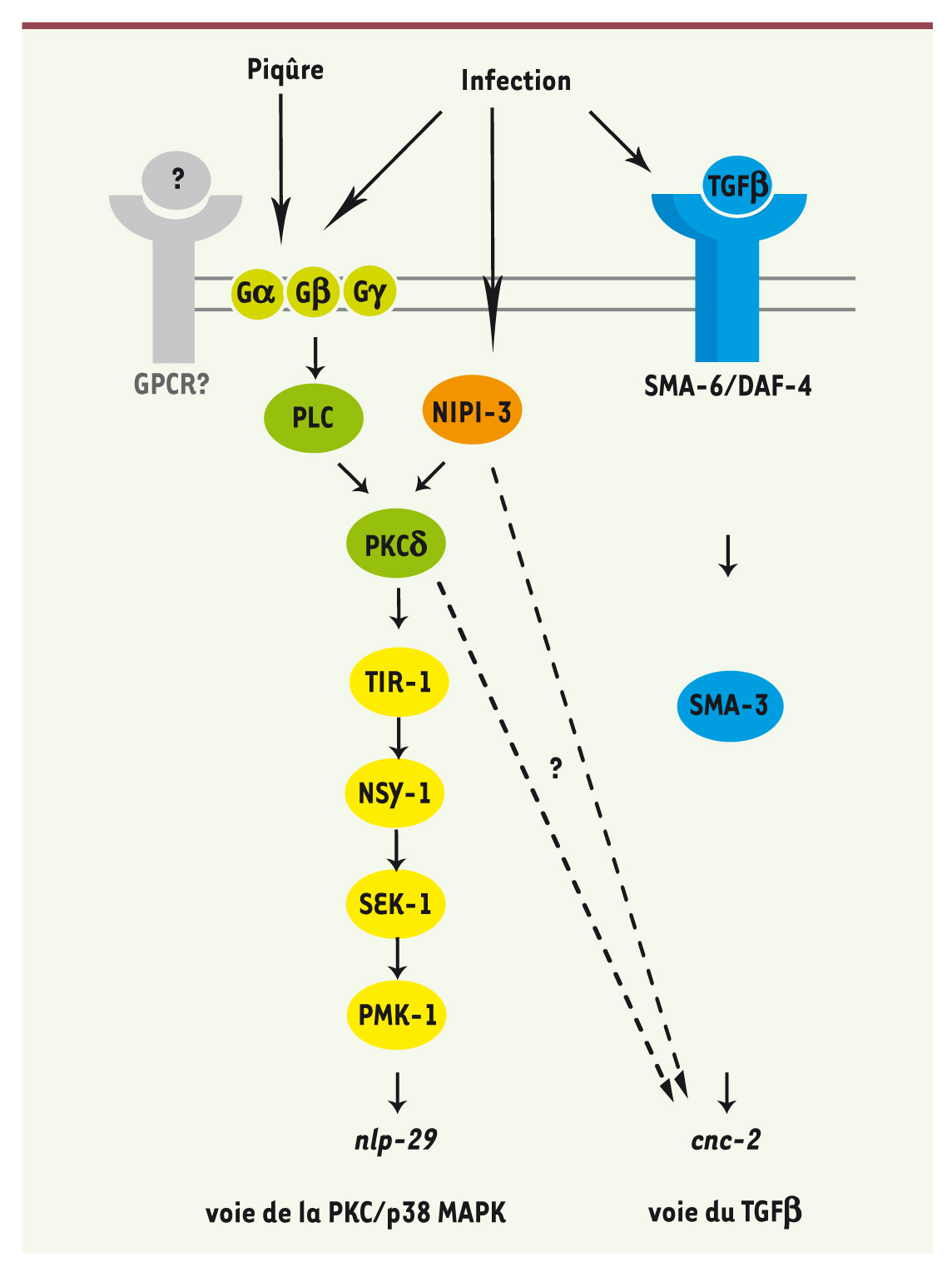
La protéine kinase C fait le lien entre la signalisation des protéines G et la voie de la p38 MAPK Lors du même crible génétique, l’identification de deux allèles du gène tpa-1 codant la protéine kinase C (PKCδ) a permis de révéler une cascade de signalisation, passant par la protéine Gα (GPA-12) et les phospholipases (EGL-8 et PLC-3), en amont de la PKCδ (TPA-1) dans la régulation de l’expression du gène nlp-29 en réponse à une piqûre ou une infection. Les GPCR qui réguleraient cette voie n’ont pas été identifiés, il y en a plus de 1 200 chez le ver. Les deux GPCR récemment impliqués dans l’infection bactérienne, FSHR-1 et NPR-1, sont des candidats à tester dans le futur. Des études épistatiques suggèrent que l’activation par les protéines G agit en amont de la voie de la p38 et que NIPI-3 relaierait le signal spécifique de l’infection qui converge avec le signal de la piqûre sur la cascade PKC-TIR-1-p38 MAPK (Figure 7) [
45]. Ces résultats sont cohérents avec l’idée que les GPCR représentent un mécanisme ancien et conservé de reconnaissance du stress cellulaire, en détectant par exemple les activités anormales de protéases, mécanisme probablement remodelé pour contribuer à la défense contre les infections [18]. 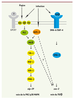 | Figure 7.
Schéma de l’activation des différentes voies produisant l’expression des peptides antimicrobiens des groupe nlp et cnc. |
L’action paracrine du TGF-β contrôle l’expression des gènes AMP du groupe cnc-2 dans l’épiderme La régulation du groupe des gènes cnc-2 apparaît tout autre. La sécrétion du ligand TGF-β par les neurones est en effet nécessaire à l’activation de la transcription des gènes du groupe cnc-2 dans l’épiderme en réponse à l’infection. Ce signal paracrine est lu par le récepteur hétérodimérique SMA-6/DAF-4 au niveau de l’épiderme et est transmis par la protéine SMAD SMA-3 (Figure 7) [ 38]. Une partie seulement de la voie de signalisation canonique du TGF-β étant utilisée, il sera intéressant de chercher à identifier les nouveaux partenaires permettant la transcription du groupe cnc-2 et d’analyser l’implication de ces nouveaux partenaires chez les mammifères. |
C. elegans possède des mécanismes de défense inductibles, régulés par une surprenante diversité de voies de signalisation. L’implication des GPCR dans la reconnaissance directe des pathogènes reste à démontrer. Les données récentes sur l’implication du système nerveux dans la réponse immunitaire du nématode vont permettre de mieux comprendre la communication entre les différents tissus et de voir comment l’immunité est intégrée dans la physiologie de l’organisme. |
1. Barrière A, Félix MA. Isolation of C. elegans and related nematodes. In : WormBook. The C. elegans Research Community ed. http://www.wormbook.org/
2. Hodgkin J, Kuwabara PE, Corneliussen B. A novel bacterial pathogen, Microbacterium nematophilum, induces morphological change in the nematode C. elegans. Curr Biol 2000; 10 : 1615–8. 3. Troemel ER, Felix MA, Whiteman NK, Barriere A, Ausubel FM. Microsporidia are natural intracellular parasites of the nematode Caenorhabditis elegans. PLoS Biol 2008; 6 : 2736–52. 4. Pujol N, Link EM, Liu, LX, et al. A reverse genetic analysis of components of the Toll signalling pathway in Caenorhabditis elegans. Curr Biol 2001; 11 : 809–21. 5. Pradel E, Zhang Y, Pujol N, et al. Detection and avoidance of a natural product from the pathogenic bacterium Serratia marcescens by Caenorhabditis elegans. Proc Natl Acad Sci USA 2007; 104 : 2295–300. 6. Kurz CL, Chauvet S, Andres E, et al. Virulence factors of the human opportunistic pathogen Serratia marcescens identified by in vivo screening. EMBO J 2003; 22 : 1451–60. 7. Tenor JL, Aballay A. A conserved Toll-like receptor is required for Caenorhabditis elegans innate immunity. EMBO Rep 9 2008; 9 : 103–9. 8. Fares H, Greenwald I. Genetic analysis of endocytosis in Caenorhabditis elegans: coelomocyte uptake defective mutants. Genetics 2001; 159 : 133–45. 9. Palm NW, Medzhitov R. Pattern recognition receptors and control of adaptive immunity. Immunol Rev 2009; 227 : 221–33. 10. Schulenburg H, Hoeppner MP, Weiner J, 3rd, Bornberg-Bauer E. Specificity of the innate immune system and diversity of C-type lectin domain (CTLD) proteins in the nematode Caenorhabditis elegans. Immunobiology 2008; 213 : 237–50. 11. Wong D, Bazopoulou D, Pujol N, Tavernarakis N, Ewbank JJ. Genome-wide investigation reveals pathogen-specific and shared signatures in the response of C. elegans to infection. Genome Biol 2007; 8 : R194. 12. O’Rourke D, Baban D, Demidova M, Mott R, Hodgkin J. Genomic clusters, putative pathogen recognition molecules, and antimicrobial genes are induced by infection of C. elegans with M. nematophilum. Genome Res 2006; 16 : 1005–16. 13. Pujol N, Zugasti O, Wong D, et al. Anti-fungal innate immunity in C. elegans is enhanced by evolutionary diversification of antimicrobial peptides. PLoS Pathog 2008; 4 : e1000105. 14. Means TK, Mylonakis E, Tampakakis E, et al. Evolutionarily conserved recognition and innate immunity to fungal pathogens by the scavenger receptors SCARF1 and CD36. J Exp Med 2009; 206 : 637–53. 15. Powell JR, Kim DH, Ausubel FM. The G protein-coupled receptor FSHR-1 is required for the Caenorhabditis elegans innate immune response. Proc Natl Acad Sci USA 2009; 106 : 2782–7. 16. Shivers RP, Youngman MJ, Kim DH. Transcriptional responses to pathogens in Caenorhabditis elegans. Curr Opin Microbiol 2008; 11 : 251–6. 17. Matzinger P. The danger model: a renewed sense of self. Science 2002; 296 : 301–5. 18. Chamy LE, Leclerc V, Caldelari I, Reichhart JM. Sensing of « danger signals » and pathogen-associated molecular patterns defines binary signaling pathways « upstream » of Toll. Nat Immunol 2008; 9 : 1165–70. 19. Shpacovitch V, Feld M, Bunnett NW, Steinhoff M. Protease-activated receptors: novel Partners in innate immunity. Trends Immunol 2007; 28 : 541–50. 20. Chavez V, Mohri-Shiomi A, Maadani A, Vega LA, Garsin DA. Oxidative stress enzymes are required for DAF-16-mediated immunity due to generation of reactive oxygen species by Caenorhabditis elegans. Genetics 2007; 176 : 1567–77. 21. Haskins KA, Russell JF, Gaddis N, Dressman HK, Aballay A. Unfolded protein response genes regulated by CED-1 are required for Caenorhabditis elegans innate immunity. Dev Cell 2008; 15 : 87–97. 22. Kim DH, Feinbaum R, Alloing G, et al. A conserved p38 MAP kinase pathway in Caenorhabditis elegans innate immunity. Science 2002; 297 : 623–6. 23. Troemel ER, Chu SW, Reinke V, et al. p38 MAPK Regulates Expression of Immune Response Genes and Contributes to Longevity in C. elegans. PLoS Genetics 2006; 2 : e183. 24. Nicholas HR, Hodgkin J. The ERK MAP kinase cascade mediates tail swelling and a protective response to rectal infection in C. elegans. Curr Biol 2004; 14 : 1256–61. 25. Kim DH, Liberati NT, Mizuno T, et al. Integration of Caenorhabditis elegans MAPK pathways mediating immunity and stress resistance by MEK-1 MAPK kinase and VHP-1 MAPK phosphatase. Proc Natl Acad Sci USA 2004; 101 : 10990–4. 26. Mallo GV, Kurz CL, Couillault C, et al. Inducible antibacterial defense system in C. elegans. Curr Biol 2002; 12 : 1209–14. 27. Garsin DA, Villanueva JM, Begun J, et al. Long-lived C. elegans daf-2 mutants are resistant to bacterial pathogens. Science 2003; 300 : 1921. 28. Aballay A, Ausubel FM. Programmed cell death mediated by ced-3 and ced-4 protects Caenorhabditis elegans from Salmonella typhimurium-mediated killing. Proc Natl Acad Sci USA 2001; 98 : 2735–9. 29. Shapira M, Hamlin BJ, Rong J, et al. A conserved role for a GATA transcription factor in regulating epithelial innate immune responses. Proc Natl Acad Sci USA 2006; 103 : 14086–91. 30. Singh V, Aballay A. Heat-shock transcription factor (HSF)-1 pathway required for Caenorhabditis elegans immunity. Proc Natl Acad Sci USA 2006; 103, 13092–7. 31. Irazoqui JE, Ng A, Xavier RJ, Ausubel FM. Role for beta-catenin and HOX transcription factors in Caenorhabditis elegans and mammalian host epithelial-pathogen interactions. Proc Natl Acad Sci USA 2008; 105 : 17469–74. 32. Kawli T, Tan MW. Neuroendocrine signals modulate the innate immunity of Caenorhabditis elegans through insulin signaling. Nat Immunol 2008; 9 : 1415–24. 33. Styer KL, Singh V, Macosko E, et al. Innate Immunity in Caenorhabditis elegans Is Regulated by Neurons Expressing NPR-1/GPCR. Science 2008; 322 (5 900) : 460–4. 34. Reddy KC, Andersen EC, Kruglyak L, Kim DH. A polymorphism in npr-1 is a behavioral determinant of pathogen susceptibility in C. elegans. Science 2009; 323 : 382–4. 35. Dijksterhuis J, Veenhuis M, Harder W. Ultrastructural study of adhesion and initial stages of infection of the nematode by conidia of Drechmeria coniospora. Mycological research 1990; 94 : 1–8. 36. Couillault C, Pujol N, Reboul J, et al. TLR-independent control of innate immunity in Caenorhabditis elegans by the TIR domain adaptor protein TIR-1, an ortholog of human SARM. Nat Immunol 2004; 5 : 488–94. 37. Ebert D. Host-parasite coevolution: Insights from the Daphnia-parasite model system. Curr Opin Microbiol 2008; 11 : 290–301. 38. Zugasti O, Ewbank JJ. Neuroimmune regulation of antimicrobial peptide expression by a noncanonical TGF-beta signaling pathway in Caenorhabditis elegans epidermis. Nat Immunol 2009; 10 : 249–56. 39. Pujol N, Cypowyj S, Ziegler K, et al. Distinct innate immune responses to infection and wounding in the C. elegans epidermis. Curr Biol 2008; 18 : 481–9. 40. Tong A, Lynn G, Ngo V, et al. Negative regulation of Caenorhabditis elegans epidermal damage responses by death-associated protein kinase. Proc Natl Acad Sci USA 2009; 106 : 1457–61. 41. Sorensen OE, Thapa DR, Roupe KM, et al. Injury-induced innate immune response in human skin mediated by transactivation of the epidermal growth factor receptor. J Clin Invest 2006; 116 : 1878–85. 42. O’Neill LA, Fitzgerald KA, Bowie AG. The Toll-IL-1 receptor adaptor family grows to five members. Trends Immunol 2003; 24 : 286–290. 43. Liberati NT, Fitzgerald KA, Kim DH, et al. Requirement for a conserved Toll/interleukin-1 resistance domain protein in the Caenorhabditis elegans immune response. Proc Natl Acad Sci USA 2004; 101 : 6593–8. 44. Kiss-Toth E, Bagstaff SM, Sung HY, et al. Human tribbles, a protein family controlling mitogen-activated protein kinase cascades. J Biol Chem; 279 : 42703–8. 45. Ziegler K, Kurz CL, Cypowyj S, et al. Antifungal innate immunity in C. elegans: PKδd links G-protein signaling and a conserved p38 MAPK cascade. Cell Host and Microbes 2009 (sous presse). 46. Sendid B, Jouault T, Vitse A, et al. Glycannes pariétaux de levures et anticorps spécifiques : biomarqueurs et outils d’analyse physiopathologique des candidoses et de la maladie de Crohn. Med Sci (Paris) 2009; 25 : 473–81. |