IMGT®, the international ImMunoGeneTics information system ® 1 [ 1] fête cette année son vingtième anniversaire. Créé en 1989 à Montpellier par le Laboratoire d’ImmunoGénétique Moléculaire (LIGM) des professeurs Marie-Paule et Gérard Lefranc (Université Montpellier 2 et CNRS), IMGT® est le premier et actuellement le seul système d’information intégré en immunogénétique et immunoinformatique. IMGT®, marque déposée du CNRS (France, Union européenne, Canada et États-Unis), est spécialisé dans les séquences, structures et données génétiques des immunoglobulines (IG) ou anticorps, des récepteurs des lymphocytes T (TR), du complexe majeur d’histocompatibilité (MHC, major histocompatibility complex en anglais), des protéines des superfamilles IgSF (immunoglobulin superfamily) et MhcSF (Mhc super-family), et des protéines apparentées du système immunitaire. IMGT® est composé de plusieurs bases de données (quatre de séquences, une de gènes, une de structures 3D) et d’une quinzaine d’outils interactifs (Figure 1) et de plus de 10 000 pages de ressources Web. En raison de la complexité de leur synthèse et de leur incroyable diversité, les molécules du système immunitaire, et en particulier les anticorps, doivent être traitées d’une façon spécifique. L’analyse des données et la standardisation de leur annotation ont été possibles grâce à la création d’IMGT-ONTOLOGY, la première ontologie dans le domaine [ 2, 3]. IMGT® est aujourd’hui le système de référence international en immunogénétique et immunoinformatique, utilisé en recherche fondamentale, médicale et biotechnologique.
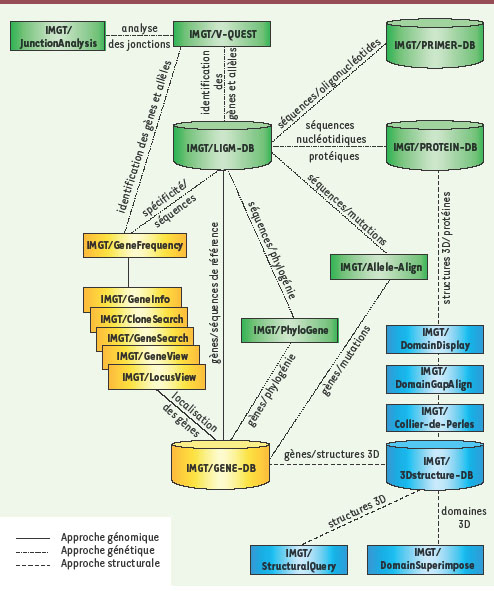
| Figure 1.
IMGT®, the international ImMunoGeneTics information system®. IMGT® (http://www.imgt.org) comprend des bases de données et des outils d’analyse (respectivement représentés sous forme de cylindres et de rectangles) et plus de 10 000 pages de ressources Web [1]. Les séquences et les gènes, ainsi que les structures tridimensionnelles, sont analysés et gérés selon les trois approches biologiques : génétique, génomique et structurale. |
Un organisme humain synthétise plus de 1012 anticorps et plus de 1012 récepteurs T différents pour faire face aux agressions extérieures de la part des virus, bactéries et parasites et pour lutter contre ses propres cellules malignes. C’est par des mécanismes complexes de réarrangements de l’ADN et, dans le cas des gènes IG, de mutations somatiques, qu’un nombre aussi important de récepteurs d’antigènes peut être obtenu (→). Créé en 1989 pour gérer cette complexité des gènes, séquences et structures des IG et TR, IMGT® est aujourd’hui une source unique de connaissances en immunogénétique au niveau international. Il n’y a pas d’équivalent en Europe, aux États-Unis, au Japon et nulle part ailleurs dans le monde. Le serveur Web d’IMGT® à Montpellier reçoit des accès de plus de 80 000 sites différents par an, avec une moyenne de plus de 150 000 requêtes par mois. Les utilisateurs sont répartis à parts égales entre l’Europe, les États-Unis et le reste du monde. IMGT® est reconnu pour la richesse et la qualité de ses données scientifiques et son interface conviviale.
(→) voir M. Fougereau, page 997
IMGT® est utilisé par des chercheurs d’équipes académiques et industrielles dans de multiples domaines de recherche : recherche fondamentale, recherche médicale (analyse des répertoires des anticorps et des sites de reconnaissance des récepteurs T dans les réponses immunitaires normales contre les agents infectieux et les cellules malignes, et lors des réponses anormales au cours de maladies auto-immunes et de syndromes lymphoprolifératifs, leucémies, lymphomes, myélomes), recherche vétérinaire (répertoire des IG et TR dans les espèces domestiques et sauvages), recherche génomique (étude de la diversité et de l’évolution des gènes de la réponse immunitaire adaptative), recherche en biologie structurale (évolution des domaines des protéines des superfamilles IgSF et MhcSF) et dans les biotechnologies relatives aux projets de l’Human proteome organisation (HUPO) et à l’ingénierie des anticorps (single chain Fragment variable scFv, banques combinatoires, phage display, anticorps chimériques, humanisés et humains) (→), recherche pour le diagnostic, le pronostic et le suivi thérapeutique des leucémies, lymphomes et myélomes (identification du ou des clone(s) malin(s) et évaluation de la maladie résiduelle) et dans les approches thérapeutiques (greffes, immunothérapie, vaccinologie). IMGT® est accessible à l’adresse suivante : http://www.imgt.org, gratuitement pour la recherche académique, mais par contrats et licences pour les sociétés comme Centocor Inc., Johnson and Johnson (États-Unis), Merck & Co Inc. (États-Unis), Amgen Inc. (États-Unis), Sanofi-Aventis GmbH (Allemagne), Chugai Pharmaceutical Co., Ltd (Japon), etc.
(→) voir A. Beck et al., page 1024 ; R. Abès et al., page 1011 ; M. Cogné et al., page 1149