| |
| Med Sci (Paris). 2010 January; 26(1): 9–11. Published online 2010 January 15. doi: 10.1051/medsci/20102619.La drosophile comme nouveau modèle d’étude des leucémies Dani Osman, Vanessa Gobert, Marc Haenlin, and Lucas Waltzer* Université de Toulouse, UPS, Centre de biologie du développement (CBD), Bâtiment 4R3, 118, route de Narbonne, 31062 Toulouse, France CNRS, CBD UMR5547, 31062 Toulouse, France MeSH keywords: Animaux, Animal génétiquement modifié, Calpain, physiologie, Cellules cultivées, métabolisme, Sous-unité alpha 2 du facteur CBF, Protéines de liaison à l'ADN, Modèles animaux de maladie humaine, Protéines de Drosophila, Drosophila melanogaster, cytologie, Régulation de l'expression des gènes dans la leucémie |
Le développement de leucémies et de lymphomes est souvent associé à des mutations ponctuelles ou à des réarrangements chromosomiques non aléatoires qui affectent des régulateurs clés du développement hématopoïétique. Ainsi, la translocation t(8 ; 21) (q22 ; q22), associée à environ 15 % des leucémies myéloïdes aiguës, affecte le gène RUNX1/AML1 (pour acute myeloid leukaemia 1) qui est essentiel à l’émergence des cellules souches hématopoïétiques et participe à leur différenciation aussi bien dans le lignage myéloïde que dans le lignage lymphoïde [
1,
11]. |
Protéine de fusion AML1-ETO et leucémie aiguë myéloïde
AML1 code un facteur de transcription de la famille RUNX qui se caractérise par la présence d’un domaine de liaison à l’ADN de type runt phylogénétiquement très conservé. La translocation t(8 ; 21) induit l’expression, sous le contrôle des séquences régulatrices d’AML1, de la protéine de fusion AML1-ETO (the eight-twenty-one corepressor) qui contient la partie amino-terminale d’AML1, dont son domaine de liaison à l’ADN, fusionnée à la presque totalité de ETO (Figure 1). ETO est une protéine nucléaire capable de s’oligomériser et de recruter différents corépresseurs de la transcription tels que NcoR, Sin3A et des histones déacétylases. Aussi a-t-il été proposé qu’AML1-ETO agit principalement en réprimant constitutivement l’expression des gènes cibles d’AML1. Toutefois, son mode d’action est probablement plus complexe [
2]. En effet, AML1-ETO peut d’une part interagir avec et modifier l’activité d’autres facteurs de transcription impliqués dans la différenciation myéloïde, et d’autre part agir comme activateur de la transcription sur certains gènes.
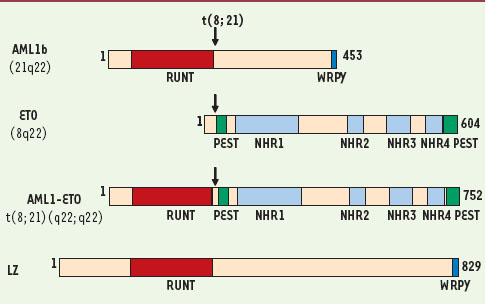
 | Figure 1.
Représentation schématique d’AML1, ETO, AML1-ETO et LZ. AML1-ETO, qui conserve le domaine RUNT de liaison à l’ADN d’AML1, interfère avec la régulation transcriptionnelle des cibles d’AML1 et agit au moins en partie comme dominant négatif. Le domaine RUNT de LZ présente 71 % d’identité avec celui d’AML1. Les différents domaines structuraux sont indiqués. WRPY : domaine d’interaction avec les corépresseurs de la famille GRO/TLE. NHR 1-4 : nervy homology regions 1-4. PEST : domaine PEST. |
Différents systèmes de culture cellulaire ou in vivo chez la souris ont été développés pour étudier le mode d’action d’AML1-ETO dans les leucémies [
3]. Ces modèles ont permis de montrer qu’AML1-ETO inhibe la différenciation myéloïde et promeut la conservation d’un nombre élevé de progéniteurs hématopoïétiques. Cependant, si la translocation t(8 ; 21) semble être un élément initiateur, des mutations secondaires (conduisant par exemple à une activation constitutive de Ras ou de Flt3) sont requises pour que les cellules exprimant AML1-ETO deviennent leucémiques. En parallèle, des analyses transcriptomiques et plus récemment par ChIP-chip ont permis d’identifier des gènes dérégulés par AML1-ETO et de mieux comprendre les voies de signalisation en aval de cet oncogène. Les facteurs qui modulent l’activité d’AML1-ETO restent en revanche pour la plupart inconnus. |
Modéliser le rôle d’AML1-ETO chez la drosophile L’identification des gènes qui interagissent fonctionnellement avec AML1-ETO est essentielle, mais sa réalisation demeure ardue dans les systèmes mammifères. Afin d’établir un modèle d’étude d’AML1-ETO qui se prête à une analyse génétique de son activité, nous avons caractérisé l’effet de l’expression d’AML1-ETO dans le système hématopoïétique de la drosophile. En effet, malgré la distance phylogénétique qui sépare ces deux espèces, différents aspects du développement des cellules hématopoïétiques ont été conservés de la drosophile à l’homme [
4]. En particulier, la drosophile possède deux classes principales de cellules sanguines (ou hémocytes), les plasmatocytes et les cellules à cristaux, qui ressemblent structurellement et fonctionnellement aux cellules myéloïdes des vertébrés. Le développement des cellules à cristaux dépend en particulier du facteur de transcription de type RUNX Lozenge (LZ) [
5] (Figure 1), qui est exprimé dans une sous-population de progéniteurs hématopoïétiques dont il va promouvoir la différenciation [
6,
7] (Figure 2). De façon intéressante, nous avons observé que l’expression d’AML1-ETO dans les cellules sanguines de la drosophile induit des phénotypes de type « préleucémique » similaires à ceux que l’on observe dans des modèles mammifères [
8] : AML1-ETO inhibe la différenciation des cellules à cristaux et induit une augmentation du nombre d’hémocytes exprimant LZ qui perdurent sous la forme de progéniteurs (Figure 2).
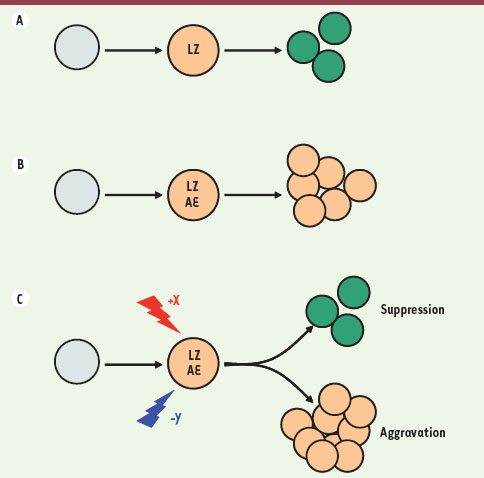
 | Figure 2.
Phénotypes induits par l’expression d’AML1-ETO (AE) dans les cellules sanguines de la drosophile exprimant LZ. A. Le facteur RUNX LZ est exprimé dans les progéniteurs (orange) des cellules à cristaux dont il induit la différenciation (vert). B. L’expression d’AML1-ETO dans les cellules LZ+ empêche leur différenciation et promeut leur expansion. C. Dans des conditions de crible génétique (induction de l’expression ectopique d’un gène X ou de la perte de fonction d’un gène Y dans les cellules LZ+), les phénotypes induits par AML1-ETO peuvent être soit supprimés soit aggravés. |
|
Identification de la calpaïne comme un substrat de AML1-ETO D’autre part, il est apparu que l’expression d’AML1-ETO sous le contrôle des régions régulatrices de LZ provoque la mort des mouches au stade pupe1,. Ce phénotype nous a servi de base pour la réalisation d’un crible génétique visant à identifier des gènes requis pour l’activité d’AML1-ETO (Figure 2). Ainsi, sur environ 1 500 gènes ciblés par interférence de l’ARN, une dizaine de suppresseurs de la létalité induits par AML1-ETO a été identifiée parmi lesquels une calpaïne [8]. Les calpaïnes forment une famille conservée de protéases dépendantes du Ca2+ qui régulent des aspects fondamentaux de la physiologie cellulaire tels que la migration ou l’apoptose, mais leur rôle dans le développement de leucémies n’avait jamais été exploré [
9,
12]. Il s’avère que la perte de fonction du gène calpainB supprime non seulement la létalité mais aussi les phénotypes préleucémiques induits par l’expression d’AML1-ETO chez la drosophile. En outre, nous avons pu montrer que l’inhibition de l’activité des calpaines chez l’homme réduit fortement la croissance et la clonogénicité de cellules leucémiques exprimant AML1-ETO. Ainsi, ces résultats suggèrent que les calpaïnes sont requises pour l’activité oncogénique d’AML1-ETO de la drosophile à l’homme. Cette étude établit le premier modèle « drosophile » d’étude de la leucémogenèse associée à la translocation t(8 ; 21) et ouvre de nombreuses perspectives. Concernant les calpaïnes, le mécanisme moléculaire par lequel ces enzymes participent à l’activité d’AML1-ETO reste à établir. Les calpaïnes clivent généralement leur substrat de manière limitée et génèrent fréquemment des fragments stables possédant des activités différentes de celles du substrat intact. Chez la drosophile comme chez l’homme, l’inhibition des calpaïnes induit une déstabilisation d’AML1-ETO au niveau post-transcriptionnel [8]. Il est possible que les calpaïnes agissent via les caspases et/ou le protéasome qui sont connus pour réguler la stabilité d’AML1-ETO [
10]. Une autre possibilité est que AML1-ETO, qui possède deux domaines de type PEST (proline, glutamate, sérine, et thréonine) (Figure 1), également présents dans de nombreux substrats des calpaïnes, soit une cible directe de ces enzymes. D’un point de vue médical, différents inhibiteurs pharmacologiques de ces enzymes sont disponibles et suscitent beaucoup d’intérêt pour le traitement de maladies neurodégénératives de type Parkinson ou Alzheimer. Le potentiel thérapeutique de ces inhibiteurs dans le cas de leucémies aiguës pourrait donc être testé relativement rapidement. |
La drosophile, quelle place comme modèle de tumorigenèse humaine ? Plus généralement, ces résultats ouvrent la voie à l’utilisation de la drosophile comme tube à essai pour identifier d’autres gènes dont les homologues humains régulent l’activité d’AML1-ETO dans les leucémies. Les puissants outils disponibles chez la drosophile permettent de réaliser différents types de cribles génétiques (Figure 2), que ce soit en perte de fonction ou en gain de fonction. Comme l’expression d’AML1-ETO n’est pas suffisante per se pour induire des leucémies chez les mammifères [3], il serait intéressant de rechercher des enhancers d’AML1-ETO chez la drosophile dans le but d’identifier les voies de signalisation qui coopèrent avec cet oncogène. Ainsi, on peut espérer que la drosophile permette de mettre à jour les réseaux génétiques conservés qui constituent la base de la transformation cellulaire et que ce modèle aidera à mieux comprendre le mode d’action d’AML1-ETO mais aussi d’autres oncogènes. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Niebuhr B, Fischer M, Tager M, et al. Gatekeeper function of the RUNX1 transcription factor in acute leukemia. Blood Cells Mol Dis 2008; 40 : 211–8. 2. Elagib KE, Goldfarb A. Oncogenic pathways of AML1-ETO in acute myeloid leukemia: multifaceted manipulation of marrow maturation. Cancer Lett 2007; 251 : 179–86. 3. McCormack E, Bruserud O, Gjertsen BT. Review: genetic models of acute myeloid leukaemia. Oncogene 2008; 27 : 3765–79. 4. Hartenstein V. Blood cells and blood cell development in the animal kingdom. Annu Rev Cell Dev Biol 2006; 22 : 677–712. 5. Lebestky T, Chang T, Hartenstein V, Banerjee U. Specification of Drosophila hematopoietic lineage by conserved transcription factors. Science 2000; 288 : 146–9. 6. Waltzer L, Ferjoux G, Bataillé L, Haenlin M. Cooperation between the GATA and RUNX factors Serpent and Lozenge during Drosophila hematopoiesis. Embo J 2003; 22 : 6516–25. 7. Ferjoux G, Augé B, Boyer K, et al. A GATA/RUNX cis-regulatory module couples Drosophila blood cell commitment and differentiation into crystal cells. Dev Biol 2007; 305 : 726–34. 8. Osman D, Gobert V, Ponthan F, et al. A Drosophila model identifies calpains as modulators of the human leukemogenic fusion protein AML1-ETO. Proc Natl Acad Sci USA 2009; 106 : 12043–8. 9. Croall DE, Ersfeld K. The calpains: modular designs and functional diversity. Genome Biol 2007; 8 : 218. 10. Corsello SM, Roti C, Ross KN, et al. Identification of AML1-ETO modulators by chemical genomics. Blood 2009; 113 : 6193–205. 11. Godin I, Cumano A. Hematopoietic stem cells: where do they come from at last ? Med Sci (Paris) 2007; 23 : 681–4. 12. Baud L, Fouqueray B, Bellocq A, Peltier J. Calpains participate in inflammatory reaction development. Med Sci (Paris) 2003; 19 : 71–6. |