| |
| Med Sci (Paris). 2010 March; 26(3): 236–238. Published online 2010 March 15. doi: 10.1051/medsci/2010263236.Variants génétiques associés au diabète de type 1 et contrôle de l’expression du récepteur de l’IL-2 Vincent Plagnol* Juvenile Diabetes Research Foundation/Wellcome, Trust Diabetes and Inflammation Laboratory, Cambridge Institute for Medical Research, University of Cambridge, Addenbrooke’s hospital, Hills Road, CB2 OXY Cambridge, Royaume-Uni MeSH keywords: Diabète, épidémiologie, génétique, immunologie, Régulation de l'expression des gènes, Variation génétique, Humains, Sous-unité alpha du récepteur à l'interleukine-2, Lymphocytes, physiologie, Polymorphisme de nucléotide simple, Facteurs de risque, Pyridines, usage thérapeutique, Transduction du signal |
Un enjeu essentiel de la génétique consiste à faire le lien entre l’ADN d’un individu et ses caractéristiques, par exemple la probabilité qu’il a de développer une maladie mais aussi des caractéristiques morphologiques comme sa taille ou son poids. Pour atteindre cet objectif, le génotypage de milliers, voire de millions de marqueurs génétiques a permis l’avènement des études cas-témoins, que la récente diminution drastique des coûts rend plus accessible [
1]. Cette approche consiste à comparer les génomes de milliers d’individus atteints par une maladie (cas) avec des individus contrôles (témoins) afin d’identifier leurs différences. Cette révolution est la source d’innombrables publications dans de prestigieuses revues comme Nature et surtout Nature Genetics, qui dédie une large fraction de sa table des matières à ce sujet. Cependant, dans la plupart des cas, ces variants ont des effets trop faibles pour pouvoir estimer avec précision la probabilité de développer les maladies qui leur sont associées. Cela explique en grande partie que la communauté scientifique reste divisée sur l’impact médical de ces découvertes. Pour beaucoup de généticiens, la question clé est maintenant de savoir comment profiter de ces découvertes fondamentales pour faire avancer la recherche médicale appliquée [
5]. |
L’hypothèse de travail : rechercher un lien entre variant génétique et risque de la maladie chez les sujets sains Notre travail collaboratif, récemment publié dans Nature Genetics [
2] et mené par Calliope Dendrou et Linda Wicker, prend comme point de départ la liste de 42 variants génétiques (pour l’essentiel des polymorphismes nucléotidiques simples ou SNP [
3]) fréquents dans la population (détectés chez au moins 10 % des individus) et qui ont été associés au risque de développer un diabète de type 1. Le diabète de type 1 est une maladie auto-immune incurable qui affecte un individu sur 200 au Royaume-Uni et qui rend les patients dépendants d’injections journalières d’insuline pour leur survie. Les mécanismes qui sont à l’origine de cette réaction auto-immunitaire restent très mal compris : les cellules T CD8+ sont responsables de la destruction des cellules pancréatiques bêta, mais il ne fait aucun doute que de multiples compartiments du système immunitaire sont impliqués. S’il était possible de démontrer que ces allèles associés au risque de diabète affectent une population spécifique de cellules immunitaires, on pourrait alors impliquer avec un degré important de certitude cette population dans le développement de la maladie, une information qui serait importante pour mieux cibler les futurs traitements. La stratégie que nous avons utilisée pour identifier les effets de ces SNP consiste à corréler la présence de ces allèles avec des paramètres quantifiables, par exemple la mesure de protéines au niveau cellulaire, dans des échantillons suffisamment larges d’individus. Intuitivement, on pourrait penser que de telles corrélations doivent être observées chez des individus diabétiques. Cependant, nous avons un point de vue un peu différent : nous pensons qu’à un stade avancé de la maladie, les mécanismes immunitaires sont probablement différents de ceux qui interviennent aux stades initiaux. Pour cette raison, plutôt que de travailler sur des échantillons prélevés chez des individus diabétiques, nous avons préféré nous intéresser à des individus sains, et explorer en quoi cette variabilité génétique pouvait modifier leur système immunitaire. |
Le choix du paramètre quantifiable (le récepteur alpha de l’IL-2) et de la population test La question est de savoir comment attaquer ce problème : que mesurer et comment le mesurer ? Parmi les SNP dont un des deux allèles augmente le risque de développer le diabète de type 1, plusieurs affectent la production d’interleukine 2 (IL-2), un messager essentiel du système immunitaire, ou la réponse à cette cytokine. Pour cette raison, notre laboratoire s’intéresse au gène codant pour le récepteur alpha de l’IL-2 (IL-2RA), une composante importante de la réponse à l’IL-2. Cet intérêt est renforcé par le fait que parmi les 42 SNP publiés qui influencent le risque de développer un diabète de type 1, trois se situent à proximité de ce gène IL2RA dans la séquence de notre ADN [
4]. La protéine IL-2RA est un récepteur localisé à la surface de plusieurs populations de cellules immunitaires. Notre hypothèse est que la quantité de récepteurs est partiellement contrôlée par les allèles correspondant aux trois SNP associés avec la maladie. Nous avions donc besoin d’une technique permettant de quantifier cette protéine IL-2RA. La technique qui s’est imposée est la cytométrie en flux qui permet la quantification de multiples protéines de surface, séparément dans chacune des cellules d’un échantillon sanguin. Pour nous donner une chance de détecter des effets potentiellement subtils, il était nécessaire de considérer des échantillons relativement grands. En conséquence, nous avons inclus 200 individus dans notre étude, un nombre compatible à la fois avec la précision demandée et le coût élevé de ces mesures. Ces efforts sont compliqués par la rareté dans la population de certains allèles parmi ces trois SNP. Par exemple, pour un des trois SNP qui nous intéressent, la plupart des individus sont porteurs de l’allèle à risque et 1 % seulement sont porteurs des deux copies de l’allèle qui confère une protection contre le diabète. Pour s’assurer d’une bonne représentation de tous les allèles parmi les 200 individus inclus dans l’étude, nous avons d’abord recruté, en collaboration avec plusieurs laboratoires de l’université de Cambridge, plus de 5 000 individus prêts à participer à une ou plusieurs études à but médical. L’ADN de ces individus a été extrait d’échantillons de salive et analysé. Nous avons ainsi identifié les individus porteurs des allèles rares qui nous intéressaient et nous leur avons proposé un prélèvement sanguin. Sans eux nos découvertes n’auraient pas été possibles et nous leur sommes extrêmement reconnaissants. |
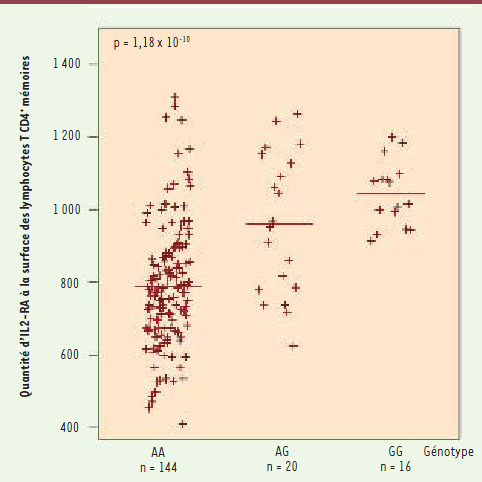
Un lien entre allèle protecteur et quantité d’IL-2RA L’analyse des données que nous avons obtenues confirme notre hypothèse. Nous avons démontré qu’en effet, dans la sous-population de lymphocytes T CD4+ mémoires, le nombre de récepteurs IL-2RA augmente avec la présence d’allèles protecteurs à un des trois SNP sélectionnés pour cette étude (en moyenne 30 % de récepteurs IL-2RA supplémentaires par allèle protecteur, voir Figure 1). Des effets similaires mais plus subtils ont aussi corrélé la présence d’un second SNP avec la quantité de récepteur IL-2RA dans deux autres populations de cellules : les lymphocytes T CD4+ naïfs et les monocytes. Cependant, les corrélations observées dans ces deux types de cellules vont dans le sens inverse du SNP précédent : les allèles protecteurs y sont associés à un nombre plus réduit de récepteurs IL-2RA. Quant au rôle du troisième SNP, nous ne l’avons pas encore déterminé.
 | Figure 1.
Lien entre quantité d’IL-2RA et présence des allèles porteurs du SNP. La quantité de récepteurs IL-2RA sur la surface des cellules T CD4+ mémoires est contrôlée par le génotype du SNP rs12722495, dont l’allèle G est associé avec une protection contre le diabète. Génotype : rs12722495 ; AA : risque ; GG : protection. |
|
L’interprétation de ces résultats n’est pas évidente car il n’est pas certain que cette corrélation soit effectivement le facteur de risque pour la maladie. Cette explication est cependant probable et si elle est correcte, nous avons appris que ces allèles interviennent probablement en altérant la quantité de récepteurs IL-2RA. Mais surtout nous savons que ces trois populations (cellules T CD4+ mémoire, T CD4+ naïves et monocytes) jouent un rôle dans le développement du diabète. C’est donc une piste que nous essayons de suivre, en espérant que ce fil d’Ariane va nous mener aux mécanismes initiaux de la maladie qui, potentiellement, pourront faire l’objet de traitements. |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1. Wellcome Trust case control consortium. Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls. Nature 2007; 447 : 661–78. 2. Dendrou C, Plagnol V, Fung E, et al. Cell-specific protein phenotypes for the autoimmune locus IL2RA using a genotype-selectable human bioresource. Nat Genet 2009; 41 : 1011–5. 3. Barrett JC, Clayton DG, Concannon P, et al. Genome-wide association study and meta-analysis find that over 40 loci affect risk of type 1 diabetes. Nat Genet 2009; 41 : 703–7. 4. Lowe CE, Cooper JD, Brusko T, et al. Large-scale genetic fine mapping and genotype-phenotype associations implicate polymorphism in the IL2RA region in type 1 diabetes. Nat Genet 2007; 39 : 1074–82. 5. Bouatia-Naji N, Bonnefond A, Froguel P. Avancées dans la génétique de la glycémie à jeun : quelles leçons pour le diabète ?. Med Sci (Paris) 2009; 25 : 897–902. |