| |
| Med Sci (Paris). 2010 March; 26(3): 325–327. Published online 2010 March 15. doi: 10.1051/medsci/2010263325.Chroniques génomiques Le boom des séquenceurs nouvelle génération Bertrand Jordan* Marseille-Nice Génopole, case 901, Parc scientifique de Luminy, 13288 Marseille Cedex 9, France MeSH keywords: Séquence nucléotidique, ADN, génétique, Génomique, tendances, Humains, Tumeurs du poumon, ARN, Études de cohortes, Adulte d'âge moyen, Enquêtes et questionnaires |
Des achats massifs, des gammes qui s’élargissent Au début de cette année sont tombées coup sur coup deux nouvelles étonnantes : le 12 janvier, BGI (Beijing Genomics Institute) annonçait l’acquisition de 128 (cent vingt huit…) séquenceurs Illumina de dernière génération (le modèle HiSeq 2000, donné pour 200 gigabases (Gb) par session) - et le 28 du même mois, un nouvel institut Nord-américain, Ignite Institute (structure sans but lucratif implantée en Virginie du Nord) rendait public l’achat de 100 (cent…) machines SOLiD dernier modèle (SOLiD 4, production annoncée de 100 à 300 Gb). Ces chiffres sont surprenants : même en tenant compte qu’il s’agit là de séquence « brute », et qu’il faut à peu près trente Gb de telles données pour une Gb de séquence finie, un run de chacune de ces machines lit plusieurs génomes humains : a-t-on réellement besoin d’une telle masse de données ? Parallèlement, les trois principaux fabricants élargissent leur offre. Le plus ancien, Roche/454, a annoncé début décembre une machine d’entrée de gamme, le GS junior, environ cinq fois moins chère que l’appareil GS FLX et produisant dix fois moins de séquence. Cette proposition est clairement dirigée vers les équipes de taille moyenne, qui disposeront ainsi, pour environ 100 000 dollars, d’une capacité importante adaptée au séquençage de novo grâce aux lectures longues (500 bases environ) qui sont l’avantage principal de ce système. Illumina, l’entreprise dominante sur ce marché, a lancé coup sur coup deux modèles, le Genome Analyser IIe, à moitié prix (et moitié performance) de son cheval de bataille le GA IIx, et le HiSeq 2000 (celui qu’achète le BGI à 128 exemplaires) qui double ou triple le nombre de gigabases par session. Enfin Life Tech (ex-Applied Biosystems) élargit sa gamme vers le haut avec le SOLiD 4 mentionné ci-dessus. |
On voit donc qu’il y a un véritable boom (ou une bulle destinée à éclater dans un avenir proche ?) sur ce marché du séquençage nouvelle génération - sans oublier la génération suivante qui se profile déjà [
1] et dont le premier représentant, Helicos, commence à réellement vendre des machines… Ce boom concerne les grands centres, mais aussi les laboratoires de taille moyenne qui, de plus en plus, s’équipent de ces machines onéreuses : environ 500 000 dollars pièce, et 5 000 à 10 000 dollars de réactifs par session. En fait, on estime (les chiffres exacts restent un secret bien gardé) qu’il s’est déjà vendu entre 1 000 et 2 000 machines, Illumina se taillant la part du lion, plus de la moitié ; le plan de relance d’Obama a joué un rôle important puisqu’il a permis aux National Institutes of Health d’allouer plus de cent quarante millions de dollars supplémentaires à des projets de séquençage. Il est légitime de se demander si cela correspond à un réel besoin, et si toutes ces machines vont vraiment être employées à la hauteur de leurs possibilités. Nous avons connu au cours des dernières décennies quelques exemples de suréquipement sans utilité scientifique très évidente : dans les années 1980, c’était la vogue des microscopes électroniques (chaque institut devait en avoir un, c’était une question de standing), puis, au début des années 1990, celle des premiers séquenceurs Applied dont de nombreux exemplaires fièrement exposés sont longtemps restés inactifs, ou encore la mode des appareils de spectrométrie de masse… Les séquenceurs de nouvelle génération exigent un environnement informatique très performant afin de gérer correctement les masses énormes de données qu’ils produisent (elles se comptabilisent en téraoctets) [
2], et leur coût de fonctionnement en réactifs est estimé à 200 000 dollars par an : autant d’éléments qui peuvent restreindre l’usage réel d’installations qui font la gloire de leurs propriétaires ! Quant à leur production scientifique réelle, au rapport qualité/prix en somme, il est encore un peu tôt pour l’évaluer. Le système le plus ancien, 454, revendique un peu plus de six cents publications scientifiques, et le chiffre d’Illumina est à peu près équivalent. SOLiD, plus récent, n’en annonce qu’une cinquantaine. Ce sont là des chiffres notables, mais il faut les mettre en regard avec le fait que plusieurs centaines de ces machines sont en fonction depuis déjà un an ou deux ; par ailleurs ces articles ne sont pas tous parus dans Science ou Nature…
|
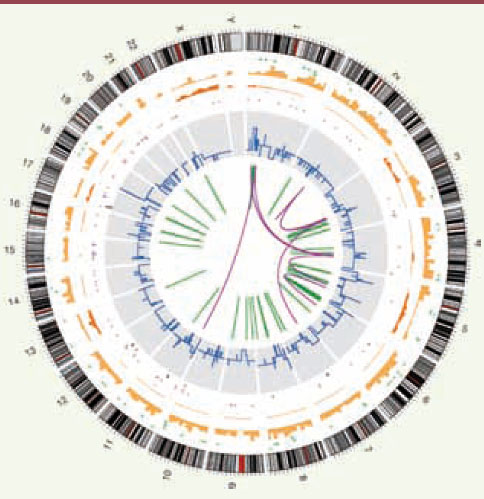
Il me semble pourtant que ce changement dans le rythme et l’accessibilité du séquençage constitue une évolution importante pour la recherche biologique, un « changement de paradigme » pour parler comme les magazines. Par rapport à toutes les autres approches, du séquençage ciblé vers quelques gènes aux profils d’expression en passant par l’analyse cytogénétique, le séquençage intégral apporte une vision beaucoup plus complète et détaillée de l’ensemble des altérations génomiques d’une cellule cancéreuse, pour se limiter à cet exemple (il en existe bien d’autres). La Figure 1 montre les anomalies repérées par le séquençage complet d’une cellule cancéreuse comparée à la cellule normale [
3] : il est évident que l’on serait passé à côté de la plupart d’entre elles si l’on s’était limité à l’une des techniques précédentes, ou même à la combinaison de plusieurs d’entre elles… Bien d’autres applications nouvelles deviennent possibles : l’étude quantitative et précise des transcriptomes (RNA-seq), l’investigation « en masse » de communautés microbiennes par séquençage sans séparation ni culture préalables, l’identification des motifs reconnus par diverses protéines (ChIp-seq), les études précises et exhaustives des profils de méthylation… [
4]. Encore faut-il avoir un bon projet, un environnement adéquat et une équipe aguerrie.
 | Figure 1.
Représentation de l’ensemble des anomalies détectées par le séquençage d’une lignée cellulaire dérivée d’un cancer du poumon à petites cellules (lignée NCIH209). De la circonférence vers le centre du cercle : schéma des chromosomes ; insertions (en vert clair), délétions (vert foncé) ; substitutions homozygotes (orange foncé) et hétérozygotes (orange clair) en fréquence par 10 mégabases ; substitutions dans les régions codantes (différentes couleurs selon qu’elles sont silencieuses, mis-sens, non-sens…) ; changements du nombre de copies (bleu) ; réarrangements intrachromosomiques (lignes vertes) et interchromosomiques (lignes pourpres) (tiré de [ 3]). |
Il est en effet clair qu’ilne suffit plus - si tant est que cela ait jamais été le cas - d’acquérir un séquenceur ultra-rapide pour publier dans une revue de haut niveau… Cela d’autant plus que les champs d’application les plus évidents, le low-hanging fruit comme disent les Anglo-saxons, ont déjà été exploités, et qu’il faut maintenant des idées astucieuses, des ressources biologiques privilégiées et des collaborations solides pour bâtir des projets tirant réellement parti de ces nouvelles possibilités. Il faut aussi se demander si les données massives qui s’accumulent dans les bases de données publiques comme celle du 1 000 genomes project
1 ne peuvent pas répondre à certaines questions, ou au contraire, pour des projets de plus faible dimension, s’il n’est pas plus pertinent de faire appel au séquençage à façon (de très nombreuses entreprises proposent maintenant ce séquençage en mode service) plutôt que se lancer dans la délicate mise en place d’une infrastructure locale. Bref, il vaut mieux réfléchir avant de se lancer… Il n’en reste pas moins que le séquençage ultra-rapide fait maintenant partie intégrante de la recherche biologique et que son emploi - selon diverses modalités - devient un ingrédient indispensable de très nombreuses recherches. |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Jordan B. La génération suivante, déjà… Med Sci (Paris) 2009; 25 : 649–50. 2. Prepare for the deluge.Nat Biotechnol 2008; 26 : 1099. 3. Pleasance ED, Stephens PJ, O’Meara S et al. A small-cell lung cancer genome with complex signatures of tobacco exposure. Nature 2010; 463 : 184–92. 4. Metzker ML. Sequencing technologies - the next generation. Nat Rev Genet 2010; 11 : 31–46. |