Depuis des décennies, les regards croisés de la phylogenèse et de la cytogénétique portés sur le chromosome Y n’avaient révélé qu’une longue histoire de régression. À partir d’un proto-Y, aussi riche en gènes que le proto-X, il y a 200 millions d’années, le chromosome Y s’est progressivement amenuisé, rendant impossible l’appariement avec son ancien homologue, et devenant si négligeable - avec son hétérochromatine et ses quelques centaines de gènes - qu’on pouvait même se demander s’il n’allait pas poursuivre son déclin jusqu’à l’extinction finale, phénomène qui a déjà été observé chez un rongeur fouisseur des plaines du Caucase (Ellobius lutescens), désormais dépourvu de chromosome Y [ 1].
Mais grâce à la persévérance de David Page et de son équipe au MIT (Massachusetts Institute of Technology, MA, États-Unis), la génomique est actuellement en voie de réhabiliter le chromosome Y humain. En 2003, ce groupe avait réussi à établir la séquence de la quasi-totalité de l’euchromatine de ce gonosome [ 2, 3]. Dans la région mâle spécifique (MSY), la présence de séquences répétées, amplicons et palindromes (un quart de la région MSY), n’avait pas facilité le séquençage. On savait que ces séquences étaient présentes aussi dans l’Y du chimpanzé, et qu’elles devaient donc préexister à la séparation des deux lignages, estimée à 6 millions d’années environ.
En 2005, une étude comparative des régions dégénérées de l’X dans les chromosomes Y de l’homme et du chimpanzé montrait que de nombreux gènes avaient conservé leur fonctionnalité chez l’homme alors que les gènes orthologues du chimpanzé l’avaient perdue [ 4]. Mais tant que le séquençage de la région MSY du chimpanzé n’avait pas été réalisé, l’étude ne pouvait être qu’approximative. C’est désormais chose faite et ce travail ingrat, toujours effectué par l’équipe de David Page, n’a pas été inutile car l’étude comparative des deux régions MSY a révélé quelques surprises : alors que l’ensemble des autosomes de ces deux proches parents a évolué conjointement (avec 98 % d’homologie), les chromosomes Y ont subi une évolution étonnamment divergente [ 5]. On trouve deux fois plus de palindromes chez le chimpanzé que chez l’homme. De nombreux gènes présents chez l’ancêtre commun ont disparu chez le singe. Par exemple, trois des neuf familles de gènes à expression testiculaire, conservées chez l’homme, ont subi des mutations inactivantes (XKRY ou XK-related protein on Y chromosome, entre autres). Les familles de gènes HSFY (heat-shock transcription factor, Y-linked), PRY (PTPBL-related gene on Y) sont absentes dans la séquence MSY du chimpanzé (et absentes aussi chez cinq autres chimpanzés et deux bonobos où elles ont été recherchées) alors qu’elles sont présentes, non seulement chez l’homme, mais aussi chez le macaque et le chat (entre autres).
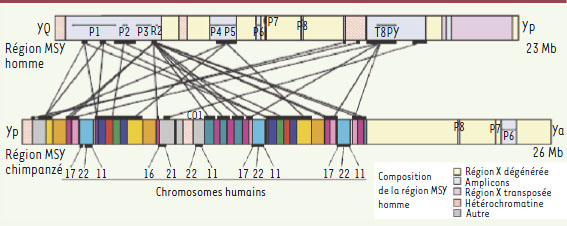
Il en résulte que l’évolution ne s’est pas faite vers une dégénérescence progressive, comme on le supposait, mais qu’il s’est produit un remodelage intense et divergent dans les deux lignages : la recherche des régions d’homologie entre les deux lignages montre qu’elles sont très courtes et dispersées du fait des nombreux remaniements survenus (Figure 1). Ceux-ci n’ont pu se faire que sous l’action de facteurs agissant en synergie, dont trois sont propres à l’Y : (1) la capacité de changement de structure dans les régions ampliconiques ; (2) en l’absence de crossing-over, les effets d’autostop génétique à partir desquels une mutation mâle avantageuse entraînerait la fixation des polymorphismes ou de mutations qui lui sont associés ; (3) la sélection préférentielle pour les gènes impliqués dans la production du sperme.
| Figure 1.
Segments homologues des régions MSY de l’homme et du chimpanzé. (d’après [5] [suppl] © Nature). |
Quant à la divergence des Y homme/singe, elle pourrait s’expliquer par la différence des comportements sexuels : la forte compétition entre les mâles pour la reproduction peut avoir favorisé une pression de sélection vers une amélioration du pouvoir fécondant chez les chimpanzés. Le séquençage de la région MSY d’autres mammifères doit venir compléter cette étude phylogénétique.
Et sans attendre, la grande presse n’a pas manqué de réhabiliter le chromosome de la masculinité : « L’étrange Monsieur Y » 1, « n’est pas pourri » 2,, au contraire, il est « celui qui évolue le plus vite »3. Bravo pour lui !