| |
| Med Sci (Paris). 2010 May; 26(5): 468–470. Published online 2010 May 15. doi: 10.1051/medsci/2010265468.Identification d’une protéine-clé pour le contrôle des sites de recombinaison méiotique Frédéric Baudat, Jérôme Buard, Corinne Grey, and Bernard de Massy* Institut de génétique humaine, UPR1142 CNRS, 141, rue de la Cardonille, 34396 Montpellier Cedex 5, France MeSH keywords: Animaux, Assemblage et désassemblage de la chromatine, Crossing-over, physiologie, Endodeoxyribonucleases, Esterases, métabolisme, Histone-lysine N-methyltransferase, composition chimique, Histone, Humains, Lysine, Méiose, génétique, Souris, Complexes multiprotéiques, Structure tertiaire des protéines, Relation structure-activité, Doigts de zinc |
Lors de la méiose, une seule réplication du génome suivie de deux divisions permet à une cellule diploïde de produire des gamètes haploïdes. La première division se distingue par le fait que les chromosomes homologues d’origines maternelle et paternelle ségrègent. Les échanges réciproques entre chromosomes homologues, ou crossing-over, formés par recombinaison homologue lors de la prophase de cette division, jouent un rôle essentiel pour la formation de gamètes euploïdes fonctionnels. En effet, ils conduisent à la formation d’un lien physique entre les chromosomes homologues, nécessaire à leur ségrégation. La recombinaison méiotique a également pour conséquence de provoquer un réassortiment des allèles dans le génome, ce qui contribue à générer de la diversité génétique. Le mécanisme moléculaire de la recombinaison méiotique, disséqué chez la levure Saccharomyces cerevisiae, est conservé au moins dans ses grandes lignes chez les eucaryotes supérieurs [
1]. La première étape moléculaire est la formation de cassures double-brin de l’ADN catalysées par la protéine Spo11 [
13]. Ces cassures double-brin sont réparées par recombinaison avec le chromosome homologue pour aboutir à la formation des produits de recombinaison, en particulier les crossing-over. |
Concentration des crossing-over dans les points chauds Les crossing-over ne sont pas répartis de manière aléatoire dans le génome [
2]. En particulier, ils sont groupés dans des intervalles d’environ 2 kilobases, appelés points chauds de crossing-over, séparés par des régions longues en moyenne de quelques dizaines de kilobases. Ces points chauds correspondent à des sites d’initiation de la recombinaison méiotique, c’est-à-dire des sites préférentiels de formation de cassures double-brin par SPo11. Chez la souris et l’homme, seul un petit nombre de points chauds de crossing-over ont été identifiés et caractérisés directement. Néanmoins, des méthodes basées sur l’analyse de la diversité génétique dans des populations humaines ont permis d’estimer indirectement le taux de crossing-over à haute résolution dans l’ensemble du génome. Ces méthodes permettent d’identifier les points chauds de crossing-over, et 25 000 à 50 000 points chauds de crossing-over ont ainsi été identifiés chez l’homme [
3]. Une proportion importante de points chauds est associée à un motif partiellement dégénéré de séquence d’ADN (le 13-mère CCNCCNTNNCCNC), sa présence ayant été perçue comme responsable de l’activité de 41 % de ces points chauds [
4]. Néanmoins, l’analyse de la localisation des points chauds n’a pas permis de mettre en évidence de relation forte avec des éléments fonctionnels du génome malgré leur tendance à être situés en dehors des gènes. De ce fait, malgré la description exhaustive des sites de crossing-over dans le génome humain, savoir pourquoi la recombinaison est ainsi ciblée vers des sites précis du génome et comment sont choisis ces sites, sont des questions restées sans réponse jusqu’à la découverte très récente d’un facteur majeur impliqué dans ce processus. |
Un élément génétique contrôle en trans l’activité des points chauds dans l’ensemble du génome Notre équipe a identifié un élément génétique chez la souris, appelé Dsbc1 pour double-strand break control 1, qui contrôle l’activité de points chauds de recombinaison [
5]. La présence d’un allèle spécifique (Dsbc1wm7
), qui provient de la sous-espèce Mus musculus molossinus originaire du Japon, permet l’activation de points chauds spécifiques à des sites où la recombinaison méiotique est indétectable dans d’autres lignées et modifie globalement la distribution des crossing-over le long des chromosomes. Cela a suggéré que Dsbc1 est un déterminant majeur de la distribution de la recombinaison méiotique dans le génome murin. Parmi les gènes présents dans l’intervalle contenant Dsbc1, Prdm9 est un candidat particulièrement intéressant. D’une part Prdm9 est exprimé spécifiquement lors de la prophase de méiose ; d’autre part son produit est une histone méthyltransférase qui catalyse la triméthylation de la lysine 4 de l’histone H3 (H3K4) [
6]. Or, notre équipe a montré récemment que cette modification est associée à l’initiation de la recombinaison à des points chauds chez la souris [
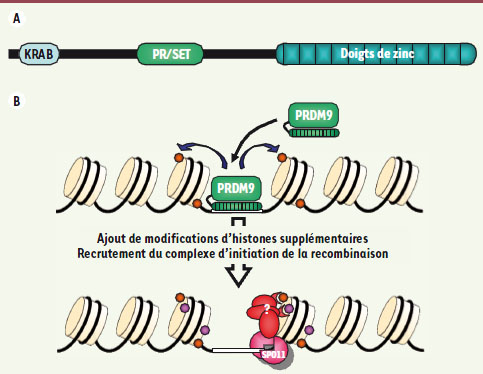
7]. La triméthylation de H3K4, qui est fréquemment associée aux promoteurs de transcription actifs, est associée aux sites de recombinaison de manière indépendante de la transcription de ces régions. En plus du domaine PR/SET responsable de son activité catalytique de méthyltransférase, la protéine PRDM9 contient un domaine carboxy-terminal constitué d’une série de doigts de zinc se succédant en tandem (Figure 1A). Les doigts de zinc sont des éléments capables de lier des séquences spécifiques d’ADN qui peuvent être prédites dans une certaine mesure d’après l’identité des acides aminés présents à trois positions importantes sur chaque doigt de zinc.  | Figure 1.
L’histone méthyltransférase PRDM9 spécifie la position des événements de recombinaison méiotique. A. Domaines fonctionnels de la protéine PRDM9. Le domaine KRAB (Kruppel-associated box) est potentiellement un domaine d’interaction entre protéines. Le domaine PR/SET possède l’activité catalytique d’histone méthyltransférase. Le domaine contenant une série de doigts de zinc permet la reconnaissance de séquences spécifiques d’ADN. B. Modèle de ciblage des événements de recombinaison méiotique par PRDM9. Grâce à ses doigts de zinc, PRDM9 se lie à un motif de séquence d’ADN spécifique (rectangle blanc) et triméthyle la lysine 4 de l’histone H3 des nucléosomes les plus proches (pastilles orange). D’autres modifications post-traductionnelles d’histones sont mises en place (pastilles mauves), peut-être par des protéines recrutées par PRDM9. L’ensemble spécifique des modifications d’histones présentes forme une marque qui est lue par un complexe protéique (en rouge), non identifié à ce jour (?), qui va permettre le recrutement et l’activation du complexe d’initiation de la recombinaison symbolisé par la protéine SPO11 (en rose). |
De façon remarquable, l’allèle wm7 de Prdm9 diffère de l’allèle de la lignée de souris de référence (C57BL/6J) spécifiquement au niveau des séries de doigts de zinc, de telle manière que l’on peut prédire que les deux formes de PRDM9 reconnaissent des séquences d’ADN différentes [
8]. Ces observations nous ont conduits à proposer que la fixation de PRDM9 sur des motifs spécifiques de séquence d’ADN permet la triméthylation de H3K4 sur les nucléosomes les plus proches et favorise localement l’initiation de la recombinaison selon un mécanisme qui n’a pas encore été défini (Figure 1B). |
PRDM9 définit une fraction importante des sites de crossing-over chez l’homme Nous avons ensuite examiné la série de doigts de zinc de la protéine PRDM9 humaine dont la structure est similaire à celle de la souris. De manière frappante, la séquence d’ADN dont on peut anticiper qu’elle sera reconnue par cette série de doigts de zinc contient le 13-mère associé aux points chauds de crossing-over identifiés par l’analyse de la diversité génétique dans des populations humaines. Nous avons confirmé in vitro la fixation spécifique de PRDM9 à un fragment d’ADN double-brin contenant ce motif [8]. Une analyse génétique corrélant la position des sites de crossing-over dans le génome avec l’allèle de PRDM9 présent nous a permis de démontrer l’implication de PRDM9 dans le choix des sites de crossing-over. Dans la population que nous avons étudiée, l’allèle majoritaire de PRDM9 (> 90 %) contient la série de doigts de zinc capable de reconnaître le 13-mère associé aux points chauds. Chez les individus porteurs de cet allèle, environ 60 % des crossing-over ont lieu au niveau de points chauds déjà identifiés par l’analyse de la diversité génétique. Ceci est cohérent avec l’hypothèse selon laquelle une grande partie des points chauds de crossing-over identifiés par l’analyse de la diversité génétique dans des populations humaines sont des points chauds déterminés par l’allèle majoritaire de PRDM9. Quelques individus de la population étudiée portent également un allèle minoritaire (allèle I) contenant une série de doigts de zinc différente et qui reconnaît une séquence d’ADN différente. Chez les individus portant l’allèle I de PRDM9, seuls 20 % des crossing-over ont lieu dans les points chauds identifiés par l’analyse de la diversité génétique. Cette différence spectaculaire dans le choix des sites de crossing-over est très significative (p < 10-11) et permet de conclure que les doigts de zinc de PRDM9 jouent un rôle majeur pour déterminer la position des crossing-over chez l’homme [8]. |
Dynamique évolutive de PRDM9 Les travaux présentés ci-dessus montrent que la position d’une fraction importante des crossing-over est déterminée par la fixation de la protéine PRDM9 sur les séquences définies par les motifs reconnus par les doigts de zinc de cette protéine. La fixation de PRDM9 sur ses cibles permet, selon un mécanisme qui reste à découvrir, de spécifier des sites préférentiels d’initiation de la recombinaison à l’origine des points chauds. Des approches différentes ont permis à deux autres équipes d’identifier indépendamment le rôle de PRDM9 dans la spécification des points chauds de recombinaison chez la souris et chez l’homme [
9,
10]. Une particularité remarquable de ce mécanisme est que des changements dans les doigts de zinc de PRDM9, s’ils modifient la séquence d’ADN reconnue, suffisent à changer les cibles génomiques de PRDM9 et par conséquent à renouveler les points chauds de recombinaison dans l’ensemble du génome. Par exemple, les protéines PRDM9 de l’homme et du chimpanzé diffèrent fortement au niveau de leurs doigts de zinc, ce qui permet d’expliquer que, malgré des génomes aux séquences très similaires (moins de 1,2 % de divergence), les points chauds de crossing-over ne soient pas conservés entre ces deux espèces [9]. L’étude de PRDM9 a permis de mettre en évidence une évolution extrêmement rapide de ses doigts de zinc qui concerne essentiellement les acides aminés impliqués dans la spécificité des séquences d’ADN reconnues [
11]. Cette évolution rapide se traduit par une grande diversité intra-spécifique et une forte divergence entre espèces de PRDM9. L’évolution rapide des doigts de zinc de PRDM9 semble être contrainte par sélection positive, c’est-à-dire une sélection favorisant la fixation de nouveaux allèles dont on ne connaît pas l’origine [11]. Il est intéressant de noter que Prdm9 a été identifié comme un gène responsable de stérilité hybride chez la souris, par un mécanisme qui n’est pas connu à ce jour [
12]. PRDM9 pourrait donc avoir une ou plusieurs fonctions s’ajoutant à la spécification des sites de recombinaison. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1. Handel MA, Schimenti JC. Genetics of mammalian meiosis: regulation, dynamics and impact on fertility. Nat Rev Genet 2010; 11 : 124–36. 2. Buard J, de Massy B. Playing hide and seek with mammalian meiotic crossover hotspots. Trends Genet 2007; 23 : 301–9. 3. Myers S, Bottolo L, Freeman C, et al. A fine-scale map of recombination rates and hotspots across the human genome. Science 2005; 310 : 321–4. 4. Myers S, Freeman C, Auton A, et al. A common sequence motif associated with recombination hot spots and genome instability in humans. Nat Genet 2008; 40 : 1124–9. 5. Grey C, Baudat F, de Massy B. Genome-wide control of the distribution of meiotic recombination. PLoS Biol 2009; 7 : e35. 6. Hayashi K, Yoshida K, Matsui Y. A histone H3 methyltransferase controls epigenetic events required for meiotic prophase. Nature 2005; 438 : 374–8. 7. Buard J, Barthes P, Grey C, et al. Distinct histone modifications define initiation and repair of meiotic recombination in the mouse. EMBO J 2009; 28 : 2616–24. 8. Baudat F, Buard J, Grey C, et al. PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice. Science 2010; 327 : 836–40. 9. Myers S, Bowden R, Tumian A, et al. Drive against hotspot motifs in primates implicates the PRDM9 gene in meiotic recombination. Science 2010; 327 : 876–9. 10. Parvanov ED, Petkov PM, Paigen K. Prdm9 controls activation of mammalian recombination hotspots. Science 2010; 327 : 835. 11. Oliver PL, Goodstadt L, Bayes JJ, et al. Accelerated evolution of the Prdm9 speciation gene across diverse metazoan taxa. PLoS Genet 2009; 5 : e1000753. 12. Mihola O, Trachtulec Z, Vlcek C, et al. A mouse speciation gene encodes a meiotic histone H3 methyltransferase. Science 2009; 323 : 373–5. 13. Baudat F, de Massy B. SPO11 : une activité de coupure de l’ADN indispensable à la méiose. Med Sci (Paris) 2004; 20 : 213–8. |